─ 265 ─
ⓒ The Korean Society of Limnology. All rights reserved.
This is an open-access article distributed under the terms of the Creative Commons Attribution Non-Commercial License(http://creativecommons.org/licenses/by-nc/3.0/), which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provide the original work is properly cited.
서 론
보구치(Pennahia argentata)는 농어목 민어과에 속하는 난류성 어종으로 민어과 물고기 중 개체수가 많은 종이 다(Koh et al., 2014). 암수에 따른 보구치의 비율은 53:47 로 암컷 비율이 수컷보다 높게 나타났으며 크기는 평균 전장(TL)이 암컷은 24.2cm, 수컷은 23.7cm로 암컷의 크 기가 수컷보다 다소 크게 나타났다(Jeon et al., 2020). 보 구치의 주 먹이원은 새우류로 먹이원 분석 연구에서 153 개체 보구치 위(gut)에서 발견된 217개체의 먹이생물 중 가장 주요한 먹이 생물이 새우류(Macrura)로 출현 빈 도는 68.6%를 차지하였고, 개체수 비 66.4%, 습중량 비미토콘드리아 DNA의 cytochrome c oxidase subunit I을 이용한
보구치
(Pennahia argentata) 계통 분석
박재원1·박기연2·곽인실1,2,*
1전남대학교 해양융합과학과, 2전남대학교 수산과학연구소
Phylogenetic Analysis Using Cytochrome c Oxidase Subunit I of Silver Croaker(Pennahia argentata) Mito chondria DNA. Jae-Won Park1 (0000-0002-4067-7089), Kiyun Park2 (0000-0003-2965-6970) and Ihn-Sil Kwak1,2,*
(0000-0002-1010-3965)(1Department of Ocean Integrated Science, Chonnam National University, Yeosu 59626, Republic of
Korea; 2Fisheries Science Institute, Chonnam National University, Yeosu 59626, Republic of Korea)
Abstract Silver croaker(Pennahia argentata) is a turbulent species that is widely distributed worldwide and is mainly found in the bottom of the ocean. In the study, we characterized the cytochrome c oxidase subunit I
(COI) gene of the mitochondrial DNA(mtDNA) on P. argentata inhabiting Gwangyang Bay and analyzed the
phylogenetic location of marine fish species. As a result of multiple arrangement of 605bp COI sequences,
high homology of mtDNA nucleotide sequences was confirmed in the silver croakers from Gwangyang Bay (98~100%). However, the nucleotide variation was different according to the catching points of the inland and the open seas of Gwangyang Bay. The nucleotide sequence variation in COI was high in P. argentata from
the open seas of Gwangyang Bay(43.2~70.3%). Furthermore, the phylogenetic analysis of 13 fish showed
that P. argentata from Gwangyang Bay were grouped into one clade with P. argentata reported in Taiwan, and the evolutionary distance was 0.036. In addition, it was identified that the evolutionary distance was close
to that of fish belonging to the Mi-iuy croaker(Miichthys miiuy) and the Big-head pennah croaker(Pennahia
Macrocephalus)(0.041~0.048). The result of these studies will be used as the key genetic information for fisheries resources monitoring and species diversity management according to the coastal environment.
Key words: Pennahia argentata, cytochrome c oxidase subunit I(COI), phylogenetic analysis, Gwangyang Bay, coastal environment
Manuscript received 11 June 2020, revised 16 August 2020, revision accepted 7 September 2020
* Corresponding author: Tel: +82-61-659-7148, Fax: +82-61-659-7149, E-mail: inkwak@hotmail.com /iskwak@chonnam.ac.kr
ISSN: 2288-1115(Print), 2288-1123(Online)
는 43.3%, 상대중요도 지수비는 78.2%로 보고되었다 (Koh et al., 2014; Huh et al., 2018). 보구치는 주로 수심
20~140m에 달하는 바닥의 모래나 개펄인 저층에 주로 서식한다(Huh et al., 2018). 보구치의 분포는 우리나라의 남해, 서해, 동해, 일본 해역, 중국 해역, 서태평양 및 인도 양 등 전 세계적으로 광범위하게 보고되고 있다(Koh et al., 2014). 또한 계절에 따른 보구치 분포는 여름 금강 하 구역에서 26.9% 우점율이 나타났고(Lee et al., 2014), 광 양만 대도주변에서는 조사된 74종 8,133개체 중 7.2% 우 점종으로 보고되었다(Kwak et al., 2012). 보구치는 상업성 이 높은 어종으로 주로 쌍끌이 또는 외끌이 대형 기선 저 인망, 연안 복합, 안강망, 근해 자망 어업 등에 의해 어획 되며, 1984년까지 10,655톤의 어획량을 보이다가 1990년
대 이후 감소와 증가를 반복하다(Lee and Zhang, 2001)
최근에는 2,700톤의 어획량으로 과거보다 크게 감소하 여 관리 방안이 필요한 어종으로 보고되었다(Huh et al., 2018; Jeon et al., 2020). 보구치에 관한 연구는 주로 연 안이나 근해에 서식하고 있는 보구치에 대한 생식과 성 장(Kwon et al., 1999), 해당 지역의 지리적 특성, 변이 에 따른 성장, 생식 주기 및 성숙에 관한 연구(Higuchi et
al., 2003; Yamaguchi et al., 2004, 2006), 보구치 후기자
어(post-larva)의 먹이 선택(Cha and Park, 2001) 등에 대
해 연구가 진행되었다. 이러한 연구들은 보구치에 대한 자 원생태학적 연구와 분포 특성 등에 대해 국한되어 이루어 졌다. 해양생태계는 어업, 양식업, 해양 운송 및 여가 활동 등 에 인위적인 활동에 영향을 받는다. 또한 연안생태계 환 경의 복잡성으로 지역별, 개체 간 변이로 서식하는 어류 의 생태학적 형태분류가 어려운 종도 있다. 계통유전학적 (Phylogenetic) 연구를 통한 어류의 유전적 기원(origin)과 생물 다양성 정보는 해양자원관리 및 환경관리를 위한 주 요한 정보를 제공한다(Iyiola et al., 2018). 예로 담수어류 인 누치의 미토콘드리아 DNA를 이용한 분자 계통학적 연
구(Park et al., 2019)와 민어(Miichthys miiuy)에서 열충격
단백질의 계통진화적 유전적 특성(Wei et al., 2012)에 관 한 일부 연구가 보고되었다. 수환경 변화는 어류의 유전적 B5 2018-09-09 B site(127.47, 34.49) 115 92 15.92 B7 2018-09-09 B site(127.47, 34.49) 110 85 12.61 C1 2018-09-09 C site(127.49, 34.45) 203 170 96.62 C2 2018-09-09 C site(127.49, 34.45) 196 165 87.48 C3 2018-09-09 C site(127.49, 34.45) 186 158 73.24 C4 2018-09-09 C site(127.49, 34.45) 130 105 22.31 C5 2018-09-09 C site(127.49, 34.45) 114 91 15.31 C6 2018-09-09 C site(127.49, 34.45) 110 88 13.74 C7 2018-09-09 C site(127.49, 34.45) 105 84 12.11 D1 2018-09-09 D site(127.49, 34.42) 201 170 98.07 D2 2018-09-09 D site(127.49, 34.42) 193 163 84.16 D3 2018-09-09 D site(127.49, 34.42) 184 155 72.63 D6 2018-09-09 D site(127.49, 34.42) 170 142 58.35 D8 2018-09-09 D site(127.49, 34.42) 188 158 79.31
다양성을 감소시키고 유전적 변이 정도가 낮아지면 환경 변화에 민감한 영향을 받아 생존에 영향을 미칠 수 있다. 생물 다양성의 변화는 계통유전학적 정보 분석을 통해 수 산자원을 관리할 수 있다. 이에 본 연구에서는 한국 광양 만 연안 일대에 서식하는 국내산 보구치를 대상으로 미토
콘드리아 DNA인 cytochrome c oxidase subunit I(COI) 유
전체를 분석하여 계통유전학적 위치와 어류 간 진화적 근 연관계를 확인하고, 이를 근거로 보구치 분자 계통유전학 적 기준을 제공하고자 한다. 이러한 결과는 연안환경 변화 에 따른 어류의 유전적 모니터링과 수산자원의 다양성 분 석 및 자원보존-관리를 위한 기초자료로 활용될 수 있을 것이다.
재료 및 방법
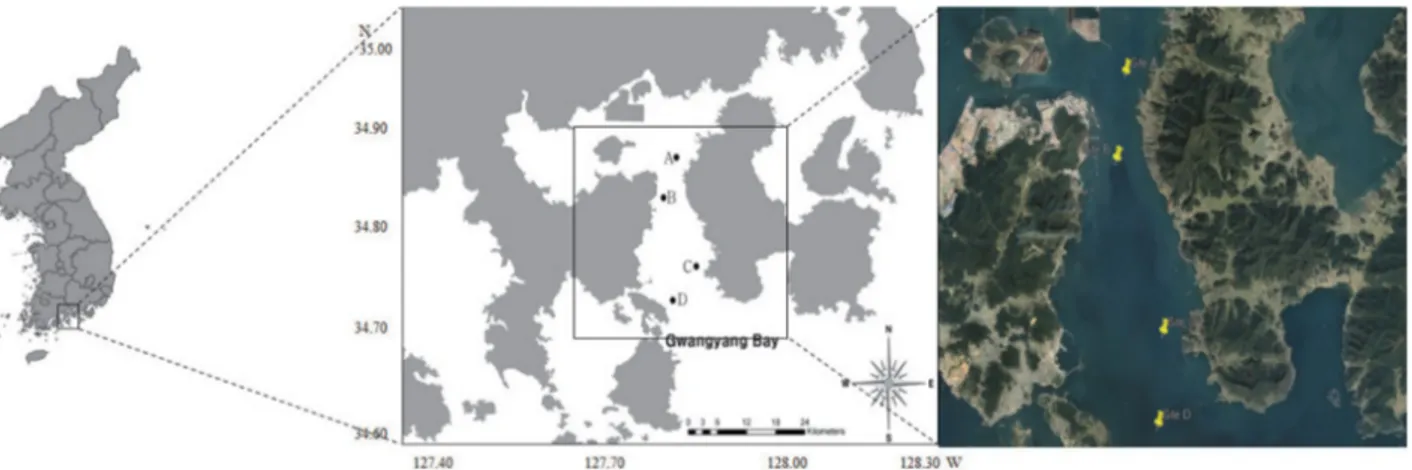
1. 보구치 어획 지점 및 샘플보관 본 연구에서 사용된 보구치(Table 1)는 2018년 9월 광 양만 일대에서 새우조망어구를 이용하여 어획하였다. 광 양만 어획 지점은 A(127°48′W, 34°52′N), B(127°47′W, 34°49′N), C(127°49′W, 34°45′N), D(127°49′W, 34°42′N) (Fig. 1)로 총 4 지점에서 실시하였다. 채집한 보구치는 총 23마리로 실험실로 옮기는 즉시 생체량(Body Weight,BW), 전장(Total Length, TL), 체장(Standard Length, SL)
을 측정한 뒤 근육 조직을 해부하여 샘플을 - 80°C Deep
freezer(Thermo Fisher Scientific)에 보관하였다.
2. Genomic DNA 추출
보구치 total genomic DNA 추출은 Bioneer의 AccuPrep
genomic DNA extraction kit를 사용하여 제공되는 Protocol 에 따라 수행하였다. 실험 과정은 EP tube에 TL buffer 200 μL를 넣고 근육조직을 균질하게 homogenize하고
Protei-nase K 20μL, RNase A 10μL를 넣어 단백질과 RNA를 제
거하였다. 60°C에서 1~2시간 Lysis 과정을 거치고 GB
buffer와 Ethanol을 넣고 원심분리 후 상등액을 취하였다. WA1과 WA2 buffer를 통해 washing 과정을 거친 후 EA
buffer로 Genomic DNA를 elution하여 사용시까지 - 80°C
Deep freezer(Thermo Fisher Scientific)에 보관하였으며 각
지점 위치, 종명, 추출한 날짜 등의 정보를 기록해 두었다.
추출된 DNA의 정량은 1.0% Agarose gel(w/v)로 나노드럽
(NanoDrop ND-2000 spectrophotometer)(Implen, Munich,
Germany)으로 측정 확인하였다.
3. 보구치 COI 유전자의 PCR 증폭 및 염기서열 분석 보구치의 미토콘드리아 DNA에서 COI 유전자 증폭을 위해 Bioneer의 Taq DNA polymerase를 사용하여 PCR (Polymerase chain reaction)을 수행하였다. 증폭에 이용 된 Primer는 Forward: 5′-TCAACCAACCACAAAGAC-ATTGGCAC-3′와 Reverse:
5′-ACTTCAGGGTGACCGA-AGAATCAGAA-3′이다(Ward et al., 2005). 30μL PCR
mixture에는 20μL 멸균된 ultrapure water, 3.0μL 10×PCR
buffer, 1μL of 각각의 primer(20mmol L-1), 1.5 μL dNTPs
(2.5mmol L-1 each), 0.5μL Taq DNA polymerase(2.5
U μL-1, Bioneer, Korea), 그리고 3.0μL genomic DNA
template(25ng μL-1) 등이 포함된다. PCR cycle 조건은
95°C에서 7분간 초기 변성 후 95°C에서 40초 변성, 55°C
에서 40초 primer 결합, 72°C에서 1분간 합성 과정을 40회
반복하여 증폭하였으며, 반복 완료 후 최종 합성은 72°C에
Fig. 2. Multiple alignment of COI sequences in P . ar gentata of Gw angyang B ay of K orea. The red dotted line indicated multiple sequences
line indicated multiple sequences of
P
. ar
gentata
from B site, the purple dotted line indicated multiple sequences of
P
. ar
gentata
from C site, and the green dotted lines indicated multiple
sequences of
P
. ar
gentata
서 10분간 이루어졌다. PCR 결과는 1.5% Agarose gel을 이 용한 전기 영동을 통해 시각적으로 확인하였다. 또한 PCR
증폭된 결과물들(PCR amplification products)은 DNA Gel
Extraction Kit(Solgent, Daejon, Korea)를 이용해 젤 조각
(gel slices)으로부터 정제하고(purified), ABI3730xl DNA
Analyzer 플랫폼(Macrogen, Korea)으로 Sequencing 하여
염기서열을 분석하였다. 4. 데이터 분석
광양만에서 어획된 보구치의 다중배열 분석은 Gene Doc
Program(version 2.6.001)을 사용하여 나타내었다. COI 유
전자 시커스를 통한 단백질 데이타베이스 분석은 NCBI Gen Bank Database에 있는 보구치 또는 민어과 등 서로
다른 13종의 어류들의 상동 서열(homologous sequences)
을 Blastx에 배열하여 단백질 데이타베이스를 비교하였
다. 계통유전학적 분석(phylogenetic analysis)은 Mega X
software(version 10.04)을 이용하여 1000 bootstrap으로
neighbor-joining trees로 분석하였고, 쌍의 진화적 거리
(pairwise genetic distances)도 Mega X software(version
10.04)에 있는 Maximum Composite Likelihood method를 통해 계통진화적분석을 수행하였다.
결과 및 고찰
본 연구에서 보구치 또는 민어과를 포함하는 다양한 어 종들 간의 단백질 데이타베이스 기반 계통유전학적 분석 으로 진화적 근연관계를 비교하였다. 광양만 일대에서 어 획된 보구치의 평균 전장(TL), 체장(SL), 생체량(BW)은 162.97mm(TL), 134.44mm(SL), 56.40g(BW)(최소: 98 mm(TL), 79mm(SL), 10g(BW), 최대: 210mm(TL), 180 mm(SL), 111.53g(BW))로 관찰되었다(Table 1). 최근 보 고된 조사결과에서 2018년 1월에서 12월까지 남해안에서 어획된 보구치 2,939 개체의 전장 범위를 측정한 결과 암 컷 13.5~35.7cm, 수컷 12.8~34.3cm이며, 평균 전장(TL) 의 길이는 암컷 24.2cm, 수컷 23.7cm로 보고되었다(Jeon et al., 2020). 연구에서 조사된 광양만 보구치는 같은 연도 에 어획된 남해안 보구치의 평균 전장(TL) 보다 작은 개체 로 확인되었다.Fig. 3. Phylogenetic tree of the COI sequences in P. argentata constructed by neighbor-joining analysis(bootstrap value 1000). The
num-bers at the nodes are the percentage bootstrap values. The color indicated individuals each sampling site(Red: A site, Blue: B site, Purple: C site, Green: D site).
광양만 보구치에서의 미토콘드리아 DNA내 605bp COI 유전자를 발굴하여 염기서열을 다중 배열한 결과가 Fig. 2 이다. 광양만 각 지역에서 어획된 보구치 COI의 염기서열 은 전체 개체들에 대해 98~100% 염기서열 상동성을 나타 내었지만, 어획지역별로 상동성이 다르게 나타났다. COI 염기서열 내 뉴클레오타이드 변이는 A지점 보구치에서 는 16.2%, B지점 보구치에서는 24.3%, C지점 보구치에 서는 70.3%, D지점 보구치에서는 43.2%로 나타났다. 이 동성이 용이한 어류지만 광양만 내해와 외해에서 어획된 보구치의 미토콘드리아 내 염기서열 변이는 내해보다 외
해쪽 채집지역 보구치에서 높게 나타났다(Fig. 1 and Fig.
2). 광양만의 내해와 외해는 다양한 환경요인(온도, 염분,
TOC, TC, TN, TP, Chl-a, EC, TIC)들의 차이와 환경 DNA
(eDNA) 분석을 통해 종분포의 다양함이 보고되었다(Jo et
al., 2019). 광양만 외해는 내해에 비해 높은 염분, EC, TIC
와 낮은 Chl-a와 TN이 나타났다. 또한 광양만 내, 외해에 서 eDNA를 이용한 OTU 분석 결과 우점하는 OTU는 산호
속(Acropora sp.)이였고 아우점은 요각류인 Acartia sp.으 로 나타났지만 OTU의 구성요소의 분포는 외해와 내해가 다르게 나타났다(Jo et al., 2019). 본 연구에서 광양만 내, 외해에 따른 보구치 미토콘드리아 DNA의 염기서열 변이 는 내, 외해의 다른 환경요인들과 종분포에 따른 환경변화 적응의 유전적 반응으로 연관될 수 있지만 더 많은 보구치 개체에서의 변이 분석을 통한 연구가 필요할 것이다. 이러 한 결과는 광양만에 서식하는 보구치들의 진화적 근연관
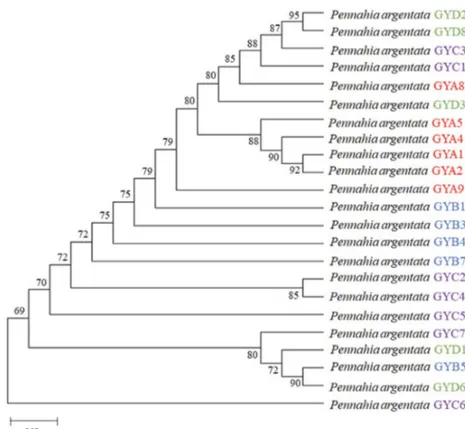
계에서도 나타났다(Fig. 3). 보구치 COI DNA 시컨스로 계
통유전학적 분석결과 A와 B지점의 보구치들에 비해 C와 D지점 보구치들의 유전적 진화관계가 더 다양하게 나타남
을 확인하였다(Fig. 3).
나아가 본 연구에서는 Genbank에 등록된 보구치와
민어를 포함한 13종의 어류(P. argentata, Pennahia
Mac-rocephalus, M. miiuy, Argryosomus japonicus, Otoli thes
ruber, Stellifer lanceolatus, Stellifer microps, Stellifer
rast-rifer, Umbrina bussingi, Umbrina robinsoni, Plagioscion
Fig. 4. Phylogenetic tree of the COI sequences in 13 species of fish constructed by neighbor-joining analysis(bootstrap value 1000). The
numbers at the nodes are the percentage bootstrap values. The red doted rectangle indicated the clade with closely related P. argentata and P. argentata individuals in the study.
squamosissimus, Roncador stearnsii, Nerbris microps) COI 단백질 아미노산 시컨스를 바탕으로 계통유전학적 관계를
분석하였다(Fig. 4 and Table 2). 광양만 보구치들은 계통
유전학적으로 중국과 타이완에서 보고된 보구치와 진화적 근연관계가 있음이 확인되었다. 특히 중국의 Rongcheng Bay 보구치(AFB82994)와 한국 광양만 보구치들이 하나 의 계통군(clade)을 이루었다. 이러한 결과는 보구치 종내 에서 시컨스 상동성이 타이완, 중국에서 보고된 보구치와 유사함을 나타낸다. 본 연구는 각 지역 보구치 미토콘드리 아의 유전적 기원이 근연관계를 파악할 수 있음을 보여주 었다. 하지만 보구치 종내에서도 광양만 보구치 시컨스와
유연관계가 높은 보구치(sequence identity(SI)=99.83%
in Table 2)는 한 계통군을 이루지만 유연관계가 다소 낮은
보구치들은(SI=88.34~88.76) 같은 종임에도 불구하고 다
른 계통군을 이루는 것으로 쏘가리와 같은 담수 어류에 비 해 연안 보구치의 유전적 다형성이 높고 분화시간이 상대
적으로 짧지 않은 것으로 분석된다(Kim and Song, 2011).
그 외 대두이석태(P. Macrocephalus)와 민어(M. miiuy)와
진화적 근연관계를 나타냈다. 다른 어류에서는 N. microps,
P. squamosissimus, O. ruber, M. miiuy, A. japonicus들이
각 어종끼리 가까운 계통군을 형성하였고, S. lanceolatus 와 S. microps는 같은 계통군을 형성하는 것을 확인하였다. 또한 Maximum Composite Likelihood method 방법을 이용 하여 어류 종간의 근연관계 분석을 위해 DNA 염기서열 염 기 치환 정도를 수치화 한 결과, 광양만에서 어획된 보구치 들은 진화적 거리가 0.005 이하로 나타났다(Table 3). 중국 과 타이완에서 보고된 보구치와의 진화적 거리는 0.036으 로 나타나고 다른 어종들과의 진화적 거리는 0.036~0.058 사이로 민어(M. miiuy)와 대두이석태(P. Macrocephalus)에 속한 어종과 진화적 거리가 가장 가까운 것으로 나타났 다. 어류에서 미토콘드리아 DNA 발굴 및 계통진화적 분 석을 통한 신종 분석은 최근에 보고되고 있다. 27종의 어 류에서 계통진화적 분석결과 Johnius taiwanensis가
Joh-nius trewavasae(MG 917694)와 같은 계통군을 이루며 다
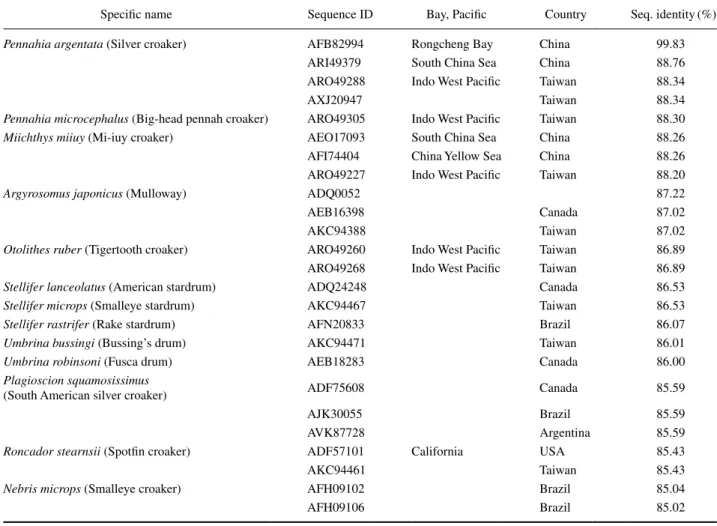
Table 2. List of 13 fish species for phylogenetic analysis.
Specific name Sequence ID Bay, Pacific Country Seq. identity(%) Pennahia argentata (Silver croaker) AFB82994 Rongcheng Bay China 99.83
ARI49379 South China Sea China 88.76 ARO49288 Indo West Pacific Taiwan 88.34
AXJ20947 Taiwan 88.34
Pennahia microcephalus (Big-head pennah croaker) ARO49305 Indo West Pacific Taiwan 88.30
Miichthys miiuy (Mi-iuy croaker) AEO17093 South China Sea China 88.26
AFI74404 China Yellow Sea China 88.26 ARO49227 Indo West Pacific Taiwan 88.20
Argyrosomus japonicus (Mulloway) ADQ0052 87.22
AEB16398 Canada 87.02
AKC94388 Taiwan 87.02
Otolithes ruber (Tigertooth croaker) ARO49260 Indo West Pacific Taiwan 86.89 ARO49268 Indo West Pacific Taiwan 86.89
Stellifer lanceolatus (American stardrum) ADQ24248 Canada 86.53
Stellifer microps (Smalleye stardrum) AKC94467 Taiwan 86.53
Stellifer rastrifer (Rake stardrum) AFN20833 Brazil 86.07
Umbrina bussingi (Bussing’s drum) AKC94471 Taiwan 86.01
Umbrina robinsoni (Fusca drum) AEB18283 Canada 86.00
Plagioscion squamosissimus
(South American silver croaker) ADF75608 Canada 85.59
AJK30055 Brazil 85.59
AVK87728 Argentina 85.59
Roncador stearnsii (Spotfin croaker) ADF57101 California USA 85.43
AKC94461 Taiwan 85.43
Nebris microps (Smalleye croaker) AFH09102 Brazil 85.04
Table 3.
P
airwise distance matrix inferred from the nucleotide substitution of fish CO1 sequences.
Total 29 indi
viduals consist of 4 indi
viduals in this study (A1, B1, C1, D1) and 25 indi
viduals of 13 fish species in the
Table 2. 1 2 3 4 5 6 7 8 9 10 11 12 13 14 15 16 17 18 19 20 21 22 1 -2 0.000 -3 0.005 0.005 -4 0.000 0.000 0.005 -5 0.036 0.036 0.041 0.036 -6 0.036 0.036 0.041 0.036 0.000 -7 0.036 0.036 0.041 0.036 0.000 0.000 -8 0.036 0.036 0.041 0.036 0.000 0.000 0.000 -9 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 -10 0.053 0.053 0.058 0.053 0.016 0.016 0.016 0.016 0.010 -11 0.048 0.048 0.053 0.048 0.016 0.016 0.016 0.016 0.010 0.010 -12 0.048 0.048 0.053 0.048 0.010 0.010 0.010 0.010 0.005 0.005 0.005 -13 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 -14 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 0.000 -15 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 0.000 0.000 -16 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 -17 0.036 0.036 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 0.000 -18 0.036 0.036 0.041 0.036 0.000 0.000 0.000 0.000 0.005 0.016 0.016 0.010 0.005 0.005 0.005 0.005 0.005 -19 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 0.010 0.010 0.010 0.010 0.010 0.005 -20 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 0.000 0.000 0.005 0.010 -21 0.036 0.036 0.041 0.036 0.000 0.000 0.000 0.000 0.005 0.016 0.016 0.010 0.005 0.005 0.005 0.005 0.005 0.000 0.005 0.005 -22 0.047 0.047 0.052 0.047 0.010 0.010 0.010 0.010 0.015 0.016 0.016 0.010 0.005 0.005 0.005 0.005 0.005 0.010 0.015 0.005 0.010 -23 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 0.000 0.000 0.005 0.010 0.000 0.005 0.005 24 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 0.000 0.000 0.005 0.010 0.000 0.005 0.005 25 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.010 0.010 0.005 0.010 0.010 0.010 0.000 0.000 0.005 0.010 0.000 0.005 0.005 26 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 0.010 0.010 0.010 0.010 0.010 0.005 0.010 0.010 0.005 0.015 27 0.041 0.041 0.047 0.041 0.005 0.005 0.005 0.005 0.010 0.021 0.021 0.016 0.010 0.010 0.010 0.010 0.010 0.005 0.010 0.010 0.005 0.015 28 0.047 0.047 0.052 0.047 0.010 0.010 0.010 0.010 0.015 0.016 0.016 0.010 0.015 0.015 0.015 0.005 0.005 0.010 0.015 0.005 0.010 0.010 29 0.047 0.047 0.052 0.047 0.010 0.010 0.010 0.010 0.015 0.016 0.016 0.010 0.015 0.015 0.015 0.005 0.005 0.010 0.015 0.005 0.010 0.010 1. Pennahia ar gentata
(A1 in the study), 2.
Pennahia ar gentata (B1 in the study), 3. Pennahia ar gentata (C1 in the study), 4. Pennahia ar gentata (D1 in the study), 5. Pennahia ar Pennahia ar gentata (AR O49288), 8. Pennahia ar gentata (AXJ20947), 9. Pennahia micr ocephalus (AR O49305), 10. Miic hthys miiuy (AEO17093), 11. Miic hthys miiuy (AFI74404), 12. (ADQ0052), 14. Ar gyr osomus japonicus (AEB16398), 15. Ar gyr os omus japonicus (AKC94388), 16. Otolithes ruber (AR O49260), 17. Otolithes ruber (AR O49268), 18. Stellifer lanceolatus Stellifer r astrifer (AFN20833), 21. Umbrina b ussingi (AKC94471), 22. Umbrina r obinsoni (AEB18283), 23. Pla gioscion squamosissimus (ADF75608), 24. Pla gioscion squamosissimus 26. Roncador stearnsii (ADF57101), 27. Roncador stearnsii (AKC94461), 28. Nebris micr ops (AFH09102), 29. Nebris micr ops (AFH09106)
른 Johinus 어종들과 진화적 근연관계를 나타내는 신종
으로 확인되었다(Chao et al., 2019; Lin et al., 2020). 또한
20종의 어류에서 계통진화적 분석결과 Nibea diacanthus
(Blackspotted Croaker)가 Pennahia argentata(KC545800)
와 같은 계통군을 이루며 Nibea에 속하는 어종과 진화적
근연관계를 나타냈다(Cheng and Zhang, 1987; Hu et al.,
2016; Hu et al., 2020). 해양어류에서 유전적 정보을 기반
으로 한 종 다양성연구는 제한적으로 연구되고 있다(Wei
et al., 2012; Song et al., 2015; Hu et al., 2020). 이에 본 연 구와 같은 어류의 유전적 변이 및 진화적 근연관계 분석
정보는 기존의 식성 및 분포에 대한 자료와 더불어(Kwon
et al., 1999; Koh et al., 2014; Huh et al., 2018) 해양어류 자 원의 관리와 보존을 위해 주요하게 활용될 수 있을 것이 다. 어류의 미토콘드리아 DNA 계통진화적 분석은 지리적
변이나 분포에 따른 형태적(체색, 크기 등) 분류를 위한 어
류자원의 유전적 정보를 제공하고 미기록종의 발굴이나 외래종 유입에 따른 국내종 선별을 위한 기준을 제공한다 (Kim and Song, 2011; Song et al., 2015).
적 요
보구치는 난류성으로 전 세계적으로 널리 분포하며 해 양 저층에 주로 서식하는 어종이다. 광양만에 서식하는 보 구치의 미토콘드리아 DNA에서 cytochrome c oxidase
sub-unit I(COI) 유전자를 발굴하고 해양 어류종에서의 계통유 전학적인 위치를 분석하였다. 발굴된 미토콘드리아 DNA 내 605bp COI 시컨스의 다중배열 결과 광양만 보구치들 에서는 높은 염기서열 상동성을 확인하였다(98~100%). 하지만 광양만 내해와 외해의 어획지점에 따라 염기서 열 변이가 다르게 나타나는 것을 확인하였다. 외해지점 의 보구치들에서 COI 내 염기서열 변이가 높게 나타났다 (43.2~70.3%). 나아가 13종 어류의 COI 계통유전학적 분 석결과 광양만 보구치는 타이완에서 보고된 보구치와 하 나의 계통군(clade)으로 묶이고 진화적 거리는 0.036으 로 나타났다. 또한 민어(M. miiuy)와 대두이석태(Pennahia Macrocephalus)에 속한 어종과 진화적 거리가 가까운 것 으로 나타났다(0.041~0.048). 본 연구의 결과는 국내산 보 구치의 분자 계통유전학적 정보를 제공함으로 연안환경에 따른 어류자원 모니터링 및 종다양성 관리에 주요한 유전 적 자료로 활용될 것이다. 저자정보 박재원(전남대학교 학생), 박기연(전남대학교 연 구교수), 곽인실(전남대학교 교수) 저자기여도 개념설정: 곽인실, 방법론: 곽인실 & 박기연 & 박재원, 분석: 박재원 & 박기연, 자료제공: 곽인실 & 박재 원 & 박기연, 자료관리: 곽인실 & 박기연, 원고 초안작성: 박재원 & 박기연, 원고 교정: 곽인실, 박기연, 박재원, 원고 편집 및 검토: 곽인실, 박기연, 박재원, 과제관리: 곽인실, 연 구비 수주: 곽인실. 모든 저자는 논문의 결과에 동의하였 고, 출판될 최종본을 검토하고 동의하였습니다. 이해관계 이 논문에는 이해관계 충돌의 여지가 없음. 연구비 이 논문은 한국연구재단 중점연구사업의 지원을 받아 수행된 연구임[NRF-2018-R1A6A1A-03024314].
REFERENCES
Cha, S.S. and K.J. Park. 2001. Feeding selective of postlarvae of white croaker Argyrosomus argentatus in Kwangyang Bay, Korea. Journal of Korean Fish Society 34: 27-31. Chao, N.L., C.W. Chang, M.S. Chen, C.C. Guo, B.A. Lin, Y.Y.
Liou, K.N. Shen and M. Liu. 2019. Johnius taiwanensis, a new species of Sciaenidae from the Taiwan Strait, with a key to Johnius species from Chinese waters. Zootaxa 4651: 259-270.
Cheng, Q.T. and B.S. Zhang. 1987. Sciaenidae, index of classi-fication of China. Beijing: Science Press 1: 317-324. Higuhi, T., A. Yamaguchi and T. Takita. 2003. Age and growth
of white croaker, Pennahia argentata, in ariake sound, Japan. Bulletin of the Faculty of Fisheries, Nagasaki
Uni-versity 84: 47-51.
Hu, Z., X. Chai, Y. Wang, Y. Zhu and D. Zhu. 2016. Complete mitochondrial genome of the giant croaker Nibea
japoni-ca(Perciformes, Sciaenidae) and phylogenetic analysis of the Sciaenidae. Mitochondrial DNA Part A 27: 1-3. Hu, Z., X. Chai and Y. Wang. 2020. Characterization of the
complete mitochondrial genome sequence of Nibea
dia-canthus and its phylogenetic implication. Mitochondrial
DNA Part B 5: 525-527.
Huh, S.H., H.C. Choi and J.M. Park. 2018. Feeding Relationship between Co-occurring Silver Croaker(Pennahia argentata) and Japanese Sillago(Sillago japonica) in the Nakdong River Estuary, Korea. Korean Journal of Ichthyology 30: 224-231.
Iyiola, O.A., L.M. Nneji, M.K. Mustapha, C.G. Nzeh, S.O. Oladipo, I.C. Nneji, A.O. Okeyoyin, C.D. Nwani, O.A. Ugwumba, A.A.A. Ugwumba, E.O. Faturoti, Y.Y. Wang, J. Chen, W.Z. Wang and A.C. Adeola. 2018. DNA barcoding of economically important freshwater fish species from north-central Nigeria uncovers cryptic diversity. Ecology
habits of white croaker, Pennahia argentata in the coastal waters off Sejon island, Korea. Journal of the Korean
So-ciety of Fisheries and Ocean Technology 50: 139-146. Kwak, S.N., S.H. Huh and H.W. Kim. 2012. Change in fish
assemblage inhabiting around Dae Island in Gwangyang bay, Korea. Journal of the Korean Society of Marine
En-vironment & Safety 18: 175-184.
Kwon, D.H., Y.J. Kang and D.W. Lee. 1999. A population eco-logical study of White croaker, Argyrosomus argentatus Houttuyn in Korean waters. II. Age and growth. Journal
of the Korean Society of Fisheries Resource 2: 54-60. Lee, S.I. and C.I. Zhang. 2001. Prediction of the Variation in
Annual Biomass of White Croaker Argyosomus argenta-tus in Korean Waters using Leslie Matrix. Journal of the
Korean Fisheries Society 34: 423-429.
Lee, S.K., M.S. Choi, Y.I. Seo and J.B. Lee. 2014. Seasonal species composition and cluster analysis of catches by shrimp beam trawl in the Geum river estuary. Journal of
the Korean Society of Fisheries and Ocean Technology
182.
Ward, R.D., T.S. Zemlak, B.H. Innes, P.R. Last and P.D. Hebert. 2005. DNA barcoding Australia’s fish species.
Philo-sophical Transactions of the Royal Society B: Biological Sciences 360: 1847-1857.
Wei, T., Y. Sun, G. Shi, R. Wang and T. Xu. 2012. Characteri-zation and SNP variation analysis of a Hsp70 gene from
Miiuy croaker and its expression as related to bacterial challenge and heat shock. Fish & Shellfish Immunology 33: 632-640.
Yamaguchi, A., G. Kume and T. Takita. 2004. Geographic vari-ation in the growth of white croaker, Pennahia argentata, off the coast of northwest Kyushu, Japan. Environmental
Biology of Fishes 71: 179-188.
Yamaguchi, A., T. Todoroki and G. Kume. 2006. Reproductive cycle, sexual maturity and diel-reproductive periodicity of white croaker, Pennahia argentata(Sciaenidae), in Ariake Sound, Japan. Fisheries Research 82: 95-100.