대한임상병리사회지 : 제 26 권 제 1 호
1994.
B형 간염바이러스 표면항원 양성 혈청에서 B형간염 바이러스의 e항원 및 DNA 측정에 관한 분석
서울의과학 연구소 • 서울임상병리검사센터 • 고려병원 임상병리과*
최철석·김은아·홍창식·이경옥·박창봉*
Kèy words : HBsAg, HBeAg, Hybridization with DNA probe, Polymerase chain reaction.
1 •
서 폰 counter로 측정한다. 이 방법에 의한 측정 감
도는HBV
DNA 의 약3
X 104에 서 3
X10
5copy 수에 해당하는 1 -- 10-
1pg 정도까지 측정할 수 있으나, 그 특이성이 다소 떨어지고 있다6) 그 러나 최근 유전자 1 개 copy 에 해당하는 량 (1 fg) 까지도 측정할 수 있는 고도의 감도와 특 이성을 지닌, 일명 중합효소 연쇄반웅 (PCR;
polymerase chain reaction) 법 이 개 발 되 어 HBV를 확진하고, 또 치료 경과를 관찰하는데 보다 유용하게 되었다.
본 저자들은 HBV 표면항원 (HBsAg) 의 혈청 학적 양성을 나타낸 혈청 검체를 대상으로 하 여 혈청학적 방법의 HBeAg 측정과, DNA probe 법 에 의 한 DNA 측 정 및 DNA poyme-
rase 측정을 하고 이에 대한 결과를 확인하기
위한 수단으로 PCR을 이용한 DNA 증폭을 시 도하여 그 결과를 분석하였다.
B형 간염 바이 러스 (hepatits B virus;HBV) 는 아직도 전세계적으로 중요한 보건학적 문제 의 하나로 남아있다. 특히 남부 아프리카나 동 남아시아 지역에서는 주민의 50 % 정도가 혈 청학적 항체 양성을 나타내고 있다 1) 우리 나 라도 다른 선진국의 감염 양성률 1--2 %에 비 하면 약 6% 로서 높은 감염률을 나타내고 있 는 실정이다1). B 형 간염을 진단하기 위하여 대 체로 혈청학적 측정법을 이용하여 질병의 진행 과정을 예측하고 있는데, 이 과정에서 확진을 하기에는 다소 문제점이 있었던 것도 사실이 다. 비록 B형 간염 바이러스를 측정하는 데 있 어 혈청학적 marker가 예민하고 편리한 방법 이라 할지라도 이것이 항상 바이러스의 활동성 을 의미하는 훌륭한 지표가 되지는 못한다2) 이런 관점에서 볼 때 바이러스의 DNA 라든가
DNA를 중합하는 DNA 중합효소 (DNA poly- II . 재료 및 방법 merase) 를 측정하는 것이 혈중의 바이러스 존
재를 나타내는 데 보다 정확한 신뢰성과 증거
1.재료
를 줄 수 있다2-6)
DNA를 측정하기 위하여 현재 이용되고 있
1)대상 검체
는 방법으로 혈청 중에 존재하는 HBV DNA를 의뢰된 임상혈청 검체 중 면역 혈청학적 방 추출하여 filter paper나 nylon membrane 에 옮 법 (radioimmunoassay; RIA) 에 서 HBsAg 양성 겨 고정한 후 특이한
:J:-JAprobe 로 hybridi- 으로 판명된 17 예를 선별하여 시험 대상으로 zation해 서 autora diography 혹은 scintilla tion 하였으며
,이 과정 에 서 단지 HBsAb, HBcAb만
m ω
혈청학적 양성을 나타낸 검체는 대상에서 제외 하였다.
2) DNA probe hybridization법
DNA probe hybridization 법 에 의 한 HBV DNA 측정은 시판 kit(Abbott Laboratories
,USA) 를 이용하였으며 1251 표지 probe를 DNA hy bridiza tion 에 이 용하였다.
3) DN A polymerase측정
DNA 합성 을 위 한 이igomer deoxynucleotides
는
poly(dA). poly(dT) 12-18(Pharmacia ,
USA) 를사용하였으며 양성대조에 사용한 DNA 중합효
소는T
4DNA polymerase(Pharmacia ,
USA) 를사용하였다.
본 시험에 대상으로한 검체 17 예 중 9 예만
DNA probe법에 의한 HBV DNA 측정올 하였 으며 측정 방법은 kit 내에 동봉된 안내서의 지시에 따라 수행하였다. 즉 간단하게 서술하 면 다음과 같다. 우선 주 실험에 들어가기 전 에 sepharose column을 수직 rack에 걸고 24
시간 정도 방치하여 column내에 충전되어 있 는 sepharose gel 이 완전히 충전되도록 하였다.
이어, 음성, 양성 및 검체 시험올 위해 멸균 된
1.5 ml microfuge tube를 준비하고 각 용기 에 NaCl 완충액 100μl 넣은 후 음성, 양성대 조액 및 검체 혈청을 각각 100μl씩 넣었다.
희석된 각 반웅 용기에 바이러스 단백올 분 해 하기 위 하여 10 μl 단백 분해 효소( proteinase
K) 를 첨가하여 혼합하고 간단하게 micro-fuge
한 후 실온에 1 시간 동안 방치하였다. 방치후
4) 중합효소 연쇄반응의 oligonucleotides 20μ1 NaOH 완충액으로 실온에서 30분간 처리
primer 하여 바이러스 외부 단백질올 완전히 소화시켰
사용된 primer는 HBV genome 의 core 부분을 다. 용기 내의 용액중에 방출된 HBV DNA 에 암호화한 유전자8) 로 각각 1763 번과 2032 번 위 이 바이러스 DNA 에 특이한 l젠 표지 probe
치에서 시작되는 유전자, 5' - GCTTTGGGGC 70μl를 각 반웅 용기에 첨가한 후 65
0C 에서
ATGGACATTGACCCGACTATAA-3 과 5 - 약 18 시 간 동안 방치 하여 hybri-dization 시 켰
CTGACTACTAATTCCCTGGATGCTGGGTCT 다. 이 어 hybridization 된 반웅물을 sepharose -3 의 한쌍을 외측 primer(primer 1) 로 사용 column으로 분리 용출하여 용출액을 감마 하고 270 base pair를 증폭하도록 합성하여 사 counter로 10분동안 측정하였다.
용하였다. 또, 이에 대한 내측
primer (primer
2) 로서 1778 번 위치에서 시작되는 유전자, 5
- GACGAATTCCATTGACCCGTATAAAGA
ATT-3 ’와 2017 번 위치에서 시작되는 유천자,
5
’ -A TGGG A TCCCTGG A TGCTGGGTCTTC CAAA-3 의 한쌍을 선 정 하고 258 base pair
증폭을 목표로 하였다.
2.
방법1) HBeAg
측정시험 대상으로 한 검체 17 예 중 9 예만 HBeAg을 통위 원소 면 역 분석 법 (radioimmun- oassay ;
RIA) 으로 측정하고 그 측정값에 따라음성, weakly 양성, 양성으로 표시하였다.
2) DNA
probe 법3)
HBV의DN A polymerse
측정시험 검체 100μl를 동량의 세포 용해 완충
액(25 mM Tris-HCl, pH '7.5, 5 mM dithiothreitol , 50 mM KCI 50 % glycerol , 2.5
% Triton X -100) 으로 처리하여 세포 단백올 파괴 하였다. 0.5 ml micro tube 에 양성, 음성 대조 및 검체를 표시한 후 각 tube 에 template -pnmer 용액 [0.5% Triton X-100 , 50mM KC 1 , 0.5 % BSA , 1
μ1poly(dA) -oligo(dT)
12-18J 를 50μl 씩 넣었다. 양성 대조에 2μl
T4 DNA polymerase(New England Biolab ,
England) 를 음성대조에는 증류수, 그리고 검체
용기에는 혈청 검체를 각각 5μl 씩 넣고 잘 혼
합한 후 얼음에 15 분간 방치하였다. 다음에 반
응 혼합액 [1M Tris- HCl
,pH 7.5
,10 mM
DTT
, 1.0M MgCb
,50 mM dNTP(ATP
,CTP
,GTP)
,5 μ1 3H-TTP
(1mci/m
l),D.W. 58.5 μIJ 를 85μe 씩 넣고 혼합한 후 37
0C 에서 1 시간 동안 반웅 시킨 다음 얼음에 10분간 정치하였 다.
반웅 검체 100 띠를 glass fiber filter(GFF) 에 spot하고 200 띠의 cold 10 % trichloroacetic acid/
O
.lM sod. pyrophosphate(TCA
-5PP) 를 가하여 polynucleotide를 침 전 시 켰 다. 이 어 cold 5
% TCA-5PP 용액으로 세척하고 다시 냉 알
콜로 세척하였다. GFF를 건조한 다음 liquid scintillation vial에 넣고 cocktail (PPO 5g , bis - M5P 0.5g
,toluene 11 ml)을 넣고 scintilla- tion counter로 방사능을 측 정 하 였다.
4) 중합효소연쇄반응 (PCR) 에 의한 HBV DNA 측정
HBV DNA 추출은 일반적으로 실시되고 있
는 단백 분해효소( proteinase K) 처리 및 유기 용매에 의한 방법으로 추출하였다9)
(1)
1 차 PCR
사전에 제조하여 분주된 PCR 반웅 완충액
(10 mM Tris-HCI pH 8.8 ,
1.5 mM MgCb , O.
1 % Triton X -100 , 10 pmole primer 1 , O.
2 mM dNTP
,50 mM KCI) 에 음성대조에 증류
수, 양성대조에HBV DNA
, 검체 용기에는 각시험검체 DNA를 2μl 씩, 그리고 2.5 unit 의
Taq DNA polymerase를 각 반응 용기에 가하 고 최종 반웅액의 량은 20μl가 되도록 하였다.
반웅 용기를 thermal cycler로 옮기고 94
0C 1
분간 denaturation
,55 Oc 광1. 5 분간 annealing
,72
0C 3 분간 extension을 한 주기로 하여 전체 40 회 실시하였다.
(2) 2 차
PCR 반응 종료된 1 차 PCR 산물 중 음성을 나타낸 검체는 반응 산물 2μl를 1 차 PCR 에
사용 된 pnmer에 대 한 내 측primer(primer 2)
를 사용하여 2 차 PCR을 하였다. 이 때 pnmer
를 제외한 모든 반응액 성분과 증폭 조건은
1차 증폭과 같게 하였고 전체 증폭 회수는 30
회 실시하였다.
(3)
PCR산물 확인최종 반웅이 종료된 반응액의 일부를 gel loading 완충액에 혼합하고 l μg/ml ethidium
bromide가 함유된 2 % agarose gel 상에서 전기 영동을 하였다. 전기 영동이 완료되면 자외선 투사기 (ultra violet transilluminator) 상에 서 원 하는 크기의 band 를 확인하였다.
III.
결 과
이번 조사에서는 측정된 HBeAg 의 결과는 9
예 중 3 예 (52, 53
,55) 가 양성을, HBV DNA
probe는 51
,52
,54
,59 의 4 예가 양성을 보였 다. 반면 이 들 9 예 중에 DNA polymerase는 1
예 (52) , PCR은 3 예 (51 , 52
,55) 가 양성을 나 타냈다(표 1). PCR 측정은 양성 대조는 원하 는 크기의 DNA 단편이 증폭되었고 음성 대조
Table 1. Detection of HBeAg , HBV DNA probe , DNA polymerase and PCR in 17 HBsAg pos-
ltI
ve sera.
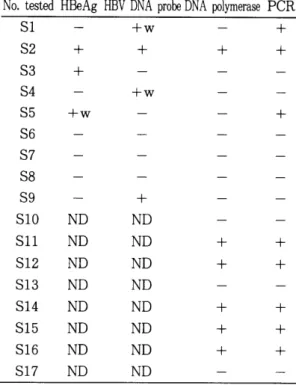
No. tested HBeAg HBV DNA probe DNA polymerase PCR
nu
1L oι q니 A낸 다니
CU
?l
-2
3 4 5 6 7 8 9
U
tμ나 μμ μL
--- S S S S S S S
s s s
ε
S S
E E
S S+ +
+w
+
+ + +
+w
+w +
+
ND ND
ND ND + +
ND ND + +
ND ND
ND ND + +
ND ND + +
ND ND + +
ND ND
+w : weakly positive.
ND : assay not done.
-72 -
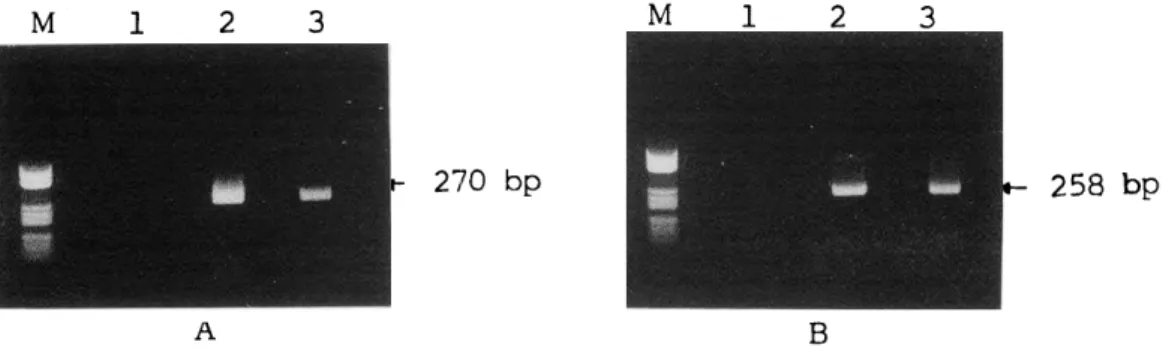
에는 DNA 증폭은 발견되지 않아 본 시험에서 는 외부 DNA 혼입은 인정되지 않았다(그림
1.2). 또, 17 예의 PCR 대상 검체 중 1 차 PCR
에서 5 예가, 2 차 PCR 에서 3 예가 각각 증폭되 어 전체적으로 8 예 (47.9 %)가 양성을 보였다.
HBeAg를 사용하는데 이는 체내 감염된 바이 러스 활성 기전을 이해함에서 비롯된 것으로 생각된다2). HBeAg 이 과거에는 바이러스의 증 식 및 감염력과 밀접한 관계가 있는 것으로 생 각되어 왔었다. 그러나 일률적으로 생각하여 S3는 HBeAg
,S4와 S9 는 HBV DNA probe 임상적 소견을 판단하는 것은 곤란하다 12 , 13) 에서만 각각 양성을 나타내 서로 상반된 결과 HBeAg는 바이러스의 증식이 없어도 간세포 를 보였다. 반면, S5 는 PCR 에서도 양성을 나 유전자에 삽입된 HBV DNA 에 의해서 생성이 타내어 혈청내 DNA 가 존재함이 증병되었다. 가능할 수 있으므로 체내 바이러스 증식을 정
한편, 모든 방법에서 양성을 보인 겸 제는S2
확하게 의미 한다고는 할 수 없으며 14 , 15) 실제뿐이였다. DNA polymerase와 PCR만 측정한 HBeAg 양성인 검체에서 HBV DNA가 검출되 나머지 S10--S17 까지의 8 검체는 두 방볍의 지 않았던 연구 보고도 있다 16) 이는 측정 방 결과가 100 % 일치하였다. 이 검체는 간 기능 법의 감도상에서 비롯된 것이 아니라 HBeAg
효소 활성치가 급격히 상승(자료 별도로 제시 에서 anti-HBeAb 까지의 혈청 전환기 동안은 되지 않음)된 것으로서 이는 체내 바이러스의 바이러스 간기 (intermitant) 로서 17) 이 기간 동 증식이 활발하게 이루어 지고 있음을 입증하여 안에 혈청내에 HBV DNA는 측정되지 않지만 여전히 HBeAg는 양성을 타나내는 것으로 알 려졌다. 따라서 HBeAg 의 역가나 임상 경과와 는 밀접한 상관 관계가 없는 것으로 보고되었 다. 한편, HBV 의 활성 지표로서 DNA poly-
간염의 진행 과정을 관찰하기 위한 수단으로 merase를 측정하여 판단하려는 시도도 15) 있어
HBsAg
,anti - HBc 를 측정하여 사용하여 왔는 왔지만 방법상의 예민도가 문제가 제기되었다.
데 이는 실제 임상과 상당한 차이가 있는 것으 본 연구에서 대상으로한 17 예의 HBsAg 양 로 알려졌다. 이런 이유에서 또 다른 지표로 성 검체에 대해서 HBeAg 양성인 겸체는 3 예
주었다.
N. 고 찰
M l 2 3
270 bp
A
M 1 2 3
• 258 bp
B
Fig. 1 PCR amplification of hepatitis B virus DNA. PCR product was electrophoresed through 2 %
agarose gel and detedted by UV -transilluminator after staining with ethidium bromide.
A is result of first PCR products , 270 bp DNA fragment was amplified for positive of HBV DNA.
B is 2nd PCR products which shown 258 bp for positive of HBV DNA.
M; molecular marker
,Lane 1 through 3; negative control
,positive control
,sample
,respectively.
~ ~ r11 o] ~ 1 ojl(S2) ~ SJ.. ~ 1:3J- t{] oJl Ai
0J:;.d
% 1..-t E} 4l ol ~ i!} ~
2_£ ~ rrJ1. ~ ~~ ~]4l oJJ Ai t1} o 1 c~ ~ ~ * ~l ~ ti~ ~ ~% ~ o] ~ q
.Jlw
-T- ~139.
q. .=rc~ 4 S3 ~ q~ £
:§-1:3J- t{] oJl Ai
g ).d
g_4 E} 'tR ~ .g.
.=I~ ~% ~ ~ <5} ~l ~-f ~
-T-~ ~7-]'i}- ~Ai£<:8-B-
cs-}~7-]'i}- t~}o]c~~~~
;.d cycle o l serovonversion Tt! 7-ll oJl
~7-] ?l-
g_77} "'] 4.!fl
q. S5~ HBeAg7} oJ:;.d oJ1 °] ol
PCR OJ:).d .2..£ DNA e~~ ~ ~~ cs-tetl ~ ~
li! oJ1, probe t{]
~% ;.d g_ 4 E} 4l ol probe t{] o l {J-£7} ~qJL ~ -T- ~q . .=rc~4 S9~ Jtl-7-1 DNA probe~ oJl 'i! ~ :Aj ~<Xi q~ ~ ~ o] ~ 7}
1}
~7-] ?l- 7-] 'i} o l.C probe 1:3J-
~ ~:; ;.d g_
.Jl~ ~ ~ rrJ1 tlj:; oj ~ ~ ~%oJ1 9-j ~ Ai ~ OJ:J.d
o] o} \:! 7} "]
z.}~q.
DNA probet{] o]4 PCRoJl ~ ~ HBV DNA
~ 78 ~ ~ ~oJl DNAe~~ etllf- 'i! ~ 78 <5}~ ~ o]
E-£~oJl Ai £ ~ ift>}~ ~ o] ~ ~ ~ AJ~ ~
¥ftt!t>}~ ~ ~ q~ .!f-~7} ~
g_~ ~q. o]
Jtl ~ -T-oJl Ai ~ 78 ¥1 87R (S10----S17) ~ ~ ~1 ~
ff% {}~
g_Oll?:!~ ~ ~~ ~~ Z!-71 'o .:s:.~
7.]7}
~o] AJ1).!fl ~~l£Ai ~lt.H tl}o]c-1~~
tt;.d
g_~ 78 <5} 7] ~ ~ -&~ ~ t>-}4 £ DNA polymerase9l- PCRoJl ~~DNA ~~'i}g_ A]
.£ ~ ~ o] q. o] 7d ~oJl .:g-
JiL.§-~l £ f- 1:3J-t{j ~
~i!}7} ~7-]~g_ .!ii_~q.
o] :Jd~ DNA polymerase e~~ ~ DNA~
~~1 t>}etl Pi .g. t~}o] e-1.~ DNA~ 'i} ~ Jl ~ 7]
uJl ~oJ1 o1 ~ ~ -2- DNA~ e~~ ~ 4E}4l7l £
~q. rr}c.}Ai PCRoJl ~~ DNA~ ~~ t>] ~ 78~ ~o]q.
~ ~~
_Q_£oj Jtl ~ -T-oJJ Ai 3:A}.!f]_ ~i!}, ~ HBeAg ~
0J:;.d o]4 ~ 78 DNA ~:i!l- ~ g J.d, :E.C HBeAg.C g ;.d
g_.!ii_o]:A]~ HBV DNA~
~ ~!@ oJ1 ~ ~ rrJ1 Bi8 {}~ g_ ~ Jtl-t>}JL Oll +
~ :t!-~~71 ~ t>~ Ai ~ HBeAg~ 78 + ~ ti~ <5}
etl ~ol %1:~ DNA£ ~ 78 7}?;~ PCRoJl ~ ~ DNA ~~o] .!ii_q %%~ l:iJ"t{]Blo]c.} A}li~
q.
Detection of Hepatitis B e Antigen and Hepatitis B Viral DNA in Sera of Hepatitis Surface Antigen Positive
Choi, C. S., Kim, U. A;, Hong, C. S., Lee, K. 0., Park, C. P.*
Seoul Medical Science Institute, Seoul Clinical Laboratory Dept. of Clinical Pathology, Korea Hospital, Seoul Korea.*