PUBLIC HEALTH WEEKLY REPORT, PHWR
Vol.11 No.3
2018
www.cdc.go.kr
pISSN 2005-811X
eISSN 2586-0860
CONTENTS
CONTENTS
0054
국내 분리 참진드기 및 사람 유래 중증열성혈소판 감소증후군 바이러스의 분자유전학적 특성0063
네트워크 생물학과 질병 연구0074
주요 감염병 통계 환자감시 : 전수감시, 표본감시 병원체감시 : 인플루엔자 및 호흡기바이러스 급성설사질환, 엔테로바이러스국내 분리 참진드기 및 사람 유래
중증열성혈소판감소증후군 바이러스의
분자유전학적 특성
Molecular genomic characterization of tick- and human-derived severe fever with
thrombocytopenia syndrome viruses isolated in South Korea
Yun Seok-Min, Park Sun-Whan, Lee Won-Ja, Lee Joo-Yeon
Division of Emerging Infectious Disease and Vector Research, Center for Infectious Diseases Research, KNIH, KCDC Park Su-Jin, Choi Young-Ki
College of Medicine and Medical Research Institute, Chungbuk National University
Background: Severe fever with thrombocytopenia syndrome (SFTS) is an emerging tick-borne viral disease caused by the
SFTS virus (SFTSV). During the entomological surveillance of SFTSV infections among Korean ticks collected in SFTS outbreak areas, we isolated one SFTS V, which was designated KAGWT. In addition, we isolated three SFTSVs from patients with typical SFTS symptoms.
Methods/Results: We aimed to analyse the complete genomic sequences of these tick- and human-derived Korean SFTSV
isolates and compare their genetic and evolutionary characteristics. Genetic and phylogenetic analyses of these sequences revealed that the tick-derived Korean SFTSV strain clustered into the genotype B, the most prevalent genotype in South Korea, and was closely related to other SFTSVs in the same group. Furthermore, our results demonstrate that there are at least four different genotypes of SFTSV strains co-circulating in South Korea.
Conclusion: These findings provide information regarding the molecular epidemiology, genetic diversity, and evolution of
SFTSV in East Asia.
Keywords: Severe fever with thrombocytopenia syndrome virus, Tick, Human, South Korea
질병관리본부 국립보건연구원 감염병연구센터 신종감염병·매개체연구과 윤석민, 박선환, 이원자, 이주연* 충북대학교 의과대학 미생물학교실 박수진, 최영기 *교신저자 : ljyljy@nih.go.kr/ 043-719-8490
몸 말
본 연구에서는 사람과 진드기에서 유래된 국내 분리주 4주에 대한 유전자 정보를 분석하였다. 이중 인체 유래주 3주는 충북지역 SFTS 환자 혈청으로부터 분리되었으며(CB1, CB2, CB3), 진드기 유래 1주는 2013년 SFTS 환자발생지역(강원 삼척)에서 채집한 작은소피참진드기로부터 분리되었다(KAGWT) (Table 1)[6]. 이 바이러스에 대한 전체 유전자 정보 분석은 Sanger 방법을 통해 확인하였다. 이후 기존에 중국, 일본 및 국내에서 확인된 SFTS 바이러스와의 염기서열 및 계통 유연관계 분석, 분절 간 재조합 (reassortment) 여부를 분석하였다. 참진드기 및 사람 유래 국내 분리주의 전체 염기서열 확인 결과, 기존 phlebovirus 속에 속하는 바이러스들의 유전자 구성과 동일한 3개 유전자로 분절화 되어있음을 확인하였다. 즉, 6,368 nucleotides (nt) 크기의 L 분절, 3,378 nt 크기의 M 분절 및 1,746 nt 크기의 S 분절로 구성됨을 확인하였다. 또한 기존 bunyaviruses에 대한 연구 결과, 각 분절의 상보적인 시퀀스에 의해 형성되는 panhandle-like 구조는 transcription initiation, viral RNA replication/ encapsidation 및 viral genome packaging에 있어서 중요한 역할을 한다고 알려졌는데[8], 이 연구를 통해 확인된 참진드기, 사람 유래 국내 분리주 및 기존 분리주에서도 이 부분의 염기서열은 비교적 잘 보존된 것을 확인할 수 있었다(Figure 1).들어가는 말
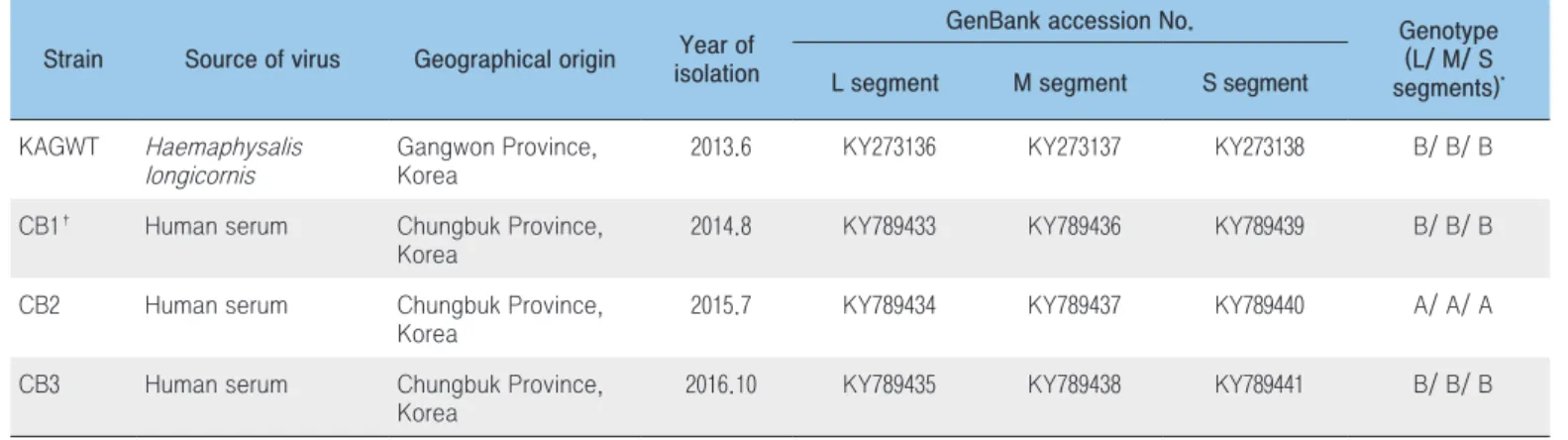
중 증 열 성 혈 소 판 감 소 증 후 군 ( S e v e r e f e v e r w i t h thrombocytopenia syndrome, SFTS)은 고열, 소화기 증상(구토, 설사, 구역질 등), 혈소판 감소, 백혈구 감소 등을 특징으로 하는 중증열성 바이러스성 질환으로, 2011년 중국에서 처음으로 보고된 이후 2013년에 일본 및 국내에서도 환자발생이 확인되었다[1-3]. 원인 병원체는 Bunyaviridae과 Phlebovirus 속에 속하는 SFTS 바이러스로서, 음성가닥의 RNA를 유전자로 갖는데 이는 총 3개의 분절(L, M, S)로 구성되어있다[1]. 전파 경로는 SFTS 바이러스에 감염된 참진드기 교상에 의한 감염이 일반적이지만, 혈액이나 체액을 통한 사람 간 전파도 보고되어있다[4]. 국내의 경우, 2013년 강원지역에서 SFTS에 의한 첫 사망환자가 보고된 후로, 2013년 36건, 2014년 55건, 2015년 79건, 2016년 165건이 발생하여 매년 환자가 증가하였으며, 치명률도 평균 22%로 확인되었다[5]. 이 글에서는 국내 서식 작은소피참진드기(Haemaphysalis longicornis) 및 SFTS 환자 혈청에서 분리된 SFTS 바이러스의 전체 유전자 염기서열 비교 분석을 통해 참진드기 및 사람 유래 SFTS 국내 분리주의 분자유전학적 특성에 대해 알아보고자 하였다.Table 1. Sequence information for SFTSV strains identified in this study
(Ref: PLoS Neglected Tropical Diseases 2017:11(9);e0005893) *The SFTSV genotypes were classified according to the phylogenetic results of previous study[7].
†This strain was isolated from the serum of deceased human patient.
Strain Source of virus Geographical origin Year of isolation
GenBank accession No. Genotype (L/ M/ S segments)*
L segment M segment S segment
KAGWT Haemaphysalis longicornis
Gangwon Province, Korea
2013.6 KY273136 KY273137 KY273138 B/ B/ B
CB1† Human serum Chungbuk Province,
Korea
2014.8 KY789433 KY789436 KY789439 B/ B/ B
CB2 Human serum Chungbuk Province, Korea
2015.7 KY789434 KY789437 KY789440 A/ A/ A
CB3 Human serum Chungbuk Province, Korea
KAGWT (Korea) CB1 (Korea) CB2 (Korea) CB3 (Korea) Gangwon/Korea/2012 KASJH (Korea) KAGWH3 (Korea) KAGBH5 (Korea) KAGBH6 (Korea) KACNH3 (Korea) KADGH (Korea) KAGNH (Korea) KAGNH4 (Korea) KAJNH2 (Korea) YG1 (Japan) SPL003A (Japan) HB29 (China) SDLZtick12-2010 (China)
virus / segment
L 5 ’ end 3 ’ end M 5 ’ end 3 ’ end S 3 ’ end 5 ’ endACACAAAGAACCCCC
GGGGGTTCTTTGTGT
C
1. Nucleotide sequence alignment of complementary sequences within the 5’ and 3’-noncoding regions (NCRs) of L (blue boxes), M (yellow boxes), and S boxes) segments of 18 completely sequenced SFTSV strains, including tick- and human-derived Korean strains (KAGWT, CB1, CB2, and CB3). Compared the sequence of the KAGWT strain, the different, and deleted sequences are indicated by open boxes, and hyphens, respectively. The closed boxes representthe three segments. (Ref: PLoS Neglected Tropical Diseases 2017
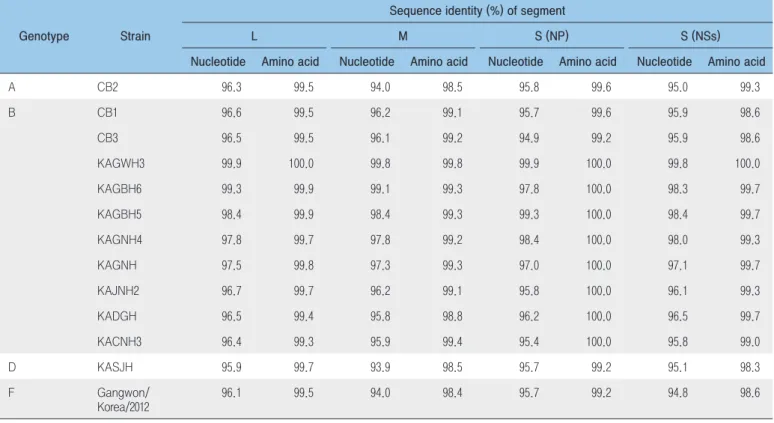
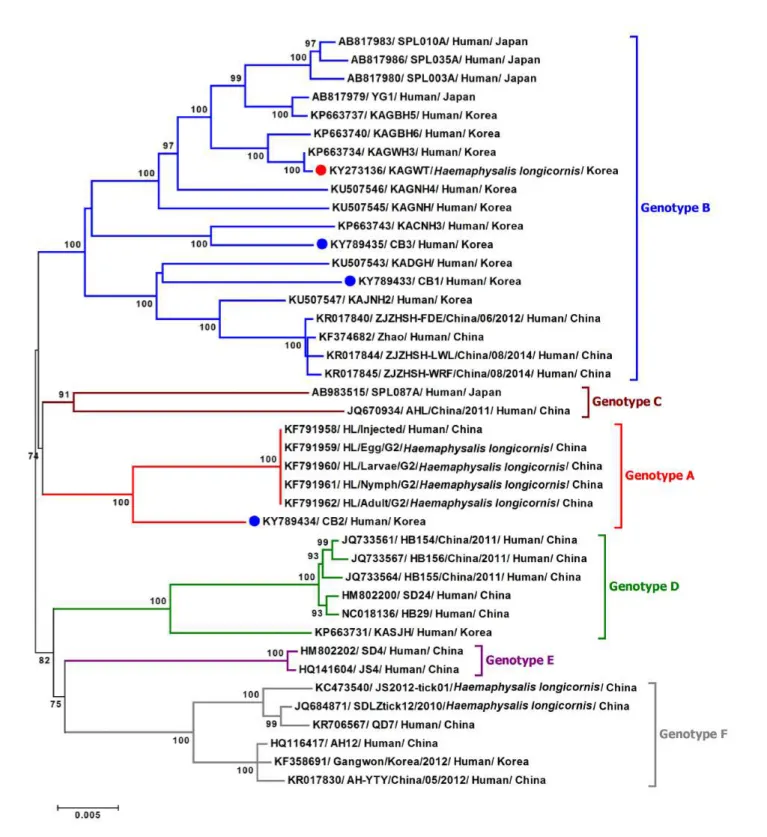
참진드기 유래 국내 분리주인 KAGWT주의 경우, 사람 유래 국내 분리주 염기서열(아미노산 서열)과 비교한 결과, L 분절은 95.9(99.3)~99.9(100)%, M 분절은 94.0(98.4)~99.8(99.8)%, S 분절은 94.9(98.3)~99.9(100)%의 염기서열(연역된 아미노산 서열) 상동성을 보였다. 특히, 국내 분리주 중 같은 지역(강원 삼척) SFTS 환자로부터 분리된 KAGWH3주와 가장 높은 상동성을 나타냈다(Table 2). 또한, 기존 SFTS 바이러스주의 L, Gn, Gc, NSs, NP 단백질을 이루는 아미노산 서열과 비교한 결과, 참진드기 유래 국내 분리주에서만 나타나는 특정 아미노산 치환은 M 분절 내 Gn 단백질의 66번 위치에서 확인되었다(Alanine에서 Serine으로 치환, A66S). 아직까지 이 부위의 아미노산 치환과 SFTSV virulence, pathogenesis 등의 연관성에 관해서는 확인된 바는 없지만, M 분절이 관여하는 중화항체 유도, 바이러스 세포 침입(entry)에 필요한 수용체 결합 등에 영향을 미칠 수 있기 때문에 이와 관련해서는 추가적인 연구가 필요하다. 기존 분리주와의 계통유연관계를 각 유전자 분절별로 분석한 결과, KAGWT주는 총 6가지 유전형 (A~F) 중 유전형 B에 속했고, 상동성 결과와 동일하게 같은 지역에서 분리된 사람 유래 분리주인 KAGWH3주와 높은 유연관계를 보였다. 그러나 국내 분리주에서의 분절 간 재조합(reassortment)은 확인되지 않았다(Figure 2~4). 또한, 참진드기 유래 국내 분리주를 포함한 총 196주의 중국, 일본, 국내 분리주에 대한 유전형 분포를 분석한 결과, 중국에서는 6가지 유전형이 모두 분포하였으며, 그 중 유전형 F가 가장 많이 분포하는 것으로 나타났다. 반면, 일본에서는 기존 확인된 재조합(reassortant) 1주(SPL087A)를 제외하고는 모두 유전형 B에 속함을 확인할 수 있었다. 국내의 경우, 이 연구 및 기존에 보고된 바이러스 총 14주에 대한 유전형 분포를 조사한 결과, 총 4가지 유전형(A, B, D, F)이 확인되었고, 그 중 대부분은 유전형 B에 속했으며, 예외적으로 사람 유래 분리주인 CB2 주가 유전형 A에 속함을 처음으로 확인하였다(Table 3). 그러나 이 연구에 이용된 국내 분리주의 수가 적은 제한점으로 인해 보다 정확한 국내 SFTS 유행 양상 파악을
Table 2. Comparison of the full-length nucleotide sequence (ORF region) and deduced amino acid sequence identities between the tick-derived KAGWT strain and other human-derived Korean SFTSV strains
(Ref: PLoS Neglected Tropical Diseases. 2017;11(9):e0005893)
Genotype Strain
Sequence identity (%) of segment
L M S (NP) S (NSs)
Nucleotide Amino acid Nucleotide Amino acid Nucleotide Amino acid Nucleotide Amino acid
A CB2 96.3 99.5 94.0 98.5 95.8 99.6 95.0 99.3 B CB1 96.6 99.5 96.2 99.1 95.7 99.6 95.9 98.6 CB3 96.5 99.5 96.1 99.2 94.9 99.2 95.9 98.6 KAGWH3 99.9 100.0 99.8 99.8 99.9 100.0 99.8 100.0 KAGBH6 99.3 99.9 99.1 99.3 97.8 100.0 98.3 99.7 KAGBH5 98.4 99.9 98.4 99.3 99.3 100.0 98.4 99.7 KAGNH4 97.8 99.7 97.8 99.2 98.4 100.0 98.0 99.3 KAGNH 97.5 99.8 97.3 99.3 97.0 100.0 97.1 99.7 KAJNH2 96.7 99.7 96.2 99.1 95.8 100.0 96.1 99.3 KADGH 96.5 99.4 95.8 98.8 96.2 100.0 96.5 99.7 KACNH3 96.4 99.3 95.9 99.4 95.4 100.0 95.8 99.0 D KASJH 95.9 99.7 93.9 98.5 95.7 99.2 95.1 98.3 F Gangwon/ Korea/2012 96.1 99.5 94.0 98.4 95.7 99.2 94.8 98.6
Figure 2. Phylogenetic analysis based on the complete nucleotide sequences of L segment of SFTSV strains using the Maximum Likelihood (ML) method based on the Kimura 2-parameter model. The numbers on the branches indicate bootstrap percentages based on 1,000 replications, and the scale bar indicates the nucleotide substitutions per site. The phylogenetic branches were supported with greater than 70% bootstrap values. The red, blue, brown, green, purple, and grey branches were designated as SFTSV strains belonging to the genotypes A, B, C, D, E, and F, respectively. The tick- and human-derived Korean SFTSV strains analyzed in this study are marked with red and blue closed circles, respectively. (Ref: PLoS Neglected Tropical Diseases 2017.11(9) e0005893)
Figure 3. Phylogenetic analysis based on the complete nucleotide sequences of M segment of SFTSV strains using the Maximum Likelihood (ML) method based on the Kimura 2-parameter model. The numbers on the branches indicate bootstrap percentages based on 1,000 replications, and the scale bar indicates the nucleotide substitutions per site. The phylogenetic branches were supported with greater than 70% bootstrap values. The red, blue, brown, green, purple, and grey branches were designated as SFTSV strains belonging to the genotypes A, B, C, D, E, and F, respectively. The tick- and human-derived Korean SFTSV strains analyzed in this study are marked with red and blue closed circles, respectively. (Ref: PLoS Neglected Tropical Diseases 2017.11(9) e0005893)
Figure 4. Phylogenetic analysis based on the complete nucleotide sequences of S segment of SFTSV strains using the Maximum Likelihood (ML) method based on the Kimura 2-parameter model. The numbers on the branches indicate bootstrap percentages based on 1,000 replications, and the scale bar indicates the nucleotide substitutions per site. The phylogenetic branches were supported with greater than 70% bootstrap values. The red, blue, brown, green, purple, and grey branches were designated as SFTSV strains belonging to the genotypes A, B, C, D, E, and F, respectively. The tick- and human-derived Korean SFTSV strains analyzed in this study are marked with red and blue closed circles, respectively. (Ref: PLoS Neglected Tropical Diseases 2017.11(9) e0005893)
위해서는 지속적인 감시 및 광범위한 분자유전학적 분석이 필요할 것으로 사료된다.
맺는 말
이 연구에서는 국내에서 처음으로 분리, 동정된 1주의 참진드기 유래 SFTS 바이러스(KAGWT)의 전체 염기서열을 확인하였고, 사람 유래 분리주와 분자유전학적 특성을 비교·분석하였다. 전체 염기서열을 기초로 한 유전자 염기서열 및 계통 유연관계 분석 결과, 참진드기 및 사람 유래 SFTS 바이러스간의 연관성을 확인할 수 있었고, 국내에는 적어도 4가지 유전형(A, B, D, F)이 분포함을 확인하였다. 이 연구 결과는 SFTS 바이러스의 분자역학, 유전학적 다양성에 대한 기초자료로써 백신 및 치료제 개발뿐만 아니라 공중보건학적 관리정책에 유용한 정보로 활용될 수 있을 것으로 생각된다.이 원고는 PLoS Neglected Tropical Diseases. 2017;11(9):e0005893[9]에 게재된 논문을 요약, 재편집한 내용입니다.
참고문헌
1. Yu, X. J., et al. Fever with thrombocytopenia associated with a novel bunyavirus in China. N Engl J Med. 2011;364(16):1523-32.
2. Kim, K. H., et al. Severe fever with thrombocytopenia syndrome, South Korea, 2012. Emerg Infect Dis. 2013;19(11):1892-4.
3. Takahashi, T., et al. The first identification and retrospective study of severe fever with thrombocytopenia syndrome in Japan. J Infect Dis. 2014;209(6):816-27.
4. Gai, Z., et al. Person-to-person transmission of severe fever with thrombocytopenia syndrome bunyavirus through blood contact. Clin
Infect Dis. 2012;54(2):249-52.
5. 질병관리본부, 감염병웹통계시스템, Available URL:http://www.is.cdc. go.kr/dstat/index.jsp.
6. Yun, S. M., et al. First isolation of severe fever with thrombocytopenia syndrome virus from Haemaphysalis longicornis ticks collected in severe fever with thrombocytopenia syndrome outbreak areas in Table 3. Genotype distribution of SFTSV strains in China, Japan, and South Korea
Geographical origin No. Total strains* Genotype No. of SFTSV strains in each genotype
L segment M segment S segment
China 139 A 30 30 31 B 17 17 17 C 1 1 0 D 26 21 26 E 2 2 2 F 63 68 63 Japan 43 B 42 42 42 C 1 0 1 D 0 1 0 South Korea 14 A 1 1 1 B 11 11 11 D 1 1 1 F 1 1 1
(Ref: PLoS Neglected Tropical Diseases. 2017;11(9):e0005893) *In total, 196 SFTSV strains containing full-length sequences of L, M, and S segments were included in the analysis.
the Republic of Korea. Vector Borne Zoonotic Dis. 2016;16(1):66-70. 7. Fu, Y., et al. Phylogeographic analysis of severe fever with
thrombocytopenia syndrome virus from Zhoushan Islands, China: implication for transmission across the ocean. Sci Rep.
2016;6:19563.
8. Guu, T. S., et al. Bunyavirus: structure and replication. Adv Exp Med
Biol. 2012;726:245-66.
9. Yun, S. M., et al. Molecular genomic characterization of tick- and human-derived severe fever with thrombocytopenia syndrome virus isolates from South Korea. PLoS Negl Trop Dis. 2017;11(9):e0005893.
연구단신, Brief report
네트워크 생물학과 질병 연구
질병관리본부 국립보건연구원 유전체센터 생명정보연구과 허형삼, 조성범* *교신저자 : sbcho@korea.kr, 043-719-8850들어가는 말
현대 분자생물학의 주요 연구방법은 생체 구성 분자 를 하나씩 분리하여 개별적인 기능을 밝히는 환원주의적 방법을 통해 각 분자의 생명현상을 규명하였다. 예를 들어 특정 유전자의 발현을 억제함으로써 발생되는 반응을 통해 해당 유전자의Network biology and disease research
Heo Hyoung-Sam, Cho Seong BeomDivision of Bio-Medical Informatics, Center for Genome Science, KNIH, KCDC
The functional identification of biological molecules at the system level is known as systems biology, which aims to uncover biological mechanisms through analysis of the biological elements within a system by identifying the connectivity of a large number of variables involved in a particular biological phenomenon. Systems biology is a field of research that identifies relationships among biological molecules such as genes and proteins, and incorporates the relationships and interpretations at the system level. One of the most effective methods that can model the interactions among biological molecules is network biology.
Multiple genes and their interactions are involved in disease mechanisms. This network biological view of disease pathology is now beginning to guide translational research. Network-based methods play several roles in disease research, such as (1) prioritizing disease-related genes, (2) identifying the disease module, and (3) stratification of complex diseases. In this review, we introduce the basic concepts of network science and network biology, and discuss the application of network biological approaches in disease research.
Keywords: Bioinformatics, Connectome, Disease, Network Biology, Systems Biology
기능을 유추하고 규명하였다. 그러나 수많은 생체 분자들은 복잡한 상호작용을 하고 있는 유기적인 시스템이며, 따라서 생명 현상의 규명을 위한 기존의 환원주의적 연구방법은 명확한 한계를 포함하고 있다. 인간 게놈 프로젝트(Human Genome Project, HGP) 이후 염기서열 해독의 고속화 및 자동화가 진행되었고 대용량 오믹스 자료의 생산 및 분석이 가능하게 되었으며, 이를
통해 점차 분자생물학은 다양한 오믹스 기반의 자료 분석을 통해 시스템 수준에서 생체 구성 분자들의 기능을 유추하고 규명하는 전체주의적 패러다임으로 전환되고 있다. 시스템 생물학이란 특정 생명 현상에 관여하는 생체 분자의 수많은 상호작용 변수들의 연결을 규명함으로써 생체 시스템 내부의 근원적인 작용기전을 이해하고자 하는 학문분야로 유전자, 단백질 등과 같은 생체 분자들 간의 관계를 규명하고, 규명된 관계를 시스템 수준에서 통합, 해석하는 연구 분야이다. 시스템 생물학 기반의 연구를 위해서는 대용량 오믹스 자료의 생산과 오믹스 자료 기반의 생체 분자들의 관계를 예측, 규명하고 그 결과를 효과적으로 모델링하여 생체 분자의 기능과 작용 기전을 규명할 수 있는 연구 방법이 필요하다. 이러한 생체 분자들 간의 상호관계를 가장 효과적으로 모델링 할 수 있는 연구 기반 중 하나가 네트워크다. 네트워크를 이용한 연구 방법은 시스템 생물학에서 가장 기본적이고 핵심적인 연구 방법이며, 네트워크 과학을 이용한 생명과학 분야 적용에 관한 연구 방법을 네트워크 생물학이라고 한다. 연구자들은 네트워크 생물학을 이용하여 시스템 수준의 관찰을 통해 생체 분자들의 생명현상을 규명하고, 각 생체 분자들의 창발적 기능(각 생체 분자들의 단일 기능에서는 발견 할 수 없는 기능이 시스템 수준의 기능 발현에서 관찰 되는 현상)을 규명하고자 한다. 이번 글에서는 네트워크 생물학의 응용 분야 중 질병 연구를 중심으로 실제 연구 적용 사례를 살펴보고, 현재 질병연구 분야 네트워크 생물학의 동향에 대해 소개하고자 한다.
몸 말
생물학적 네트워크의 구성과 종류
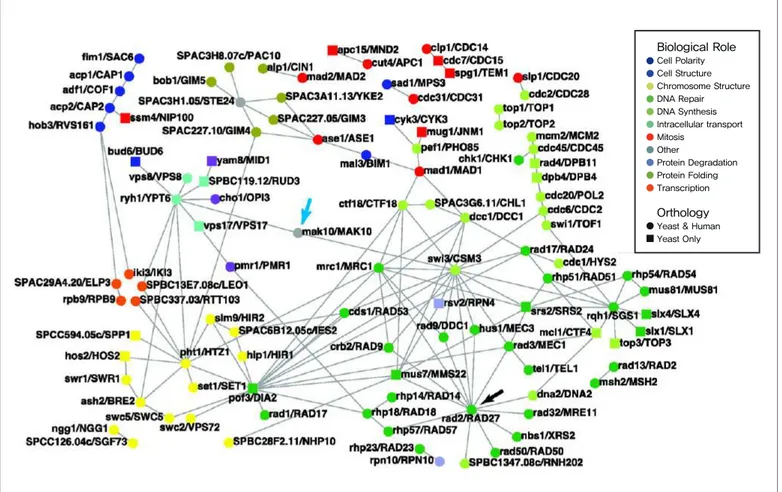
기 본 적 인 네 트워 크 의 구 성 은 네 트워 크 구 성 요 소 를 표현하는 정점(node 또는 vertex)과 구성 요소들 간의 관계를 표현하는 간선(edge 또는 arc)으로 구성되며(Figure 1), 생물학적 네트워크에서는 유전자, 단백질과 같은 생체분자를 정점으로, 생체분자들 간의 상호작용을 간선으로 표현한다.가장 많은 연구와 응용이 진행 중인 생물학적 네트워크의 종류는 유전자 상호작용 네트워크(genetic interaction network), 단백질-단백질 상호작용 네트워크(protein-protein interaction network), 전사 조절 네트워크(transcriptional regulatory network)
제시하였다. 유전자 상호작용 네트워크에서 두 유전자 간의 유전자 상호작용은 실제 두 유전자가 기능적 관계를 공유하고 있음을 의미한다(Figure 2).
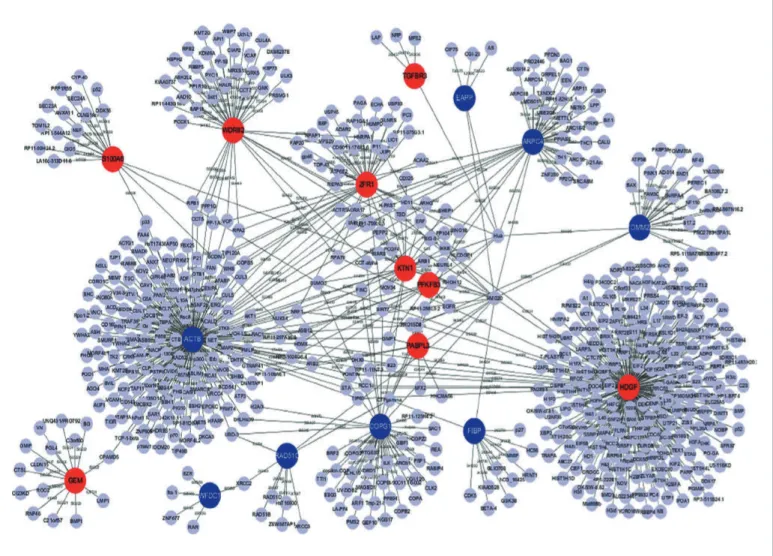
(2) 단백질-단백질 상호작용 네트워크 (protein-protein interaction network)
단백질은 생체분자 중 생체 구성성분 또는 세포 내의 각종 화학반응의 촉매물질로써 중요한 생체분자이며, 단백질-단백질 상호작용의 규명은 단백질-단백질과 상호작용하는 다른 단백질-단백질을 발견하거나 그들의 신호 전달과정을 규명하는, 분자생물학에서 중 요한 연 구 방 법 중 하 나 이다. 단 백 질 - 단 백 질 상 호작 용 네트워크에서 정점은 단백질, 간선은 단백질 간의 물리적 결합 또는 접촉을 표현한다(Figure 3). 단백질-단백질 상호작용을 규명하기 위해 여러 실험적인 방법이 보고되어 있으며, 일반적으로 가장 등이 있으며, 네트워크를 이용한 대사 반응(metabolic reaction)과
신호 전달(signal transduction)을 포함하는 생물학적 경로(biological pathway)의 표현이 대표적이다.
(1) 유전자 상호작용 네트워크 (genetic interaction network)
두 유 전자 간의 유 전자 상 호작용 은 일반적 으 로 이들 돌연변이의 표현형이 각각의 돌연변이 개체에서 예상되었던 것과 다르게 표현된다. 지난 수년 간 효모(Saccharomyces cerevisiae) 종을 이용한 합성 유전자 배열(synthetic genetic array) 기술과 유전자 발현 억제를 이용한 기능 연구를 통해 효모 종의 유전자 상호작용 네트워크의 게놈 수준 연구가 완료되었다. 그 결과는 유전자 상호작용의 기능적 해석과 유전 및 분자 상호작용의 관계, 이를 활용한 유전자 기능의 추론 등에 관한 여러 가지 질문을 Biological Role Cell Polarity Cell Structure Chromosome Structure DNA Repair DNA Synthesis Intracellular transport Mitosis Other Protein Degradation Protein Folding Transcription Orthology
Yeast & Human Yeast Only
많이 사용되는 실험적인 방법은 효모단백질잡종법(yeast two-hybrid system)이다. 효모단백질잡종법은 효모에서 발현되는 전사인자와 효모의 생육에 필수적인 단백질을 생산하는 유전자를 표지자로 이용하여 원하 는 단백질에 특정의 결합 단백질이 결합되면 유전자발현의 하부단계(downstream)의 필수 유전자가 발현하여 단백질을 생산함으로써 원하는 단백질과 결합하는 미지의 단백질을 찾아내는 시스템이다. 효모단백질잡종법의 장점은 유전자 재조합기술을 기반으로 함으로 간편하며, 단백질 특성과 관계없이 실험을 대규모로 진행 가능하여 단백질 상호작용 분석에 용이하고 단백질의 구조 변형 없이 약한 상호작용도 확인할 수 있다. 반면에 표지 유전자의 발현을 확인하여 단백질 상호작용을 간접적으로 측정하므로 위양성(false positive) 확률이 높다. (3) 전사 조절 네트워크
(transcriptional regulatory network)
유전자 발현 조절, 전사조절인자 연구는 기존 개별 유전자와 전사조절인자와의 조절 기작 및 발현 연구를 벗어나 각 유전자의 발현 조절 기전 내에서 상호연관성을 규명하는 시스템 수준의 연구로 발전하였다. 전사조절인자와 전사조절요소의 상호작용을 규명하기 위한 방법으 로는 전사조절인자 에 특이적 항 체를 이용하여 전사조절인자와 전사조절요소의 복합체를 크로마틴 면역침전(chromatin immunoprecipitation, ChIP) 후 전사조절요소를 분리, 동정하는 방법이 사용되고 있다. 전사조절요소의 서열을 분석하는 방법에 마이크로어레이가 사용되는 방법을 ChIP-chip, 차세대서열결정법이 사용되는 방법을 ChIP-seq이라 한다.
단계로 간주 할 수 있다. 생물학적 경로는 생체분자의 상태를 변화시키는 생명현상 또는 특정 요인에 의한 반응을 정의한다. 촉매를 포함한 결합, 활성화, 전이, 열화 및 생화학적인 반응과 사건은 모두 반응으로 간주 할 수 있다.
네트워크 생물학을 이용한 알레르기 질환 연구의 예
질병은 특정 질병요인에 의한 상호작용의 연쇄반응을 통해 발병하며, 이러한 질병 기전은 생물학적 네트워크 내에서 질병 요인에 의해 영향을 받는 질병 관련 생물학적 경로로 한정 할 수 있다. 특정 질병 관련 생물학적 경로는 생물학적 경로의 유사성 또는 공유를 통해 또 다른 질병을 유발 할 수 있다. 생명정보연구과에서는 알레르기 행진(allergic march) 관련 질환인 알레르기 비염(allergic 등 을 표현하며, 간선은 전사 조절의 활성과 저해의 특성을 표현한다(Figure 4).네트워크를 이용한 생물학적 경로(biological
pathway)의 표현
생물학적 경로는 대사 경로(metabolic pathway), 신호 전달 경로(signaling pathway)와 같은 특정 생명현상 또는 특정 요인에 의한 상호작용의 연쇄반응을 뜻한다(Figure 5). 그에 비해 생물학적 네트워크는 생물학적 경로를 포함하는 생명체의 전체 상호작용의 총합으로 생각 할 수 있다. 네트워크를 이용한 생물학적 경로의 표현에서 정점은 생물학적 경로 내의 생체 분자를 표현하며, 간선은 경로와 반응의 매개변수를 표현한다. 반응은 생물학적 경로에서(A)
Figure 6. Overview of analytic process gy Allergic rhinitis Allergic asthma Atopic dermatitis Food allergy Disease DBs Disease Genes
PPI / Genetic network DBs
Association index
Network similarities
Construction of Disease Network
& Network similarities across allergic disease-related molecular networks
AR: module (5) AA: module (5) AD: module (5) FA: module (5)
Network module Correlation test
Network module Network module
Finding network modules among allergic disease genes & Similarity measures across disease networks
Association index
AA -AD
Category Term Count % PValue
GOTERM_BP_FAT GO:0009611~response to wounding 181 22.54047 2.24E- 97 GOTERM_BP_FAT GO:0006952~defense response 167 20.79701 3.94E- 72 GOTERM_BP_FAT GO:0006954~inflammatory response 124 15.44209 1.08E- 71
AA -AR
Category Term Count % PValue
GOTERM_BP_FAT GO:0006952~defense response 73 29.43548 6.47E-42 GOTERM_BP_FAT GO:0006954~inflammatory response 56 22.58065 3.23E-40 GOTERM_BP_FAT GO:0009611~response to wounding 60 24.19355 1.02E-32
AA -AD
Category Term Count % PValue
GOTERM_BP_FAT GO:0006952~defense response 71 38.79781 2.24E-49 GOTERM_BP_FAT GO:0006954~inflammatory response 53 28.96175 1.56E-43 GOTERM_BP_FAT GO:0009611~response to wounding 56 30.60109 7.85E-36
FA-AA
Category Term Count % PValue
GOTERM_BP_FAT GO:0006952~defense response 30 19.23077 5.53E -12 GOTERM_BP_FAT GO:0006954~inflammatory response 22 14.10256 2.11E -11 GOTERM_BP_FAT GO:0009611~response to wounding 26 16.66667 1.98E -10
FA-AD
Category Term Count % PValue
GOTERM_BP_FAT GO:0006952~defense response 29 24.36975 4.64E -14 GOTERM_BP_FAT GO:0006954~inflammatory response 21 17.64706 1.42E -12 GOTERM_BP_FAT GO:0009611~response to wounding 25 21.0084 4.65E -12
FA-AR
Category Term Count % PValue
GOTERM_BP_FAT GO:0006955~immune response 26 33.76623 3.47E -15 GOTERM_BP_FAT GO:0006952~defense response 23 29.87013 3.11E -13 GOTERM_BP_FAT GO:0006954~inflammatory response 17 22.07792 8.26E -12 GOTERM_BP_FAT GO:0009611~response to wounding 18 23.37662 1.36E -09
AR
AA AD
FA
Figure 7. Number of genes and results of functional annotation for shared genes across allergic diseases-related molecular networks Food allergy Asthma Allergic rhinitis Atopic dermatitis Module 1 Module 12 Module 15 Module 8 Module 20 r = 0.13 r = 0.11 r = 0.20 r = 0.118
rhinitis), 천식(asthma), 아토피 피부염(atopic dermatitis), 음식물 알레르기(food allergy) 네 가지 질환에 대하여 네트워크 생물학 방법을 이용하여 질병 기전의 공유를 통한 다른 질병의 발병기전에 관한 연구를 진행하였다. 연구 방법(Figure 6)은 (1) 네 가지 알레르기 질환에 대한 질병 관련 유전자를 공개 데이터베이스를 이용하여 수집하였고, (2) 각 질병 관련 유전자를 이용하여 네 가지 알레르기 질환의 단백질-단백질 상호작용 네트워크를 공개 데이터베이스를 이용하여 구축하였다. 네 가지 알레르기 질환 네트워크를 이용하여 (3) 각 알레르기 질환 네트워크가 공유하는 유전자의 기능을 분석하여 각각 알레르 기 질환 들이 공 유하 는 생 물학적 기능 에 대한 주석(annotation)을 수행하였다. 또한 (4) 네 가지 알레르기 질환 네트워크에서 중요한 기능을 할 것으로 판단되는 상호작용의 집합인 네트워크 모듈을 예측, 추출하여 네 가지 알레르기 질환 각각의 네트워크 모듈 유사도 계산을 통해 각각 알레르기 질환들이 공유하는 네트워크 모듈 기능을 주석달기 하였다. 각 알레르기 질환 네트워크가 공유하는 유전자의 기능을 분석하여 각각 알레르기 질환들이 공유하 는 생물학적 기능 주석달기 분석 결과(Figure 7), 각각 알레르기 질환들은 공통적으로 방어반응과 같은 외부 자 극에 대한 반응 관련 유전자 들 을 공통적으로 포함하고 있으며, 이러한 외부 자극에 의한 결과로 발생하는 표현형인 염증반응 관련 유전자를 포함하고 있었다. 알레르기 질환 네트워크의 네트워크 모듈 추출 및 네트워크 모듈을 이용한 네트워크 유사도 분석 결과에서는 각 알레르기 질환 네트워크의 중요 모듈을 추출하고 각각 알레르기 질환 네트워크의 유사도에 관련성이 높은 네트워크 모듈을 동정하였다(Figure 8). 동정된 각각 알레르기 질환 네트워크의 유사도에 관련성이 높은 네트워크 모듈의 기능 분석을 통해서 특정 알레르기 질환이
Table 1. Biological functions of essential modules across allergic diseases-related molecular networks
Disease Functional term
Food allergy (Module 1) & Asthma (Module 12)
GO:0042221~response to chemical stimulus GO:0010033~response to organic substance GO:0009719~response to endogenous stimulus GO:0048519~negative regulation of biological process GO:0048523~negative regulation of cellular process Asthma (Module 15)
& Allergic rhinitis (Module 8) GO:0042981~regulation of apoptosis
GO:0043067~regulation of programmed cell death GO:0010941~regulation of cell death
GO:0009892~negative regulation of metabolic process GO:0006955~immune response
Allergic rhinitis (Module 8)
& Atopic dermatitis (Module 20) GO:0042221~response to chemical stimulus GO:0050896~response to stimulus
GO:0010033~response to organic substance GO:0048519~negative regulation of biological process GO:0048523~negative regulation of cellular process Atopic dermatitis (Module 20)
& Asthma (Module 15) GO:0042221~response to chemical stimulus GO:0010033~response to organic substance GO:0048519~negative regulation of biological process GO:0050896~response to stimulus
발생하여 다른 알레르기 질환을 유발하는데 관여하는 생물학적 기능을 예측 할 수 있었다(Table 1).
맺는 말
질병의 예방 및 제어를 위해서는 질병 관련 인자의 규명 및 질병 기전의 이해가 필수적이다. 대용량 오믹스 자료의 생물정보학적 연구는 질병 관련 인자의 발견 및 기능 규명에 매우 효율적인 연구 방법이지만 우리가 보유한 질병 관련 정보는 아직까지 매우 제한적인 상황이다. 네트워크 생물학 기반 연구방법의 질병 연구 분야 적용은 이러한 질병 관련 정보의 해석 및 이해에 강력한 힘을 발휘 할 것으로 기대되며, 방대한 질병 기전 연구 분야에 시간적, 물질적 비용 절감 효과를 기대 할 수 있을 것으로 사료된다.참고문헌
1. Albert R. Scale-free networks in cell biology. J Cell Sci. 2005 Nov 1;118(Pt 21):4947-57.
2. Barabási AL, Oltvai ZN. Network biology: understanding the cell's functional organization. Nat Rev Genet. 2004 Feb;5(2):101-13. 3. Ideker T, Krogan NJ. Differential network biology. Mol Syst Biol. 2012
1.1 환자감시 : 전수감시 감염병 주간 발생 현황
(2nd Week)
Table 1. Reported cases of national infectious diseases in Republic of Korea, week ending January 13, 2018 (2nd Week)*
Unit: No. of cases†
Classification of disease‡ Current
week Cum.2018
5-year weekly average
Total no. of cases by year Imported cases of current week : Country(no. of cases) 2017 2016 2015 2014 2013 Group Ⅰ Cholera 0 0 0 5 4 0 0 3 Typhoid fever 4 11 3 129 121 121 251 156 Paratyphoid fever 2 4 1 73 56 44 37 54
Shigellosis 24 34 3 111 113 88 110 294 Philippines(18), India(1)
EHEC 1 2 0 139 104 71 111 61 Viral hepatitis A 63 148 33 4,431 4,679 1,804 1,307 867 Group Ⅱ Pertussis 24 47 2 327 129 205 88 36 Tetanus 1 1 0 34 24 22 23 22 Measles 3 4 0 11 18 7 442 107 Mumps 257 510 331 16,951 17,057 23,448 25,286 17,024 Rubella 5 7 0 20 11 11 11 18
Viral hepatitis B (Acute) 7 13 3 392 359 155 173 117
Japanese encephalitis 0 0 0 9 28 40 26 14 Varicella 1,779 3,750 1,381 80,078 54,060 46,330 44,450 37,361 Streptococcus pneumoniae 36 60 5 518 441 228 36 -Group Ⅲ Malaria 1 4 1 529 673 699 638 445 Scarlet fever§ 402 811 170 22,885 11,911 7,002 5,809 3,678 Meningococcal meningitis 1 1 0 18 6 6 5 6 Legionellosis 7 9 1 198 128 45 30 21
Vibrio vulnificus sepsis 0 0 0 47 56 37 61 56
Murine typhus 0 0 0 20 18 15 9 19 Scrub typhus 40 70 16 10,607 11,105 9,513 8,130 10,365 Leptospirosis 7 8 1 135 117 104 58 50 Brucellosis 8 11 0 27 4 5 8 16 Rabies 0 0 0 0 0 0 0 0 HFRS 13 21 7 604 575 384 344 527 Syphilis 54 87 19 2,154 1,569 1,006 1,015 799 CJD/vCJD 1 5 1 69 42 33 65 34 Tuberculosis 633 1,139 599 28,577 30,892 32,181 34,869 36,089 HIV/AIDS 4 10 5 1,005 1,062 1,018 1,081 1,013
Viral hepatitis C 265 465 - 6,411 - - - - Russia(1)
VRSA 0 0 - 0 - - -
-CRE 160 295 - 5,373 - - -
-Group Ⅳ
Dengue fever 4 8 4 194 313 255 165 252 Thailand(2), Philippines(1),
Malaysia(1)
Q fever 15 35 1 136 81 27 8 11
West Nile fever 0 0 0 0 0 0 0 0
Lyme Borreliosis 5 6 0 46 27 9 13 11
Melioidosis 0 0 0 2 4 4 2 2
Chikungunya fever 0 0 0 6 10 2 1 2
SFTS 0 0 0 270 165 79 55 36
MERS 0 0 - 0 0 185 -
-Zika virus infection 0 0 - 11 16 - -
-Abbreviation: EHEC= Enterohemorrhagic Escherichia coli, HFRS= Hemorrhagic fever with renal syndrome, CJD/vCJD= Creutzfeldt-Jacob Disease / variant Creutzfeldt-Jacob Disease, VRSA= Vancomycin-resistant Staphylococcus aureus, CRE= Carbapenem-resistant Enterobacteriaceae, SFTS= Severe fever with thrombocytopenia syndrome, MERS-CoV= Middle East Respiratory Syndrome Coronavirus.
Cum: Cumulative counts from 1st week to current week in a year.
* The reported data for year 2017, 2018 are provisional but the data from 2013 to 2016 are finalized data.
† According to surveillance data, the reported cases may include all of the cases such as confirmed, suspected, and asymptomatic carrier in the group.
Table 2. Reported cases of national infectious diseases in Repu
blic of Korea, week ending January 13, 2018 (2nd Week)*
U n it: N o . o f c a se Reporting area Cholera Typhoid fever Paratyphoid fever Shigellosis
Enterohemorrhagic Escherichia coli
Viral hepatitis A
Pertussis
Tetanus
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
T otal 0 0 0 4 11 7 2 4 0 24 34 4 1 2 0 63 148 56 24 47 2 1 1 Seoul 0 0 0 0 1 1 0 1 0 3 6 0 0 0 0 13 22 11 0 2 1 0 0 Busan 0 0 0 0 0 0 0 0 0 0 3 0 0 0 0 1 2 1 0 0 0 0 0 Daegu 0 0 0 0 0 0 0 0 0 3 3 0 0 0 0 0 1 1 1 1 0 1 1 Incheon 0 0 0 0 2 1 0 0 0 2 3 1 0 0 0 1 11 4 2 3 0 0 0 Gwangju 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 2 1 0 0 0 0 0 Daejeon 0 0 0 1 1 1 0 0 0 1 1 0 0 0 0 5 7 3 0 0 0 0 0 Ulsan 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 Sejong 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 1 3 0 0 0 Gyonggi 0 0 0 2 3 1 2 2 0 8 8 2 1 1 0 20 45 21 17 34 1 0 0 Gangwon 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 2 4 1 0 0 0 0 0 Chungbuk 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 2 4 2 1 1 0 0 0 Chungnam 0 0 0 0 0 1 0 0 0 4 4 0 0 0 0 11 23 2 0 1 0 0 0 Jeonbuk 0 0 0 0 1 0 0 0 0 0 0 0 0 0 0 2 15 5 1 1 0 0 0 Jeonnam 0 0 0 0 0 1 0 1 0 2 3 1 0 0 0 0 2 2 1 1 0 0 0 Gyeongbuk 0 0 0 0 1 0 0 0 0 0 2 0 0 0 0 3 5 1 0 0 0 0 0 Gyeongnam 0 0 0 1 1 1 0 0 0 1 1 0 0 1 0 2 3 1 0 0 0 0 0 Jeju 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0
e data from 2013 to 2016 are finalized data. e all of the cases such as confirmed, suspected, and asymptomat
ic carrier in the group.
Continued
) Reported cases of national infectious diseases in Republic of
Korea, week ending January 13, 2018 (2nd Week)*
U n it: N Measles Mumps Rubella Viral hepatitis B (Acute) Japanese encephalitis Varicella Malaria Scarlet fever Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§ Current week Cum. 2018 3 4 0 257 510 625 5 7 0 7 13 3 0 0 0 1,779 3,750 2,865 1 4 2 402 0 1 0 26 54 66 1 2 0 0 2 1 0 0 0 210 512 287 0 0 1 61 0 0 0 14 35 51 0 1 0 1 1 1 0 0 0 85 137 189 0 0 0 25 0 0 0 18 29 12 0 0 0 1 1 0 0 0 0 81 180 164 0 0 0 12 1 1 0 12 30 19 0 0 0 0 1 0 0 0 0 106 234 177 0 2 0 15 0 0 0 9 13 63 0 0 0 0 0 0 0 0 0 91 178 71 0 0 0 12 0 0 0 9 18 27 0 0 0 0 0 0 0 0 0 62 108 69 0 0 0 18 0 0 0 12 23 18 0 0 0 0 0 0 0 0 0 50 92 88 0 0 0 27 0 0 0 1 2 3 0 0 0 0 0 0 0 0 0 8 11 16 0 0 0 3 0 0 0 62 115 122 0 0 0 3 3 1 0 0 0 531 1,148 826 1 1 1 91 1 1 0 4 12 29 1 1 0 0 0 0 0 0 0 36 82 117 0 0 0 6 1 1 0 5 13 9 0 0 0 0 0 0 0 0 0 64 136 59 0 1 0 9 0 0 0 10 30 20 0 0 0 0 1 0 0 0 0 68 158 135 0 0 0 12 0 0 0 6 18 71 2 2 0 0 0 0 0 0 0 77 151 139 0 0 0 19 0 0 0 17 23 35 0 0 0 0 0 0 0 0 0 61 128 132 0 0 0 31 0 0 0 12 27 17 1 1 0 1 1 0 0 0 0 69 157 123 0 0 0 21 0 0 0 35 57 52 0 0 0 1 3 0 0 0 0 109 215 209 0 0 0 34 0 0 0 5 11 11 0 0 0 0 0 0 0 0 0 71 123 64 0 0 0 6
e data from 2013 to 2016 are finalized data e all of the cases such as confirmed, suspected, and asymptomat
ic carrier in the group.
Table 2. (
Continued
) Reported cases of national infectious diseases in Republic of
Korea, week ending January 13, 2018 (2nd Week)*
U n it: N o . o f c a se s Reporting area Meningococcal meningitis Legionellosis Vibrio vulnificus sepsis Murine typhus Scrub typhus Leptospirosis Brucellosis
Hemorrhagic fever with renal syndrome
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
T otal 1 1 0 7 9 2 0 0 0 0 0 0 40 70 21 7 8 0 8 11 0 13 21 11 Seoul 0 0 0 2 2 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 Busan 0 0 0 2 2 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 0 0 0 Daegu 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 1 2 0 0 0 Incheon 0 0 0 0 0 0 0 0 0 0 0 0 1 2 0 0 0 0 0 0 0 0 0 Gwangju 0 0 0 0 0 0 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0 0 Daejeon 0 0 0 0 1 0 0 0 0 0 0 0 1 1 1 0 0 0 1 2 0 0 0 Ulsan 0 0 0 0 0 0 0 0 0 0 0 0 1 3 1 0 0 0 1 1 0 0 0 Sejong 0 0 0 0 0 0 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0 0 0 Gyonggi 0 0 0 3 3 1 0 0 0 0 0 0 2 4 2 0 1 0 2 2 0 3 5 Gangwon 0 0 0 0 0 0 0 0 0 0 0 0 0 1 0 0 0 0 0 1 0 0 0 Chungbuk 0 0 0 0 0 0 0 0 0 0 0 0 2 2 0 0 0 0 1 1 0 0 0 Chungnam 1 1 0 0 0 0 0 0 0 0 0 0 4 7 1 0 0 0 2 2 0 1 2 Jeonbuk 0 0 0 0 0 0 0 0 0 0 0 0 2 4 2 0 0 0 0 0 0 0 0 Jeonnam 0 0 0 0 0 0 0 0 0 0 0 0 3 5 5 0 0 0 0 0 0 1 3 Gyeongbuk 0 0 0 0 0 0 0 0 0 0 0 0 2 5 1 0 0 0 0 0 0 0 2 Gyeongnam 0 0 0 0 1 0 0 0 0 0 0 0 20 30 5 7 7 0 0 0 0 7 8 Jeju 0 0 0 0 0 0 0 0 0 0 0 0 1 4 1 0 0 0 0 0 0 0 0
e data from 2013 to 2016 are finalized data. e all of the cases such as confirmed, suspected, and asymptomat
ic carrier in the group.
e data from 2013 to 2016 are finalized data. e all of the cases such as confirmed, suspected, and asymptomat
ic carrier in the group.
e counts from 1st week to current week for 5(3) preceding years
.
Continued
) Reported cases of national infectious diseases in Republic of
Korea, week ending January 13, 2018 (2nd Week)*
U n it: N Syphilis CJD/vCJD Dengue fever Q fever Lyme Borreliosis SFTS
Zika virus infection
Tuberculosis
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 5-year average
§
Current week Cum. 2018 Cum. 3-year average
§
Current week Cum. 2018 Cum. 3-year average
§ Current week Cum. 2018 54 87 32 1 5 0 4 8 7 15 35 0 5 6 0 0 0 0 0 0 -633 1,139 15 26 5 0 2 0 0 2 3 0 0 0 1 2 0 0 0 0 0 0 -97 2 4 2 0 0 0 2 4 0 0 1 0 1 1 0 0 0 0 0 0 -43 1 4 2 0 0 0 1 1 1 1 2 0 0 0 0 0 0 0 0 0 -25 3 7 3 0 0 0 1 1 0 0 0 0 0 0 0 0 0 0 0 0 -29 2 3 1 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0 0 0 -9 4 5 1 0 0 0 0 0 0 1 2 0 0 0 0 0 0 0 0 0 -20 0 0 0 0 0 0 0 0 0 1 4 0 1 1 0 0 0 0 0 0 -13 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 -3 12 18 8 1 1 0 0 0 2 2 14 0 2 2 0 0 0 0 0 0 -149 1 1 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 -31 3 4 1 0 0 0 0 0 0 3 4 0 0 0 0 0 0 0 0 0 -22 4 4 1 0 1 0 0 0 0 4 4 0 0 0 0 0 0 0 0 0 -34 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 -23 3 3 2 0 0 0 0 0 0 1 1 0 0 0 0 0 0 0 0 0 -36 0 3 2 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 -41 4 5 1 0 0 0 0 0 1 1 2 0 0 0 0 0 0 0 0 0 -49 0 0 1 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 0 -9
1.2 환자감시 : 표본감시 감염병 주간 발생 현황
(2nd week)
1. Influenza, Republic of Korea, weeks ending January 13, 2018 (2nd week)
● 2018년도 제2주 인플루엔자 표본감시(17개 시·도 보건환경연구원 및 52개 의료기관) 결과, 의사환자분율은 외래환자 1,000명당 69.0명으로 지난주(72.1명)대비 감소
※ 2017-2018절기 유행기준은 6.6명(/1,000)
Figure 1. Weekly proportion of influenza-like illness per 1,000 outpatients, 2013-2014 to 2017-2018 flu seasons 0 20 10 40 30 60 70 80 90 50 100 36 38 40 42 44 46 48 50 52 2 4 6 8 10 12 14 16 18 20 22 24 26 28 30 32 34 ILI per 1,000 week 2014-2015 2015-2016 2013-2014 2016-2017 2017-2018
2. Hand, Foot and Mouth Disease(HFMD), Republic of Korea, weeks ending January 13, 2018 (2nd week)
●2018년도 제2주 수족구병 표본감시(전국 95개 의료기관) 결과, 의사환자 분율은 외래환자 1,000명당 0.4명으로 전주(0.8명) 대비 감소
※ 수족구병은 2009년 6월 법정감염병으로 지정되어 표본감시체계로 운영
Figure 2. Weekly proportion of hand, foot and mouth disease per 1,000 outpatients, 2013-2018 1 3 5 7 9 11 13 2017 2018 2016 2015 2014 2013 15 17 19 21 23 25 27 29 31 33 35 37 39 41 43 45 47 49 51 week 0 10.0 5.0 20.0 15.0 30.0 35.0 40.0 45.0 50.0 25.0 55.0
Figure 3. Weekly proportion of epidemic keratoconjunctivitis per 1,000 outpatients 0 10 20 30 40 50 60 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39 41 43 45 47 49 51 53 No. of outpatients / 1,000 week 2014 2015 2016 2017 2018
Figure 4. Weekly proportion of acute hemorrhagic conjunctivitis per 1,000 outpatients 0 1 2 3 4 5 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39 41 43 45 47 49 51 53 No. of outpatients / 1,000 week 2014 2015 2016 2017 2018
▣ Waterborne and foodborne disease outbreaks, Republic of Korea, weeks ending January 13, 2018 (2nd week)
●2018년도 제2주 보고기관(전국 254개 보건소)에서 집단발생이 5건 발생하였으며 누적발생건수는 12건(사례수 78명)이 발생함.
1.3 수인성 및 식품매개 감염병 집단발생 주간 현황(2nd week)
▶ 자세히 보기 : 질병관리본부 → 정책/사업 → 감염병감시 → 표본감시주간소식지
Figure 5. Number of waterborne and foodborne disease outbreaks reported by week, 2017-2018 0 5 15 20 25 30 10 1 3 5 7 9 11 13 15 17 19 21 23 25 27 29 31 33 35 37 39 41 43 45 47 49 51 week
2018 2017 Average no. of cases in last 5 years(2012-2016)
35
No. of outbreaks
Unit: No. of cases/sentinels
Cum: Cumulative counts from 1st week to current week in a year
† According to surveillance data, the reported cases may include all of the cases such as confirmed, suspected, and asymptomatic carrier in the group. § Cum. 5-year average is mean value calculated by cumulative counts from 1st week to current week for 5 preceding years.
※ 문의: (043)719-7118, 7132
4. Sexually Transmitted Diseases
, Republic of Korea, weeks ending January 13, 2018 (2nd week)
●2018년도 제2주 보고기관(586개)당 클라미디아 감염증 2.6건, 성기단순포진 2.6건, 임질 1.6건, 첨규콘딜롬 1.6건 발생함.
Gonorrhea Chlamydia Genital herpes Condyloma acuminata
Current week Cum.2018 Cum. 5-year average§ Current week Cum.2018 Cum. 5-year average§ Current week Cum.2018 Cum. 5-year average§ Current week Cum.2018 Cum. 5-year average§ Total 1.6 1.0 2.1 2.6 2.9 2.7 2.6 3.2 3.0 1.6 2.2 2.1
2. Respiratory viruses, Republic of Korea, Weeks ending January 13, 2018 (2nd week)
● 2018년도 제2주 호흡기검체에 대한 유전자 검사(17개 시·도 보건환경연구원 및 전국 52개 참여의료기관)결과, 71.3%의 호흡기바이러스가
검출되었음.
(최근 4주 평균 308개의 호흡기 검체에 대한 유전자 검사결과를 나타내고 있음)
※ 주별통계는 잠정통계이므로 변동가능
- HAdV : human Adenovirus, HPIV : human Parainfluenza virus, HRSV : human Respiratory syncytial virus, IFV : Influenza virus, HCoV : human Coronavirus, HRV : human Rhinovirus, HBoV : human Bocavirus, HMPV : human Metapneumovirus
※ Cum. : the rate of detected cases between December. 10. 2017. – January. 06. 2018, (Average No. of detected cases is 300 in last 4 weeks) ∀ 2017 Cum. : the rate of detected cases between January. 01. 2017. - December. 30. 2017.
Figure 6. Number of specimens positive for influenza by subtype, 2016-2017 to 2017-2018 flu season 0 100 120 140 160 80 60 40 20 0 20.0 10.0 40.0 30.0 60.0 70.0 80.0 90.0 50.0 100.0 120.0 ●2018년도 제2주에 17개 시·도 보건환경연구원 및 52개 의료기관에서 의뢰된 호흡기검체 335중 192건 양성(A/H1N1pdm09 6건, A(H3N2) 79건, B형 107건) 180 200 No. of positives Percent positives (%) week
A(H3N2) A(not subtyped) A(H1N1)pdm09 B Percent positive
34 36373839404142434445464748495051 52 1 2 3 4 5 6 7 8 9 101112131415161718192021222324252627282930313233 35
2017-2018
(week) Weeklytotal
Detection rate (%)
HAdV HPIV HRSV IFV HCoV HRV HBoV HMPV
51 69.5 1.2 1.2 8.9 47.6 5.6 4.4 0.6 0.0 52 88.3 2.1 1.4 5.3 66.8 6.7 3.9 1.1 1.1 1 87.5 3.2 0.0 8.2 67.7 5.7 2.5 0.0 0.0 2 71.3 2.1 0.6 4.5 57.3 5.7 0.6 0.0 0.6 Cum.※ 78.4 2.1 0.8 6.7 59.2 5.9 2.8 0.4 0.4 2017 Cum.∀ 56.6 3.7 6.3 4.6 10.9 4.4 19.4 2.0 5.3
2.2 병원체감시 : 급성설사질환 실험실 표본 주간 감시 현황
(1st week)
◆ Acute gastroenteritis-causing viruses
Week No. of sample No. of detection (Detection rate, %)
Group A Rotavirus Norovirus Enteric Adenovirus Astrovirus Total
2017 50 89 4 (4.5) 36 (40.4) 1 (1.1) 1 (1.1) 42 (47.2)
51 79 6 (7.6) 35 (44.3) 1 (1.3) 1 (1.3) 43 (54.4)
52 76 8 (10.5) 26 (34.2) 0 (0.0) 2 (2.6) 36 (47.4)
2018 1 45 1 (2.2) 11 (24.4) 2 (4.4) 1 (2.2) 15 (33.3)
Cum. 45 1 (2.2) 11 (24.4) 2 (4.4) 1 (2.2) 15 (33.3)
* The samples were collected from children ≤5 years of sporadic acute gastroenteritis in Korea.
◆ Acute gastroenteritis-causing bacteria
Week SampleNo. of
No. of isolation (Isolation rate, %) Salmonella
spp.
Pathogenic
E.coli Shigella spp. V.parahaemolyticus V. choleraeCampylobacter spp. C.perfringens S. aureus B. cereus Total
2017 50 155 3 (1.9) 1 (0.6) 0 (0) 0 (0) 0 (0) 1 (0.6) 4 (2.6) 0 (0) 0 (0) 9 (5.8) 51 165 1 (0.7) 0 (0) 0 (0) 0 (0) 0 (0) 0 (0) 1 (0.6) 2 (1.3) 1 (0.7) 5 (3.0) 52 225 3 (1.3) 3 (1.3) 0 (0) 0 (0) 0 (0) 1 (0.4) 2 (0.9) 4 (1.8) 3 (1.3) 16 (7.1) 2018 1 139 2 (1.4) 0 (0) 0 (0) 0 (0) 0 (0) 1 (0.7) 2 (1.4) 2 (1.4) 1 (0.7) 8 (5.8) Cum. 139 2 (1.4) 0 (0) 0 (0) 0 (0) 0 (0) 1 (0.7) 2 (1.4) 2 (1.4) 1 (0.7) 8 (5.8)
* Bacterial Pathogens ; Salmonella spp., E. coli (EHEC, ETEC, EPEC, EIEC), Shigella spp., Vibrio parahaemolyticus, Vibrio cholerae, Campylobacter spp., Clostridium perfringens, Staphylococcus aureus, Bacillus cereus, Listeria monocytogenes, Yersinia enterocolitica.
* Hospital participating in laboratory surveillance in 2018 (70 hospitals)
▶ 자세히 보기 : 질병관리본부 → 알림 → 주간 질병감시정보
▣ Detection rate of acute gastroenteritis causing virus and bacteria, Republic of Korea, Republic of
Korea, weeks ending January 6, 2018 (1st week)
● 2018년도 제1주 실험실 표본감시(17개 시·도 보건환경연구원 및 70개 의료기관) 급성설사질환 유발 바이러스 검출 건수는 15건(33.3%), 세균
0 20 40 60 80 1 5 9 13 17 21 25 29 33 37 41 45 49 53 Detection rate (%) week
January 6, 2018 (1st week)
● 2018년도 제1주 실험실 표본감시(8개 시·도 보건환경연구원, 전국 60개 참여병원)결과, 새로 검출된 엔테로바이러스 1건(2018년 누적 1건, 양성률 12.5%) 중 무균성수막염 0건(2018년 누적 0건), 수족구병 및 포진성구협염 0건(2018년 누적 0건), 합병증 동반 수족구 0건(2018년 누적 0건), 기타 1건(2018년 누적 1건)임. ◆ Aseptic meningitis ◆ HFMD and HerpanginaFigure 8. Detection rate of enterovirus in HFMD and herpangina patients from 2017 to 2018
◆ HFMD with Complications 0 5 10 20 15 25 1 5 9 13 17 21 25 29 33 37 41 45 49 53 0 4 3 2 1 5 6 7 8 9 10 1 5 9 13 17 21 25 29 33 37 41 45 49 53
2018 Enterovirus detection rate (%) 2017 Enterovirus detection rate (%)
Figure 7. Detection rate of enterovirus in aseptic meningitis patients from 2017 to 2018
2017 Enterovirus detection rate (%) 2018 Enterovirus detection rate (%)
2018 Enterovirus detection rate (%) 2017 Enterovirus detection rate (%)
Detection rate (%)
Detection rate (%)
week
<Table 1>은 지난 5년간 발생한 법정감염병과 2017년 해당 주 발생현황을 비교한 표로, 「Current week」는 2017년 해당 주의 신고건수를 나타내며, 「Cum. 2017」은 2017년 1주부터 해당 주까지의 누계 건수, 그리고 「5-year weekly average」는 지난 5년(2012-2016년) 해당 주의 신고건수와 이전 2주, 이후 2주의 신고건수(총 25주) 평균으로 계산된다. 그러므로 「Current week」과 「5-year weekly average」의 신고 건수를 비교하면 해당 주 단위 시점과 예년의 신고 수준을 비교해 볼 수 있다. 「Total no. of cases by year」는 지난 5년간 해당 감염병 현황을 나타내는 확정 통계이며 연도별 현황을 비교해 볼 수 있다.
예) 2017년 12주의 「5-year weekly average(5년간 주 평균)」는 2012년부터 2016년의 10주부터 14주까지의 신고 건수를 총 25주로 나눈 값으로 구해진다.
* 5-year weekly average(5년 주 평균)=(X1 + X2 + … + X25)/25
10주 11주 12주 13주 14주 2017년 해당 주 2016년 X1 X2 X3 X4 X5 2015년 X6 X7 X8 X9 X10 2014년 X11 X12 X13 X14 X15 2013년 X16 X17 X18 X19 X20 2012년 X21 X22 X23 X24 X25
<Table 2>는 17개 시·도 별로 구분한 법정감염병 보고 현황을 보여 주고 있으며, 각 감염병별로 「Cum, 5-year average」와 「Cum, 2017」을 비교해 보면 최근까지의 누적 신고건수에 대한 이전 5년 동안 해당 주까지의 평균 신고건수와 비교가 가능하다. 「Cum, 5-year average」는 지난 5년(2012-2016년) 동안의 동기간 신고 누계 평균으로 계산된다.
<Table 3>은 표본감시 감염병에 대한 신고현황으로, 최근 발생양상을 신속하게 파악하는데 도움이 된다.
「주간 건강과 질병, PHWR」은 질병관리본부에서 시행되는 조사사업을 통해 생성된 감시 및 연구 자료를 기반으로 근거중심의 건강 및 질병관련 정보를 제공하고자 최선을 다할 것이며, 제공되는 정보는 질병관리본부의 특정 의사와는 무관함을 알립니다. 본 간행물에서 제공되는 감염병 통계는 「감염병의 예방 및 관리에 관한 법률」 에 의거, 국가 감염병감시체계를 통해 신고된 자료를 기반으로 집계된 것으로 집계된 당해년도 자료는 의사환자 단계에서 신고된 것이며 확진 결과시 혹은 다른 병으로 확인 될 경우 수정 될 수 있는 잠정 통계임을 알립니다. 「주간 건강과 질병, PHWR」은 질병관리본부 홈페이지를 통해 주간 단위로 게시되고 있으며, 정기적 구독을 원하시는 분은 kcdc215@korea.kr로 신청 가능합니다. 이메일을 통해 보내지는 본 간행물의 정기적 구독 요청시 구독자의 성명, 연락처, 직업 및 이메일 주소가 요구됨을 알려 드립니다. 「주간 건강과 질병」 발간 관련 문의: kcdc215@korea.kr/ 043-249-3028/3003 창 간 : 2008년 4월 4일 발 행 : 2018년 1월 18일 발 행 인 : 정은경 편 집 인 : 박도준 편집위원 : 최영실, 김기순, 최병선, 조신형, 조성범, 김봉조, 구수경, 김용우, 이동한, 조은희, 이은규, 신영림, 김청식, 전경아, 권효진