서 론
B형 간염 바이러스(Hepatitis B virus, HBV)는 간경화와 간 암의 주요한 원인인자로 우리나라 성인의 약 5%가 HBV 보유자 이다[1, 2]. 만성 B형 간염 후 대략 1년에 2-7% 정도가 간경화
로 진행하고, 이중 1.5% 정도가 간암으로 진행하는 것으로 알려 져 있는데[3], 간암의 진행 정도는 HBV의 유전자형에 따라 다소 다르다는 보고가 있다[4]. HBV 유전자형은 HBV 유전체의 8%
이상에서 변이를 보일 때 다른 유전자형으로 분류되며, 현재 A- H형이 보고되었다[5]. 한국인을 대상으로 한 저자들의 이전 연 구에서 한국인은 거의 대부분 C형 유전자형을 보여, 임상적으 로 HBV 유전자형 검사는 효용성이 없다고 보고하였다[6].
최근 같은 HBV 유전자형 내에서도 아형에 따라 다른 임상적 인 경과를 보인다는 보고가 있어, 유전자아형에 대한 관심이 증 가하고 있다. HBV 유전체 염기서열에서 4-8%의 차이를 보일 때 서로 다른 유전자아형(subgenotype)으로 분류된다[7, 8]. A
53 DOI 10.3343/kjlm.2009.29.1.53
53 53 53 Received :November 18, 2008 Manuscript No :KJLM2199 Revision received :December 8, 2008
Accepted :December 18, 2008
Corresponding author :Heung-Bum Oh, M.D.
Department of Laboratory Medicine, University of Ulsan College of Medicine, Asan Medical Center, 388-1 Poongnap-2-dong, Songpa-gu, Seoul 138-736, Korea
Tel : +82-2-3010-4505, Fax : +82-2-478-0884 E-mail : hboh@amc.seoul.kr
치료 중인 만성 B형 간염 한국인 환자의 B형 간염 바이러스 유전자아형 및 혈청형 분석
Subgenotype and Serotype Analysis of Hepatitis B virus in Korean Chronic Hepatitis B Patients Under Treatment
Choong-Hwan Cha, M.D.
1, Yong-Hak Sohn, M.D.
2, Sun-Young Ko, M.D.
2, and Heung-Bum Oh, M.D.
2Department of Laboratory Medicine1, University of Ulsan College of Medicine, Gangneung Asan Hospital, Gangneung;
Department of Laboratory Medicine2, University of Ulsan College of Medicine and Asan Medical Center, Seoul, Korea 차충환1∙손용학2∙고선영2∙오흥범2
울산의대 강릉아산병원 진단검사의학과1, 울산의대 서울아산병원 진단검사의학과2
Background : Hepatitis B virus (HBV) detected in Korean patients almost belongs to genotype C, which is subdivided into subgenotype C1 (or Cs) and C2 (or Ce). It was recently reported that the risk of hepatocellular carcinoma is different between subgenotype C1 and C2. Thus, we studied the distribution of subgenotypes of HBV in Korean chronic hepatitis B (CHB) patients.
Methods : Specimens of 421 patients, who were diagnosed as CHB and underwent antiviral treat- ment, were used. After sequence analysis for HBV S gene, subgenotype was identified through phy- logenetic analysis. Utilizing the same sequence data, the distribution of serotypes was also investi- gated.
Results : Among 421 patient specimens, genotype C was found in 419 (99.5%) and genotype B in 2 (0.5%). Among the genotype C strains, 417 strains were C2 subgenotype and 2 strains were mixed subgenotypes. However, C2 was evidently found even in the mixed sequences. Serotypes of 419 HBV with genotype C were classified as follows: adr, 385 (91.9%), adw, 22 (5.3%), ayr, 2 (0.4%) and mixed serotype, 10 (2.3%). Serotype of both HBV with genotype B was adw.
Conclusions : It was found that HBV detected in Korean CHB patients under treatment almost all belong to the C2 (Ce) genotype. (Korean J Lab Med 2009;29:53-8)
Key Words : Hepatitis B virus, Genotype, Subgenotype, Serotype
형 유전자형은 A1, A2, A3으로, B형 유전자형은 B1 (Bj), B2 (Ba), B3, B4, B5으로 나뉘어진다[9]. 한국인 감염자에서 대부분 검 출되는 C형 유전자형은 크게 C1 (혹은 Cs, 남아시아에 주로 분 포)형과 C2 (혹은 Ce, 동아시아에 주로 분포)형으로 아형이 구 별된다[10, 11]. C1형과 C2형의 임상적인 의미에 대한 최근 연 구에서 C2형이 C1형에 비해 간암 위험도가 높다는 보고가 있었 다[12].
한국인은 대부분 C형 유전자형 HBV를 가지고 있다는 보고 는 있었지만, 아직까지 유전자아형의 분포를 보고한 연구는 없 었다. 유전자아형에 대한 정보를 알 수 있다면 추후 임상적 의 의에 따라 치료 및 예후 측면에서 중요한 정보가 될 수 있을 것 이다. 이에 저자들은 HBV S 유전자 부위에 대한 염기서열 분석 을 통하여 한국인에 존재하는 HBV 유전자아형의 분포를 알아 보고자 하였다.
대상 및 방법
1. 대상울산의대 서울아산병원에서 만성 B형 간염진단 하에 치료받 고 있는 환자 중 2008년 3월부터 6월까지 HBV 항바이러스제 내성검사를 실시한 421명의 환자 검체를 대상으로 하였다.
2. HBV S 유전자 염기서열 분석
HBV S 유전자와 중합효소(polymerase) 유전자에 해당하는 염기서열 중 611 염기서열(C형 유전자형에서 349-959번째 염 기서열 부위)을 직접염기서열법으로 분석하였다. 이 부위는 아 미노산 서열로 중합효소 중 rt71에서 rt273번째 아미노산에 해 당하는 부위이며, S 단백질의 63번째에서 마지막 263번째까지 아미노산을 포함하였다. 염기서열 분석은 BioCore HBV DR sequencing kit (BioCore Co., Seoul, Korea)으로 중합효소 연쇄반응(polymerase chain reaction, PCR)을 수행 후 염기서 열 분석을 사용하였으며 방법을 간략히 기술하면 다음과 같다.
환자 혈청 200 μL에서 바이러스 핵산 추출키트인 Quick- Gene DNA Tissue Kit S (Fujifilm, Tokyo, Japan)을 사용하 여 HBV DNA를 분리한 후 S 단백질의 63번째 아미노산에서 마지막 아미노산 부분을 포함하도록 PCR을 이용하여 증폭하였 다. 반응액은 6 μL의 BioCore HBV DR sequencing kit (Bio- Core Co.)에 4 μL의 분리한 DNA를 첨가하여 총량 10 μL로 반 응하였다. PCR은 DNA Engine Dyad (Bio-rad, Hercules,
CA, USA)을 이용하여 95℃에서 5분간 전변성 반응 후 변성반 응 94℃ 60초, 결합반응 61℃ 60초, 연장반응 72℃ 90초를 40 회 반복한 후 72℃에서 5분간 추가 연장반응을 시행하였다. PCR 의 산물은 2% agarose gel (Promega, Madison, WI, USA)에 5 μL를 loading 후 전기영동하여 966 bp의 PCR 산물을 확인 하였다.
잔존하는 PCR 시발체와 dNTP들을 제거하기 위해 10X buffer (670 mM glycine-KOH, 67mM MgCl2, 10 mM dithiothreitol [DTT]) 0.8 μL, Shirmp Alkaline Phosphatase (SAP) (Pro- mega) 1 μL, Exonuclease (Fermentas, Burlington, Ontario, Canada) 0.2 μL와 3차 멸균 증류수 4 μL를 첨가하여 37℃에서 30분간 반응 하였으며 SAP와 Exonuclease의 비활성화를 위해 85℃에서 15분간 반응을 실시하였다. PCR 산물에 염기서열 분 석용 dye를 붙여주기 위해 20-100 ng/μL로 희석한 PCR 산물 1 μL, 3차 증류수 2 μL, 2 pmol/μL 농도의 염기서열 분석 시발 체(CATCCTGCTGCTATGCCTC) 1 μL 및 Big-Dye 3.1 (Applied Biosystems, Foster City, CA, USA) 용액 2 μL를 혼합하여 96℃
에서 10초, 결합반응 50℃ 5초, 연장 및 label 반응 60℃ 4분을 25회 반복하였다. 염기서열분석반응 후 33 μL의 magnetic re- sin (Promega)을 사용하여 반응하지 않고 남은 산물을 제거하 고 DNA 자동분석기 ABI 3130xl Genetic Analyzer (Applied- biosystems)에 넣어 염기서열을 분석하였다.
3. 유전자형, 유전자아형 분석
이미 알려진 각 유전자형 및 아형의 염기서열과 각 검체의 염 기서열을 계통발생학적으로 비교하여 유전자형 및 아형을 결정 하였다. 비교에 사용된 HBV strain은 Norder 등이 분석한 결과 를 참조하여 National Centers for Biotechnology Informa- tion (http://www.ncbi.nlm.nih.gov/) 염기서열 데이터베이스 를 통해서 얻었으며, 각 유전형별로 사용된 염기서열의 접근번호 (accession number)는 다음과 같다[13]. A형의 경우 L13994, M57663, S50225, B형의 경우 D00329, D00330, D00331, D23678, D50521, C1 아형의 경우 AB031262, AB- 074755, AB111946, AB112348, AB112408, AF473543, AY- 217376, C2 아형의 경우 AF182805, AF411411, AF458664, AY247031, D23681, D50520, X14193, D형의 경우 L27106, M32138, X0- 2496, E형의 경우 X75657, X75664, AB091256, F형의 경우 X69798, X75658, X75663, G형의 경우 AB056515, AF405706, AF160501, H형의 경우 AB059659, AB059660, AB064315이 었다. 분석은 European Bioinfomatics Institute (http://www.
ebi.ac.uk/)의 공개프로그램인 ClustalW을 이용하였고 cluster- ing 방법은 UPGMA (unweighted pair group method with arithmetic mean)를 사용하였다. 또한 공개프로그램인 MEGA (Molecular Epidemiological Genetic Analysis) ver 4.1을 이 용하여 유전자 아형의 결과를 재확인하였다[14].
염기서열 분석 시 한 위치에서 두 개의 염기서열이 혼재되어 관찰되는 경우에는 C형 유전자형 염기서열인 X04615과 가장 유사한 염기서열을 먼저 선정하고 나머지를 또 다른 하나의 염 기서열로 간주하고 분석하였다. 두 개의 염기서열의 유전자형 및 아형이 일치하는 경우 한 개의 유전자형 및 아형으로 판정하 였으며, 두 개의 염기서열의 분석결과가 다른 경우에는 유전자 형 및 아형을 각각 판정하여 혼합형으로 명시하였다.
4. 염기서열의 상이율 분석
계통발생학적 비교에 사용되었던 A-H 유전자형 염기서열과 환자에서 발견된 HBV 염기서열들 간의 그룹내 및 그룹 간 평균 상이율을 구하였다. 유전자(아)형이 알려진 NCBI 염기서열을 비교군으로 선정하였으며, C형을 제외한 경우 각 유전자형에 따라 7개의 그룹으로, C형은 C1형과 C2형의 2가지 그룹으로 나누어 분석에 이용하였다. 염기서열 정렬 후에 염기서열 간에 차이가 있는 염기서열의 개수를 분자로, 전체 611 염기서열의 수를 분모로 하여 상이율을 계산하였으며, MEGA 프로그램의 p-distance 프로시져를 이용하여 동일 그룹내 그리고 각 그룹 간 각 쌍의 상이율의 평균값을 계산하였다.
5. 혈청형 분석
각 검체의 염기서열 중 S 유전자의 122번째와 160번째 아미 노산 서열을 이용하여 혈청형의 빈도를 알아보았다[15]. 122번 째 아미노산이 K (lysine)인 경우d, R (arginine)인 경우y로, 160번째 아미노산이 K인 경우w, R인 경우r로 판정하였다.
결 과
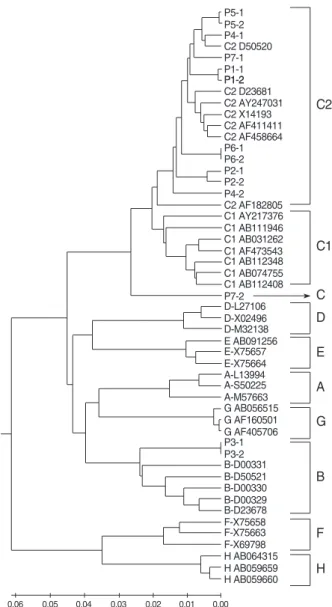
1. 유전자형 및 유전자아형유전자형은 B형 2명(0.5%), C형 419명(99.5%)으로 분석되었 다. 유전자아형 분석 결과 C형 중 417명(99.5%)은 C2 (Ce)형이 었다. 2명은 혼합 염기서열이 각각 14개 및 18개 부위에 존재하 였는데, 모두 C2형과 C형 아형으로 분류되지 않는 유전자형의
혼합이었다(Fig. 1).
2. 염기서열 상이율
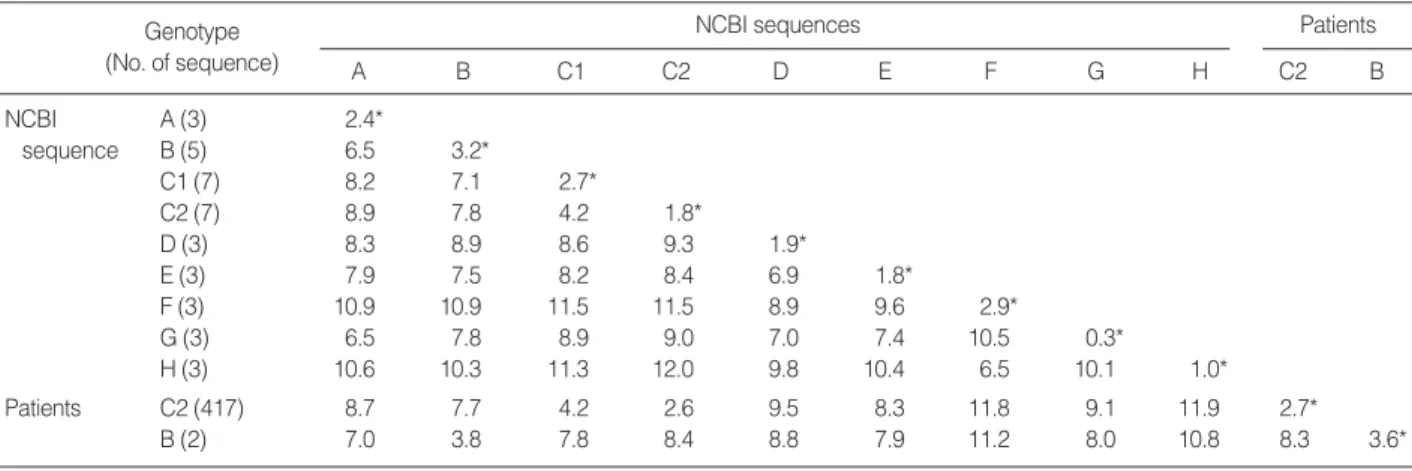
환자에서 검출된 HBV가 C2형 유전자아형으로 분석된 417명 의 유전자아형 내 평균 염기서열 상이율은 2.7%였다. B형 유전 자형으로 나온 2명의 유전자형내 염기서열 상이율은 3.6%이었 다. C2형 유전자아형을 가진 환자들은 A에서 H 유전자형과 평
Fig. 1. Phylogenetic tree of HBV S gene of 7 patients and 35 HBV sequences of NCBI database representing every genotype and C1/C2 subgenotype. Each of the first letter A to H means HBV (sub)genotype and followings are NCBI accession numbers.
Examples of patents’ HBV sequences are shown as P1, P2, etc.
A sequence with mixed peaks was subdivided into two sequences like P1-1/P1-2 in P1 and P2-1/P2-2 in P2 according to similarity to reference X04615.
P5-1
C2
C1
C D
E
A
G
F
H B
P5-2 P4-1 C2 D50520 P7-1 P1-1 P1-2 P1-2 C2 D23681 C2 AY247031 C2 X14193 C2 AF411411 C2 AF458664 P6-1 P6-2 P2-1 P2-2 P4-2 C2 AF182805 C1 AY217376 C1 AB111946 C1 AB031262 C1 AF473543 C1 AB112348 C1 AB074755 C1 AB112408 P7-2 D-L27106 D-X02496 D-M32138 E AB091256 E-X75657 E-X75664 A-L13994 A-S50225 A-M57663 G AB056515 G AF160501 G AF405706 P3-1 P3-2 B-D00331 B-D50521 B-D00330 B-D00329 B-D23678 F-X75658 F-X75663 F-X69798 H AB064315 H AB059659 H AB059660
0.06 0.05 0.04 0.03 0.02 0.01 0.00
균 7.7-11.9%의 상이율을 보였다. 같은 C형 유전자형 내에서는 C1형과 4.2%, C2형과 2.6%의 상이율을 보였다(Table 1). B형 유전자형을 가진 환자들은 A에서 H 유전자형과 평균 7.0-11.2%
의 상이율을 보였으며, 같은 B형 그룹과는 3.7%의 평균 염기서 열 상이율을 보였다.
3. 혈청형
C형 유전자형으로 동정된 419예 중adr혈청형은 385명 (91.9%), adw형은 22명(5.3%), ayr형은 2명(0.4%), 혼합형은 10명(2.3%)이었다. 10명의 혼합형은adr형과adw형의 혼합된 경우가 7명,adr형과ayr형이 혼합된 경우 2명이었고 1명에서 는 adr형과 4가지 중 어느 하나로 분류되지 않는 혈청형을 가지 고 있었다. 혈청형이 분류되지 않았던 것은 122번째 아미노산 이 aspargine (N)으로 치환된 경우이었다. B형 유전자형으로 동정된 2명은 모두adw혈청형을 보였다.
고 찰
HBV의 C형 유전자아형에 대한 그 동안의 연구보고에 따르면 C2형은 대개 임상적인 예후가 안 좋은 것과 관련되어 있다. C1 형은 HBV DNA 염기서열 1,858번째 부위에 precore stop 돌연 변이를 방지하는 것과 연관되는 cytosine이 높은 빈도로 관찰되 었고, 반대로 C2형에서는 precore stop 돌연변이가 높은 빈도 로 관찰된다고 하였다[11]. 다른 보고에서는 간 섬유화와 관련있 는 T2201C 돌연변이가 C2 유전자형에서만 검출된다고 하였다 [16]. 또한 간암의 위험이 C2형에서 C1형보다 유의하게 높다는
보고도 있었다[12]. 하지만 이와 달리 한 보고에서는 Basal core promoter (BCP) 돌연변이인 V1753과 T1762/A1764 이중 변이 가 HCC 유발과 연관되는데, 이들이 C1형에서 더 잘 일어난다고 하였다[17]. 또한 몇몇 보고들에서는 C형 유전자형의 유전자아 형의 차이가 질병 진행에 있어 임상적 영향이 없다고 하여 임상 적 중요성에 대해서는 다소 의문스러운 면이 있다고 하였다[18, 19]. 이를 통해 볼 때 한국인에서 흔히 관찰되는 HBV인 C2형은 임상적인 예후가 좋지 않으나, 본 연구결과 모두 C2형이므로 국 내에서는 이를 확인하기 위한 검사는 무의미하다고 하겠다.
본 연구에서는 유전자형의 정의가 유전자 염기서열의 차이임 을 감안하여 상이율을 구하였다. HBV 유전자형은 전체 유전자 의 8% 이상, S 유전자의 4% 이상의 차이를 보이는 경우 구분된 다[20]. 유전자아형은 전체 유전체의 4%에서 8%의 차이인 경우 구별된다고 하였으나, S 유전자에서는 확실한 기준은 없다[7].
이러한 유전자형의 구별 기준을 고려하여 분석된 염기서열의 상 이율에 대한 보고가 있었다. 전체 HBV 유전체를 대상으로 C형 유전자형인 염기서열의 상이율을 분석한 결과 C1과 C2 유전자 아형 간에는 보고에 따라 4.38-6.92%, 5.2%, 4.2% 정도의 차 이가 있다고 한다[10, 21]. 또한 전체 HBV 유전체를 대상으로 유 전자아형 내의 염기서열의 차이는 C1형은 3.1%, 1.9%, C2형은 3.9%, 2.6%의 차이를 보인다는 보고가 있었다[10, 11]. 이는 전 체 유전체를 대상으로 했을 때, 8% 이상은 유전자형에서 보이는 차이이고, 4% 이상은 유전자아형에서 보이는 차이라는 정의에 부합한다. 본 연구에서는 전체 유전체를 대상으로 분석이 이루 어지지 않아 이를 확인할 수는 없었으며, S 유전자에 국한되어서 분석을 하였다. S 유전자에 국한해서는 C형 중 C1과 C2 유전자 아형 간에 4.0%의 차이를, 유전자아형 내에는 C1형 2.9%, C2형 Genotype
(No. of sequence) A
NCBI sequences Patients
B C1 C2 D E F G H C2 B
NCBI A (3) 2.4*
sequence B (5) 6.5 3.2*
C1 (7) 8.2 7.1 2.7*
C2 (7) 8.9 7.8 4.2 1.8*
D (3) 8.3 8.9 8.6 9.3 1.9*
E (3) 7.9 7.5 8.2 8.4 6.9 1.8*
F (3) 10.9 10.9 11.5 11.5 8.9 9.6 2.9*
G (3) 6.5 7.8 8.9 9.0 7.0 7.4 10.5 0.3*
H (3) 10.6 10.3 11.3 12.0 9.8 10.4 6.5 10.1 1.0*
Patients C2 (417) 8.7 7.7 4.2 2.6 9.5 8.3 11.8 9.1 11.9 2.7*
B (2) 7.0 3.8 7.8 8.4 8.8 7.9 11.2 8.0 10.8 8.3 3.6*
Table 1. Mean percentage difference in the nucleotide sequence of the S region of HBV genome within and between 9 groups of NCBI sequences and HBV sequences from clinical samples
*, Within group difference.
2.8%의 차이를 보인 보고가 있었다[10]. 본 연구 결과에서는 C2 형 유전자아형 내의 상이율은 2.7%로 기존의 보고와 유사한 결 과이었다. 그러나, S 유전자에서 4% 이상 염기서열의 차이를 보 이는 경우 유전자형의 차이를 보인다고 하였으나, 유전자아형 간에 4.2% 정도의 차이를 보이고, 실제 유전자형 간에 7% 이상 의 차이를 보인 결과(Table 1 참조)를 감안할 때, 이에 대해서는 좀더 높은 기준(예, 5%)이 마련되어야 할 것으로 생각된다.
본 연구에서는 또한 C형 유전자형 이외에 B형 HBV가 2명 (0.5%)에서 검출되었다. 이는 이전의 국내 보고에서 조 등이 제 한효소절편길이분석법 및 염기서열 분석법을 이용한 연구에서 는 96.3% (131명/136명)에서 C형이었고 나머지 3.7% (5명/136 명)은 B형이었던 보고와 유사한 결과라 할 수 있다[22]. 그러나 본 연구에서는 유전자(아)형의 분석을 위해 S 유전자 부분만을 이용한 제한점이 있었다. 따라서 core/precore 유전자의 변이의 영향이나 Bj형 유전자형에서 보이는 B/C 유전자형 치환 또는 C/D 유전자형 치환과 같은 변이는 확인할 수 없었다[19, 23].
한국인에서 검출된 HBV의 유전자아형 분포에 대한 연구 보 고는 지금까지 없었다. NCBI 데이타베이스에 등록된 HBV 염기 서열을 토대로 한국인에서 검출된 HBV는 모두 C2형이라고 보 고한 정도가 고작이다[13, 21]. 본 연구는 한국인의 임상 검체에 서 실제 검출된 HBV를 이용하여 S 유전자를 계통발생학적으로 분석하여 C형 유전자아형의 분포를 알아보았다는 점에서 큰 의 미가 있다고 여겨진다. 그러나 만성 B형간염환자의 치료 전 HBV 염기서열을 분석에 이용한 것이 아니라 치료 후 항균제 내성검 사에 의뢰된 HBV 염기서열을 이용한 점은 본 연구의 한계라 할 수 있다. 즉, 치료로 인한 변이의 가능성을 완전히 배제할 수 없 다는 점이다. 그러나 대부분의 검체에서 C2형 유전자아형이 단 독으로 관찰된 점과 2명의 혼합주(strain)에서도 C2형 유전자아 형을 일관되게 관찰되었다는 점은 한국인 HBV 유전자아형의 대부분이 C2형임을 나타내는 것이라 할 것이다. 단지, C1형이나 C2형에 속하지 않는 바이러스주가 치료 전부터 존재했던 것인 지 아니면 치료과정 중에 발생한 변이인지는 본 연구결과만으로 정확히 알 수 없다.
결론적으로, 치료 중인 한국인 만성 B형 간염 환자의 HBV는 대부분 C2 (Ce)형임을 알 수 있었다.
요 약
배경 : 한국인 환자에서 검출되는 B형 간염 바이러스(HBV) 는 대부분 C형 유전자형이며, 이는 C1 (혹은 Cs)형과 C2 (혹은 Ce)형 유전자아형으로 구별된다. 최근 C1 및 C2 유전자아형 사
이에 간암 위험도에 차이를 보인다는 보고가 있었다. 이에 저자 들은 한국인 만성 B형 간염 환자를 대상으로 HBV 유전자아형 의 분포를 알아보고자 하였다.
방법 : 울산의대 서울아산병원에 만성 B형 간염으로 진단받 고 치료 중인 421명의 환자 검체를 대상으로 하였다. 직접염기 서열법으로 HBV S 유전자 부위를 분석한 후 계통발생학적 방 법으로 유전자아형을 결정하였다. 동일자료를 이용하여 혈청형 의 분포를 아울러 조사하였다.
결과 : 421명의 환자 검체에서 C형 유전자형은 419명(99.5%), B형은 2명(0.5%)이었다. C형 유전자형 중 단독 417명은 C2 (Ce)형 유전자아형이었고, 나머지 2명은 혼합 염기서열을 보였 다. 그러나, 혼합 염기서열 내에서도 모두 C2형의 바이러스 주 가 분명히 관찰되는 혼합 염기서열을 보였다. C형 유전자형인 419명의 혈청형은adr385명(91.9 %), adw22명(5.3%), ayr2 명(0.4%), 혼합형 10명(2.3%)이었다. B형 유전자형인 2명은 모 두adw혈청형이었다.
결론 : 치료 중인 만성B형간염 한국인 환자에서 검출되는 HBV는 거의 대부분 C2 (Ce)형임을 알 수 있었다.
참고문헌
1. Lee WM. Hepatitis B virus infection. N Engl J Med 1997;337:1733-45.
2. Joo KR, Bang SJ, Song BC, Youn KH, Joo YH, Yang S, et al. Hepati- tis B viral markers of Korean adults in the late 1990s: Survey data of 70,347 health screenees. Korean J Gastroenterol 1999;33:642-52.
(주광로, 방성조, 송병철, 윤광희, 주연호, 양수현등. 1990년대후반한국 성인의 B형 간염 바이러스표지자보유 양상: 건강검진 수검자 70,347명 의성적조사. 대한소화기학회지 1999;33:642-52.)
3. Colombo M and Lampertico P. Natural history of chronic hepatitis B and hepatocellular carcinoma. In: Thomas H, Lemon S, et al. eds.
Viral Hepatitis. 3rd ed. Oxford: Blackwell Publishing Ltd, 2005:
263-8.
4. Kao JH. Hepatitis B viral genotypes: clinical relevance and molecu- lar characteristics. J Gastroenterol Hepatol 2002;17:643-50.
5. Bartholomeusz A and Schaefer S. Hepatitis B virus genotypes: com- parison of genotyping methods. Rev Med Virol 2004;14:3-16.
6. Cha CH, Sohn YH, Jang S, Lee HJ, Lee KJ, Shin ES, et al. Genotype analysis of hepatitis B virus isolated from Korean hepatitis patients.
Korean J Lab Med 2003;23:352-6. (차충환, 손용학, 장성수, 이한주, 이 관제, 신은순, 오흥범. 국내 환자에서 분리된 B형 간염바이러스의 유전 자형분석. 대한진단검사의학회지 2003;23:352-6.)
7. Kramvis A, Kew M, Francois G. Hepatitis B virus genotypes. Vac- cine 2005;23:2409-23.
8. Bowyer SM and Sim JG. Relationships within and between geno- types of hepatitis B virus at points across the genome: footprints of recombination in certain isolates. J Gen Virol 2000;81:379-92.
9. Schaefer S. Hepatitis B virus taxonomy and hepatitis B virus geno- types. World J Gastroenterol 2007;13:14-21.
10. Huy TT, Ushijima H, Quang VX, Win KM, Luengrojanakul P, Ki- kuchi K, et al. Genotype C of hepatitis B virus can be classified into at least two subgroups. J Gen Virol 2004;85:283-92.
11. Chan HL, Tsui SK, Tse CH, Ng EY, Au TC, Yuen L, et al. Epidemi- ological and virological characteristics of 2 subgroups of hepatitis B virus genotype C. J Infect Dis 2005;191:2022-32.
12. Chan HL, Tse CH, Mo F, Koh J, Wong VW, Wong GL, et al. High viral load and hepatitis B virus subgenotype ce are associated with increased risk of hepatocellular carcinoma. J Clin Oncol 2008;26:
177-82.
13. Norder H, Courouce AM, Coursaget P, Echevarria JM, Lee SD, Mushahwar IK, et al. Genetic diversity of hepatitis B virus strains derived worldwide: genotypes, subgenotypes, and HBsAg sub- types. Intervirology 2004;47:289-309.
14. Kumar S, Nei M, Dudley J, Tamura K. MEGA: a biologist-centric software for evolutionary analysis of DNA and protein sequences.
Brief Bioinform 2008;9:299-306.
15. Kay A and Zoulim F. Hepatitis B virus genetic variability and evo- lution. Virus Res 2007;127:164-76.
16. Zhu L, Tse CH, Wong VW, Chim AM, Leung KS, Chan HL. A com- plete genomic analysis of hepatitis B virus genotypes and muta- tions in HBeAg-negative chronic hepatitis B in China. J Viral Hepat
2008;15:449-58.
17. Yuan J, Zhou B, Tanaka Y, Kurbanov F, Orito E, Gong Z, et al. Hep- atitis B virus (HBV) genotypes/subgenotypes in China: mutations in core promoter and precore/core and their clinical implications. J Clin Virol 2007;39:87-93.
18. Tseng TC, Liu CJ, Chen PJ, Lai MY, Lin CL, Kao JH, et al. Subgeno- types of hepatitis B virus genotype C do not correlate with disease progression of chronic hepatitis B in Taiwan. Liver Int 2007;27:983-8.
19. Wang Z, Hou J, Zeng G, Wen S, Tanaka Y, Cheng J, et al. Distribu- tion and characteristics of hepatitis B virus genotype C subgeno- types in China. J Viral Hepat 2007;14:426-34.
20. Norder H, Hammas B, Lofdahl S, Courouce AM, Magnius LO. Com- parison of the amino acid sequences of nine different serotypes of hepatitis B surface antigen and genomic classification of the corre- sponding hepatitis B virus strains. J Gen Virol 1992;73:1201-8.
21. Kramvis A, Arakawa K, Yu MC, Nogueira R, Stram DO, Kew MC.
Relationship of serological subtype, basic core promoter and pre- core mutations to genotypes/subgenotypes of hepatitis B virus. J Med Virol 2008;80:27-46.
22. Cho IH, Song JY, Kim DK, Lim HS, Sheen SS, Kim WS, et al. Preva- lence of HBV genotypes in Korean patients with chronic hepatitis B. Korean J Hepatol 2001;7:381-6. (조일현, 송정엽, 김덕기, 임홍석, 신 승수, 김원석등. 만성 B형 간염환자에서 B형 간염바이러스유전자형의 빈도. 대한간학회지 2001;7:381-6.)
23. Sugauchi F, Orito E, Ichida T, Kato H, Sakugawa H, Kakumu S, et al. Hepatitis B virus of genotype B with or without recombination with genotype C over the precore region plus the core gene. J Virol 2002;76:5985-92.