www.earticle.net
육우의 경제형질과 SNP간의 연관성 분석에 대한 연구현황(총설)
김훈1․박새롬1․이영섭1․이학교2․이성진1*
강원대학교 동물생명과학대학1, 한경대학교 생명공학과2
Current Status about Association of SNPs on Economic Traits in Beef Cattle (An Overview)
Hun Kim1, Sairom Park1, Young-Sub Lee1, Hak-Kyo Lee2 and Sung-Jin Lee1*
1College of Animal Life Science, Kangwon National University,
2Dept. of Biotechnology, Hankyong National University
ABSTRACT1)
The purpose of the livestock industry is to pursuit economic profits by increasing the economic productivity of livestock. Single nucleotide polymorphisms (SNPs), that is characterized by one variation out of every 1000 polymorphisms, to increase economical benefits in stock breeding has been extensively studied.
Particularly, replete number of investigations have been put forward that has associated SNPs of candidate genes with economically important traits such as carcass weight (CW), longissimus muscle area (LMA), backfat thickness (BFT) and marbling score (MS) in beef cattle. Bovine genome sequencing and HapMap projects have provided, millions of SNPs in the cattle genome. However, till date studies related to the association of the SNPs with economic traits in beef cattles are meager. Discovering of SNPs bovine whole genome would certainly be useful genetic assets for the development of SNP marker for economically important traits in beef cattle.
(Key words: SNP, Beef cattle, Economic trait, Candidate gene)
Ⅰ. 서론
축산업의 가장 중요한 목적은 축산농가의 농가 수익증대 를 위하여 품종개량, 성장촉진, 질병의 저항성 개선 등을 통 하여 농가의 가축의 경제성을 증대시키기 것을 목표로 한 다. 기존의 가축개량은 양적유전학(quantitative genetics) 이론을 바탕으로 표현형 자료(phenotypic data)와 계통 (pedigree) 정보를 이용하여 선발(selection) 및 교배(mating)
을 통해 개량하여 생산성 향상을 가져왔다. 그러나 이러한 기존의 육종 방식은 막대한 비용과 다 세대에 걸쳐 오랜 시 간이 소요되는 문제점이 지목되어져 왔다. 최근 전통적인 육종방법과 함께 기존의 육종의 문제점을 보완할 주요한 수 단으로 분자 유전학적 기법을 이용한 육종모델들이 제시되 어지고 있다.
동물의 유전체상에 약 1000개의 염기쌍 중 하나 정도의 빈도로 나타나는 염기의 변이를 단일 염기다형성, SNP
* Corresponding author: Sung-Jin Lee, Dept. of Animal Biotechnology, College of Animal Life Sciences, Kangwon National University, Chuncheon 200-701, Korea. Tel: +82-33-250-8636, E-mail: sjlee@kangwon.ac.kr
This is an Open Access journal distributed under the teams of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses(by-nc/3.0) which permits unrestricted non-commercial use, and reproduction in any medium, provided the original work is properly cited.
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
(single nucleotide polymorphisms; SNPs)라고 한다(Bell, 2002; Wang 과 Moult, 2001). 이러한 SNP는 인간 유전체의 경우 약 200~300염기마다 한 개 씩의 존재 하는 것으로 추 정되고 있다(Shin 등, 2008). 또한 최근 소의 전장유전체의 해독완료(Elsik 등, 2009) 이후에 유전체 전반에 걸쳐 확인 된 유전적 변이들 중 단일염기 다형성의 대규모 발굴(Gibbs 등, 2009; Tassell 등, 2008)과 함께 최근 차세대염기서열분 석장치(next generation sequencing; NGS) 플랫폼의 개발로 인하여 SNP 발굴이 더 가속화 되고 있다(Eck 등, 2009;
Stothard 등, 2011).
다양한 방법을 통한 SNP의 발굴과 함께 가축에서 기존 의 육종의 단점을 보안하기 위한 방법으로 SNP를 이용한 분자유전학적 검증을 통한 육종모델이 제시 되고 있다. 고 기소에서 SNP를 이용한 개량을 위해서 많은 경제형질 중 육량을 결정하는 도체중(carcass weight; CW), 등심 단면적 (longissimus muscle area; LMA), 등지방 두께(backfat thickness; BFT)와 육질에 중요한 영향을 미치는 근내 지방 도(marbling score; MS)에서 SNP와의 연관성 분석을 위한 연구가 진행되고 있다. 하지만 전장 유전체에 관한 광범위 한 SNP의 발굴 비하여 주요 경제형질간의 연관관계의 규 명이 미흡한 실정이다. 또한 SNP의 연구에 있어서 SNP들 간의 연관성 및 haplotype 등의 연구를 통하여 전장 유전체 내의 여러 SNP들과 경제형질간의 연관성 분석이 필요하다.
본 연구현황에서는 현재까지 이루어지고 있는 고기소의 SNP와 주요 경제형질간의 연관성 분석 결과를 종합하고 SNP를 통한 분자 마커 개발에 있어서의 연구 방향에 대해 논의 해보고자 한다.
Ⅱ. 본론
1. 도체중 (Carcass weight; CW)
고기소에서 도체중은 생 체중에서 두부, 내장, 족 및 가죽 등의 불가식 부분을 제외한 무게로써 실질적인 소의 가용 부분으로 고기소의 경제적 요소 중에 큰 영향을 주는 것 이 라 알려져 있다. Table 1에서는 도체중과 관련된 후보유전자 인 fatty acid synthase(FASN), Inositol 1,4,5 trisphosphate receptor type 1(IP3R1), myogenic differentiation 1 (MyoD1), fatty acid binding protein 4(FABP4), stearoyl- CoA desaturase 1(SCD1), growth-hormone-relesing hormone(GHRH), endothelial differentiation, sphingolipid
G-protein-coupled receptor 1(EDG1), melanocortin-4 receptor(MC4R), calcium channel, voltage-dependent, alpha-2/delta subunit 1(CACNA2D1), singal transducer and activator of transcription 6(STAT6)와 초위성체 좌위 (microsatellite loci, MS loci)의 ILSTS035 위치의 SNP들과 고기소의 도체중에서 연관성 연구현황을 정리하였다.
지금까지 연구된 도체중과 후보유전자간의 연관성에 사 용된 종으로는 한우, 일본흑우 등의 단일 종에서, 또 다양한 종의 시료를 모아 섞인 시료를 이용하여 도체중과 후보유 전자간의 연관성을 확인하였다. 먼저 한우에서는 FASN유 전자의 g.11280G>A(p=0.005), g.17924G>A(p=0.05, Kim 등, 2010), IP3R1유전자의 g.1428617A>G(p=0.006, Kim 등, 2009), MYOD1유전자의 g. 1274A>G(p=0.011, Bhuiyan 등, 2009), FABP4유전자의 3473T>A(p=0.01, Lee 등, 2010), SCD1유전자의 c.878T>C(p=0.32, Park 등, 2012), GHRH유 전자의 –4241A>T(p=0.005, Cheong 등, 2006)와 MS loci의 31465_446 SNP(p=0.02)와 12273_165 SNP(p=0.008, Lee 등, 2008)등에서 도체중과 연관성이 확인 되었다. 또 다른 단일 종에서 도제중과 연관성을 확인한 일본 흑우(Japanese black cattle)에서는 EDG1유전자의 c.-312A>G SNP (p=0.001)에서 유의적 차이가 보고되었다(Yamada 등, 2008).
혼합된 시료에서는 먼저 simmental, angus, hereford, charolais, limousin, qinchuan, luxi와 jinnan의 8종의 소의 시료들의 도체중과 MC4R 유전자의 G1069C SNP(p=0.047, Liu 등, 2010)와 CACNA2D1 유전자의 A526745G SNP(p=
0.044, Hou 등, 2010)에서 연관이 있는 것으로 보고되었다.
다른 혼합된 시료인 black Angus, red Angus, horned Hereford, polled Hereford, Charolais, Simmental, Limousine, Gelbvieh, Brahman, Brangus와 Wagyu의 11종 의 혼합 시료의 도체중은 STAT6유전자의 ss115492461:6>G SNP(p=0.002)에서 연관성이 보고되었다(Rincon 등, 2009).
2. 등심 단면적
등심 단면적은 소도체로부터 정육량을 예측하기 위한 목 적으로 등심단면적(배최장근단면적) 측정방법을 연구되었 다(Schoonover 과 Stratton, 1957). 이러한 등심단면적은 국 내에서 육량지수와 고급 부위육의 생산지표로 활용되고 있 다. 육량에 주요한 영향을 주는 등심단면적는 heparan sulphaye(glucosamine) 3-O-sulfotransferase 1(HS3ST1), dishevelled homolog 1(DVL1), peroxisome proliferator-
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
Table 1. The association of SNPs and CW of candidate gene in beef cattle
Gene SNP Population p-value Reference
Fatty Acid Synthase (FASN) g.11280G>A
Korean native cattle (Hanwoo)
0.005
Kim et al. (2010)
g.17924G>A 0.05
Inositol 1,4,5 trisphosphate receptor type 1
(IP3R1) g.1428617A>G 0.006 Kim et al. (2009)
Myogenic differentiation 1 (MYOD1) g.1274A>G 0.011 Bhuiyan et al. (2009)
Fatty acid binding protein 4 (FABP4) 3473T>A 0.01 Lee et al. (2010)
Stearoyl-CoA desaturase 1 (SCD1) c.878T>C 0.032 Park et al. (2012)
Growth-hormone-relesing hormone
(GHRH) -4241A>T 0.005 Cheong et al. (2006)
ILSTS035 31465_446 0.02
Lee et al. (2008)
ILSTS035 12273_165 0.008
Endothelialdifferentiation,sphingolipid
G-protein-coupled receptor 1 (EDG1) c.-312A>G Japanese Black beef cattle 0.001 Yamada et al. (2008) Melanocortin-4 receptor (MC4R) G1069C Simmental, Angus, Hereford,
Charolais, Limousin, Qinchuan, Luxi and Jinnan
0.047 Liu et al. (2010) Calcium channel, voltage-dependent,
alpha-2/delta subunit 1 (CACNA2D1) A526745G 0.044 Hou et al. (2010)
Singal transducer and activator of
transcription 6 (STAT6) ss115492461:A>G
Black Angus, Red Angus, Horned Hereford, Polled Hereford, Charolais, Simmental, Limousine, Gelbvieh, Brahman, Brangus and Wagyu
0.002 Rincon et al. (2009)
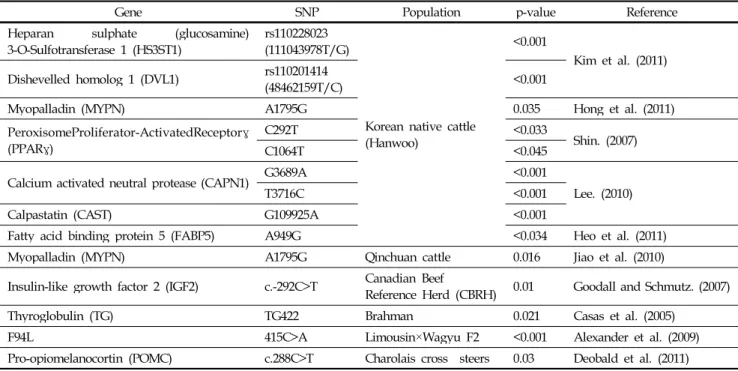
Table 2. The association of SNPs and LMA of candidate gene in beef cattle
Gene SNP Population p-value Reference
Heparan sulphate (glucosamine)
3-O-Sulfotransferase 1 (HS3ST1) rs110228023 (111043978T/G)
Korean native cattle (Hanwoo)
<0.001
Kim et al. (2011) Dishevelled homolog 1 (DVL1) rs110201414
(48462159T/C) <0.001
Myopalladin (MYPN) A1795G 0.035 Hong et al. (2011)
PeroxisomeProliferator-ActivatedReceptorγ (PPARγ)
C292T <0.033
Shin. (2007)
C1064T <0.045
Calcium activated neutral protease (CAPN1) G3689A <0.001
Lee. (2010)
T3716C <0.001
Calpastatin (CAST) G109925A <0.001
Fatty acid binding protein 5 (FABP5) A949G <0.034 Heo et al. (2011)
Myopalladin (MYPN) A1795G Qinchuan cattle 0.016 Jiao et al. (2010)
Insulin-like growth factor 2 (IGF2) c.-292C>T Canadian Beef
Reference Herd (CBRH) 0.01 Goodall and Schmutz. (2007)
Thyroglobulin (TG) TG422 Brahman 0.021 Casas et al. (2005)
F94L 415C>A Limousin×Wagyu F2 <0.001 Alexander et al. (2009)
Pro-opiomelanocortin (POMC) c.288C>T Charolais cross steers 0.03 Deobald et al. (2011)
activated receptorƔ(PPARƔ), calcium activated neutral protease(CAPN1), Calpastatin(CAST), Fatty acid binding protein 5(FABP5), Myopalladin(MYPN), Insulin-like growth factor 2(IGF2), Thyroglobulin(TG), F94L, pro-
opiomelanocortin(POMC)유전자들의 SNP와 유의성이 보 고되었다(Table 2).
종 별로 비교하였을 때 한우에서는 HS3ST1 유전자의 111043978T/G SNP(p<0.001)와 DVL1 유전자의
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
48462159T/C SNP(p<0.001, Kim 등, 2011), MYPN 유전자 의 A1795G SNP(p=0.035, Hong 등, 2011), PPARƔ 유전자 의 C292T SNP(p<0.033)와 C1064T SNP(p<0.045, Shin 등, 2007), CAPN1 유전자의 G3689A SNP(p<0.001)와 T3716C SNP(p<0.001), CAST 유전자의 G109925A SNP(p<0.001, Lee 등, 2010), FABP5 유전자의 A949G SNP(p<0.034, Heo 등, 2011)에서 유의성을 확인되었다.
다음으로 Qinchuan cattle은 MYPN 유전자의 A1795G SNP(p=0.016, Jiao 등, 2010)와 Canadian beef reference herd(CBRH)은 IGF2 유전자의 c.-292C>T SNP(p=0.01, Goodall 과 Schmutz, 2007)에서 유의적 차이가 나타났고 Brahman과 TG 유전자의 T422G SNP(p=0.021, Casas 등, 2005)에서 Limousin X Wagyu F2와 F94L 유전자의 415C>A SNP(p<0.001, Alexander 등, 2009)에서 마지막으 로 Charolais cross steer와 POMC유전자의 c.288C>T SNP (p=0.03, Deobald 등, 2011)에서 연관성이 보고되었다.
3. 등지방 두께
등지방 두께는 도체의 품질과 고기양을 평가하는데 유용 한 특성으로 등쪽의 지방의 두께로 이에 따른 등급요인 및 가격 지표에 요인이 된다. 이러한 상업적 가치 때문에 등지 방과 주요 후보 유전자의 SNP와의 연관성 분석이 실시되 고 있는 추세이다. Table 3에서는 국내외에서 보고된 등지 방과 주요 후보 유전자의 SNP간의 연관성 분석 연구 현황 을 표로 나타냈다.
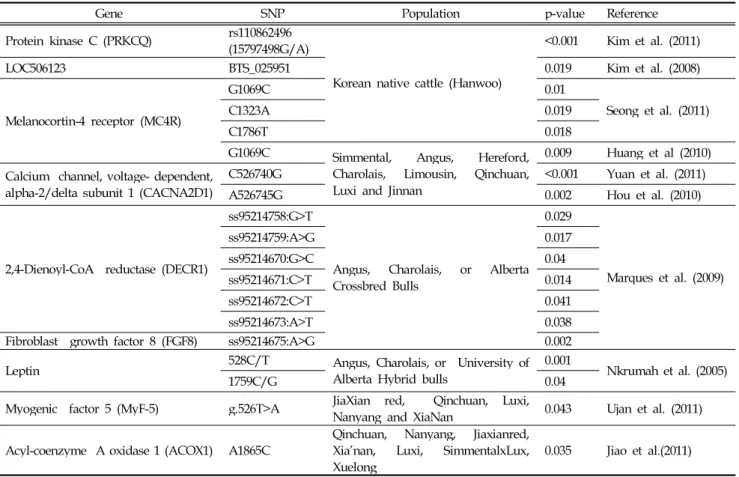
최근까지 등지방 두께와 연관성이 보고된 후보유전자의 SNP은 protein kinase C(PRKCQ)유전자의 15797498G/A SNP(p<0.001, Kim 등, 2011), MC4R유전자의 G1069C SNP(p=0.01), C1323A SNP(p=0.019), C1789T SNP(p=0.018, Seong 등, 2011) 과 MS loci인 LOC506123의 BTS_025951 SNP(p=0.019, Kim 등, 2008)에서 한우의 등지방 두께와 연 관성이 확인 되었다.
그 외 다른 종의 혼합된 시료들 중 먼저 simmental, angus, hereford, charolais, limousin, qinchuan, luxi와 jinnan의 8종의 혼합 시료와 MC4R의 G1069C SNP (p=0.009, Huang 등, 2010), calcium channel voltage- dependent, alpha-2/delta subunit 1(CACNA2D1)유전자의
Table 3. The association of SNPs and BFT of candidate gene in beef cattle
Gene SNP Population p-value Reference
Protein kinase C (PRKCQ) rs110862496 (15797498G/A)
Korean native cattle (Hanwoo)
<0.001 Kim et al. (2011)
LOC506123 BTS_025951 0.019 Kim et al. (2008)
Melanocortin-4 receptor (MC4R)
G1069C 0.01
Seong et al. (2011)
C1323A 0.019
C1786T 0.018
G1069C Simmental, Angus, Hereford, Charolais, Limousin, Qinchuan, Luxi and Jinnan
0.009 Huang et al (2010) Calcium channel, voltage- dependent,
alpha-2/delta subunit 1 (CACNA2D1)
C526740G <0.001 Yuan et al. (2011)
A526745G 0.002 Hou et al. (2010)
2,4-Dienoyl-CoA reductase (DECR1)
ss95214758:G>T
Angus, Charolais, or Alberta Crossbred Bulls
0.029
Marques et al. (2009)
ss95214759:A>G 0.017
ss95214670:G>C 0.04
ss95214671:C>T 0.014
ss95214672:C>T 0.041
ss95214673:A>T 0.038
Fibroblast growth factor 8 (FGF8) ss95214675:A>G 0.002
Leptin 528C/T Angus, Charolais, or University of
Alberta Hybrid bulls
0.001
Nkrumah et al. (2005)
1759C/G 0.04
Myogenic factor 5 (MyF-5) g.526T>A JiaXian red, Qinchuan, Luxi,
Nanyang and XiaNan 0.043 Ujan et al. (2011) Acyl-coenzyme A oxidase 1 (ACOX1) A1865C Qinchuan, Nanyang, Jiaxianred,
Xia’nan, Luxi, SimmentalxLux,
Xuelong 0.035 Jiao et al.(2011)
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
C526740G(p<0.001, Yuan 등, 2011)와 A526745G의 SNP(p=0.002, Hou 등, 2010)에서 연관성이 나타났다. 다음 으로 angus, charolais or alberta crossbreed bulls에서 2,4-dienoyl-CoA reductase(DECR1)의 6개의 SNPss95214758:G>T(p=0.029), ss95214759:A>G(p=0.017), ss95214670: G>C (p=0.04), ss95214671:C>T(p=0.014), ss95214672:C>T (p=0.041), ss95214673:A>T(p=0.038)과 Fibroblast growth factor 8(FGF8)유전자의 ss95214675:A>G SNP(p=0.002)에서 유의적인 차이가 보고되었다(Marques 등, 2009).
Leptin 유전자의 528C/T SNP(p=0.001), 1759C/G SNP (p=0.04)는 Angus, Charolais, or University of Alberta hybrid bulls에서 유의성을 확인하였다(Nkrumah 등, 2005).
Myogenic factor(MyF-5) 유전자의 g.526T>A SNP (p=0.043, Ujan 등, 2011)와 ja xian red, qinchuan, luxi, nanyang, xianan의 혼합된 소의 등지방 두께와 관련이 있 음이 보고 되었고 qinchuan, nanyang, jiaxian red, xia’nan, luxi, simmental, luxi, xuelon의 혼합된 시료에서 acyl-coenzyme A oxidase 1(ACOX1) 유전자의 A1895C SNP(p=0.035, Jiao 등 2011)에서 연관성이 확인되었다.
4. 근내 지방도
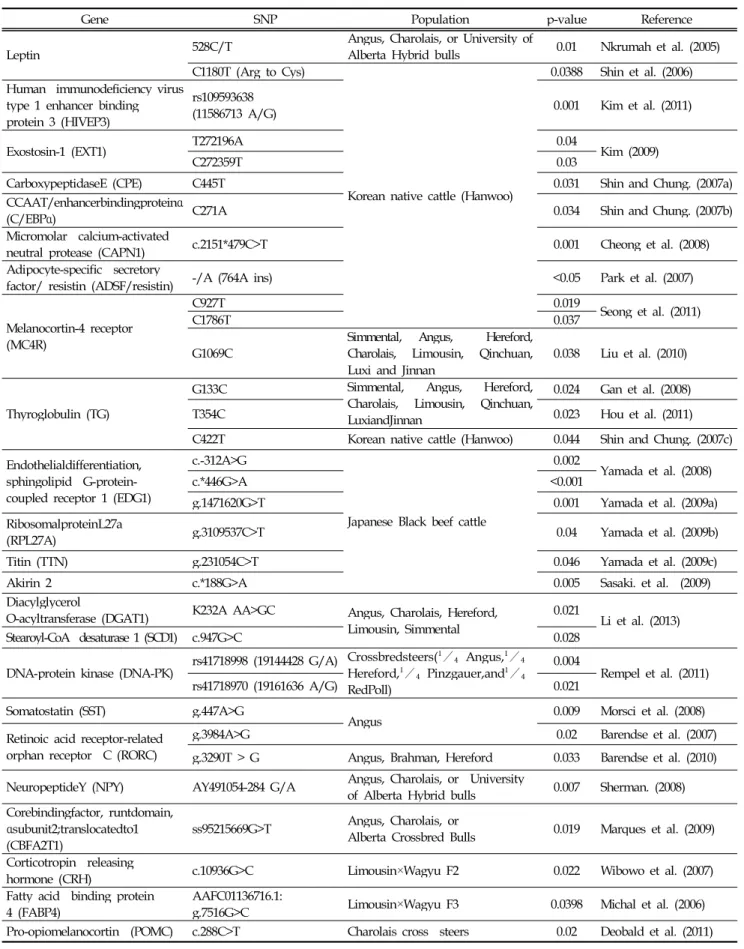
육우에 있어서 고기의 육질을 영향을 주는 요인으로 육 색, 지방색, 연도 및 근내 지방도 등이 있으며, 그 중 근내 지방도는 고기의 연도와 풍미를 증가시키는 요소이다. 이러 한 근내 지방도는 쇠고기를 구워 먹는 식문화를 가진 한국, 일본 등 국가에서 쇠고기의 경제적 가치를 결정하는 주요 한 요소이다. 이러한 근내 지방도와 후보유전자의 SNP간의 연관성 분석을 통하여 고급육 생산을 위한 연구가 활발히 이뤄지고 있다. 근내 지방도와 연관된 것으로 보고된 후보 유전자에는 leptin, human immunodeficiency virus type 1 enhancer binding protein 3(HIVEP3), exostosin-1(EXT1), carboxypeptidase E(CPE), CCAAT/enhancer binding protein α(C/EBPα), micromoiar calcium-activated neutral protease(CAPN1), adipocyte-specific secretory factor/resistin ADSF/resistin), MC4R, TG, EDG1, ribosomal protein L27a(RPL27A), akirin2, titin(TTN), diacylglycerol O-acyltransferase 1(DGAT1), SCD1, DNA-protein kinase(DNA-PK), somatostatin(SST), retinoic acid receptor-receptor-related orphan receptor C(RORC), neuropeptide Y(NPY), corebinding factor, runtdomain, α subunit 2; translocate dto 1(CBFA2T1), corticotropin
releasing hormone(CRH), fatty acid binding protein 4(FABP4), pro-opiomelanocortin(POMC) 등의 후보유전자 의 SNP들에서 근내 지방도와 연관성이 나타났다(Table 4).
특히 그 중 한우의 근내 지방도와 CAPN1유전자의 c.2151*
479C>T SNP(p=0.001, Cheong 등, 2008)에서 그리고 일본 흑우의 근내 지방도와 EDG1유전자의 c.*446G>A SNP (p<0.001, Yamada 등, 2008)와 g.1471620G>T SNP(p=0.001, Yamada 등, 2009)에서 상당히 유의적인 차이가 나타났다.
Ⅲ. 결론
고기소의 개량에 있어 전통적인 방법은 당대 및 후대 검 정을 위한 오랜 시간과 개량의 과학적 검증이 힘들며, 고비 용으로 인하여 점차 분자유전학적인 기법을 이용한 새로운 육종 모델을 선발하기 위한 연구가 진행되고 있는 추세이 다. 최근 소의 전장 유전체의 해독 이후 전장 유전체에 걸 친 단일염기다형성인 SNP의 발굴이 활발히 이루어지면서 새로운 육종 모델 중의 하나로 SNP를 이용한 조기선발 마 커 개발을 위한 연구가 이루어지고 있다. 특히 농가 소득에 주요한 영향을 주는 경제형질인 도체중, 등심단면적, 등지 방두께, 근내 지방도등과 후보유전자의 SNP간 연관성을 확 인하기 위한 연구가 진행되고 있다.
지금까지 보고된 후보유자의 SNP와 경제형질간에서 연관 성이 보고된 바에 의하면 도체중에서는 FASN, IP3R1, MyoD1, FABP4, SCD1, GHRH, EDG1, MC4R, CACNA2D1, STAT6 유전자와 초위성체 좌위 ILSTS035 위 치 등의 SNP들에서 연관성을 확인되었고 HS3ST1, DVL1, PPARƔ, CAPN1, CAST, FABP5, MYPN, IGF2, TG, F94L, POMC 등의 유전자들의 SNP와 등심단면적에 연관성에 대 한 연구가 진행된 바 있다. 또 등지방 두께와 PRKCQ, MC4R, CACNA2D1, DECR1, FGF8, Leptin, MyF-5, ACOX1 유전자들의 SNP와 연관성 보고되었고 육질에 중 요한 역할을 하는 근내 지방도와 Leptin, HIVEP3, EXT1, CPE, C/EBPα, CAPN1, ADSF/resistin, MC4R, TG, EDG1, RPL27A, akirin2, TTN, DGAT1, SCD1, DNA-PK, SST, RORC, NPY, CBFA2T1, CRH, FABP4, POMC 등의 유전자 의 SNP들에서 연관성이 확인 되었다.
이러한 많은 후보유전자의 SNP과 경제형질간의 연관성이 계속 보고되고 있으며 최근 차세대염기서열분석장치 플랫 폼을 이용한 대규모의 SNP 발굴과 함께 각 SNP와 경제형 질간의 연관성 분석뿐만 아니라 SNP와 SNP간의 연관성
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
Table 4. The association of SNPs and MS of candidate gene in beef cattle
Gene SNP Population p-value Reference
Leptin 528C/T Angus, Charolais, or University of
Alberta Hybrid bulls 0.01 Nkrumah et al. (2005) C1180T (Arg to Cys)
Korean native cattle (Hanwoo)
0.0388 Shin et al. (2006) Human immunodeficiency virus
type 1 enhancer binding protein 3 (HIVEP3)
rs109593638
(11586713 A/G) 0.001 Kim et al. (2011)
Exostosin-1 (EXT1) T272196A 0.04
Kim (2009)
C272359T 0.03
CarboxypeptidaseE (CPE) C445T 0.031 Shin and Chung. (2007a)
CCAAT/enhancerbindingproteinα
(C/EBPα) C271A 0.034 Shin and Chung. (2007b)
Micromolar calcium-activated
neutral protease (CAPN1) c.2151*479C>T 0.001 Cheong et al. (2008)
Adipocyte-specific secretory
factor/ resistin (ADSF/resistin) -/A (764A ins) <0.05 Park et al. (2007)
Melanocortin-4 receptor (MC4R)
C927T 0.019 Seong et al. (2011)
C1786T 0.037
G1069C Simmental, Angus, Hereford,
Charolais, Limousin, Qinchuan,
Luxi and Jinnan 0.038 Liu et al. (2010)
Thyroglobulin (TG)
G133C Simmental, Angus, Hereford,
Charolais, Limousin, Qinchuan, LuxiandJinnan
0.024 Gan et al. (2008)
T354C 0.023 Hou et al. (2011)
C422T Korean native cattle (Hanwoo) 0.044 Shin and Chung. (2007c) Endothelialdifferentiation,
sphingolipid G-protein- coupled receptor 1 (EDG1)
c.-312A>G
Japanese Black beef cattle
0.002
Yamada et al. (2008)
c.*446G>A <0.001
g.1471620G>T 0.001 Yamada et al. (2009a)
RibosomalproteinL27a
(RPL27A) g.3109537C>T 0.04 Yamada et al. (2009b)
Titin (TTN) g.231054C>T 0.046 Yamada et al. (2009c)
Akirin 2 c.*188G>A 0.005 Sasaki. et al. (2009)
Diacylglycerol
O-acyltransferase (DGAT1) K232A AA>GC Angus, Charolais, Hereford, Limousin, Simmental
0.021
Li et al. (2013)
Stearoyl-CoA desaturase 1 (SCD1) c.947G>C 0.028
DNA-protein kinase (DNA-PK) rs41718998 (19144428 G/A) Crossbredsteers(¹⁄₄Angus,¹⁄₄ Hereford,¹⁄₄Pinzgauer,and¹⁄₄ RedPoll)
0.004
Rempel et al. (2011)
rs41718970 (19161636 A/G) 0.021
Somatostatin (SST) g.447A>G
Angus 0.009 Morsci et al. (2008)
Retinoic acid receptor-related orphan receptor C (RORC)
g.3984A>G 0.02 Barendse et al. (2007)
g.3290T > G Angus, Brahman, Hereford 0.033 Barendse et al. (2010) NeuropeptideY (NPY) AY491054-284 G/A Angus, Charolais, or University
of Alberta Hybrid bulls 0.007 Sherman. (2008) Corebindingfactor, runtdomain,
αsubunit2;translocatedto1
(CBFA2T1) ss95215669G>T Angus, Charolais, or
Alberta Crossbred Bulls 0.019 Marques et al. (2009) Corticotropin releasing
hormone (CRH) c.10936G>C Limousin×Wagyu F2 0.022 Wibowo et al. (2007)
Fatty acid binding protein
4 (FABP4) AAFC01136716.1:
g.7516G>C Limousin×Wagyu F3 0.0398 Michal et al. (2006) Pro-opiomelanocortin (POMC) c.288C>T Charolais cross steers 0.02 Deobald et al. (2011)
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
분석, 후보유전자 내에 존재하는 다수의 SNP와 경제형질간 의 연구가 필요할 것으로 사료되며 이를 통한 높은 신뢰성 과 경제성을 갖춘 분자유전학적 육종마커 개발 및 육종 모 델 제시를 위한 연구가 계속 필요할 것으로 보인다.
Ⅳ. 요약
축산업의 가장 큰 목적은 가축의 경제적 생산성의 증진을 통한 경제적 이익을 추구하기 위해서 품종개량, 질병의 개 선 및 치료, 성장 촉진을 목표로 한다. 전통적인 육종에서는 후대검정을 통한 검정소 검정(station test)방법을 이용하였 으나 이러한 방법은 선발의 강도 및 선발의 정확도를 크게 하는데 한계가 있고 고비용과 오랜 시간이 요구되어진다. 최근 분자 유전학의 발전과 함께 경제적 생산성 증가를 도 모하기 위하여 동물의 유전체의 약 1,000개의 염기쌍 마다 염기의 변이가 일어나는 단일염기다형성인 SNP을 이용하 여 경제형질과의 연관성을 알아보기 위한 연구가 이루어지 고 있다. 특히 고기소에서는 고기의 양을 결정하는 도체중, 등심단면적, 등지방 두께와 고기의 육질에 주요한 영향을 주는 것으로 알려진 마블링에 관하여 조기 선발 및 육종을 위한 다양한 후보유전자의 SNP와 연관성에 관한 연구가 보고되고 있다. 2009년 국제 소유전체 및 반수체 프로젝트 의 발표 후, 전장 유전체에 관한 광범위한 SNP가 널리 발 굴 되었으나 아직 그 SNP와 주요 경제형질간의 연관관계 의 규명에 있어서 미흡한 실정이다. 또한 SNP간의 연관성 및 haplotype등의 연구를 통하여 전장 유전체 내의 여러 SNP들과 경제형질간의 연관성을 확인하여 산업적 활용가 치가 높은 분자 표지의 개발이 필요하다.
사사
본 연구는 농촌 진흥청 차세대 바이오그린21 사업(과제번 호 PJ008196, PJ008028)으로 이루어 졌으며 이에 감사드립 니다.
Ⅴ. References
1. Alexander, L. J., Kuehn, L. A., Smith, T. P. L.,
Matukumalli, L. K., Mote, B., Koltes, J. E., Reecy, J., Geary, T. W., Rule, D. C. and MacNeil, M. D. 2009. A Limousin specific myostatin allele affects longissimus muscle area and fatty adci profiles in a Wagyu-Limousin F 2 population. J. Anim. Sci.
87:1576-1581.
2. Barendse, W., Bunch, R. J., Kijas, J. W. and Thomas, M. B. 2007. The Effect of genetic variation of the retinoic acid receptor-related orphan receptor C gene on fatness in cattle. Genet. 175:843-853.
3. Barendse, W., Bunch, R. J. and Harrison, B. E. 2010.
The effect of variation at the retinoic acid receptor-related orphan receptor C gene on intramuscular fat percent and marbling score in Australian cattle. J. Anim. Sci. 88(1):47-51.
4. Bell, J. L. 2002. Single nucleotide polymorphism and disease gene mapping. Arthritis Research, Vol. 4, pp.
S273-S278.
5. Bhuiyan, M. S. A., Kim, N. K., Cho, Y. M., Yoon, D., Kim, K. S., Jeon. J. T. 2009. Identification of SNPs in MYOD gene family and their associations with carcass traits in cattle. Livest. Sci. 126(1):292-297.
6. Casas, E., White, S. N., Riley, D. G., Smith, T. P. L., Brenneman, R. A., Olson, T. A., Johnson, D. D., Coleman, S. W., Bennett, G. L. and Chase, C. C. Jr.
2005. Assessment of single nucleotide polymorphisms in genes residing on chromosomes 14 and 29 for association with carcass composition traits in Bos indicus cattle. J. Anim. Sci. 83:13-19.
7. Cheong, H. S., Yoon, D. H., Park, B. L., Kim, L. H., Bae, J. S., Namgoong, S., Lee, H. W., Han, C. S., Kim, J. Q., Cheong, I. C. and Shin, H. D. 2008. A single nucleotide polymorphism in CAPN1 associated with marbling score in Korean cattle. BMC Genet. 19;9:33.
8. Deobald, H. M. and Buchanan, F. C. 2011.
Characterization of two pro-opiomelanocortin gene variants and their effects on carcass traits in beef cattle. BMC Genet. 12:2.
9. Eck, S. H., Benet-Pagès, A., Flisikowski, K., Meitinger, T., Fries, R., Strom, T. M. 2009. Whole genome sequencing of a single Bos taurus animal for single nucleotide polymorphism discovery. Geno. Biol.
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
10:R82.
10. Elsik, C. G., Tellam, R. L., Worley, K. C., Gibbs, R. A., Choi, J. et al. 2009. The genome sequence of taurine cattle: a window to ruminant biology and evolution.
Sci. 324:522-528.
11. Gan, Q. F., Zhang, L. P., Li, J. Y., Hou, G. Y., Li, H.
D., Gao, X., Ren, H. Y., Chen, J. B. and Xu, S. Z.
2008. Association analysis of thyroglobulin gene variants with carcass and meat quality traits in beef cattle. J. Appl. Genet. 49(3):251-5.
12. Gibbs, R. A., Taylor, J. F., Van Tassell, C. P., Barendse, W., Choi, J. et al. 2009. Genome-wide survey of SNP variation uncovers the genetic structure of cattle breeds. Sci. 324:528-532.
13. Goodall, J. J. and Schmutz, S. M. 2007. IGF2 gene characterization and association with rid eye area in beef cattle. Ani. Genet. 38:154-161.
14. Heo, K. N., Kim, N. K., Lee, S. H., Kim, N. Y., Jeon, J. T., Park, E. W., Oh, S. J., Kim, T. H., Seong, H. H.
and Yoon, D. H. 2011. Association between the polymorphism of the fatty acid binding protein 5 (FABP5) Gene within the BTA 14 QTL region and carcass/meat quality traits in Hanwoo. J. Anim. Sci.
Technol. 53(4):311-317.
15. Hong, M. W., Kim, H., Park, S. R., Lee, S. K., Lee, Y.
S., Kim, J. W., Kim, K. B., Lee, S. Y., Song, Y. H. and Lee, S. J. 2011. Association analysis between A1795G single nucleotide polymorphism (SNP) in Myopalladin (MYPN) gene and carcass traits in Korean cattle. Ann.
Anim. Resour. Sci. 22(2):77-83.
16. Hou, G. Y., Yuan, Z. R., Gao, X., Li, J. Y., Gao, H. J., Chen, J. B. and Xu, S. Z. 2010. Genetic polymorphisms of the CACNA2D1 gene and their association with carcass and meat quality traits in cattle. Bioch. Genet. 48:751-759.
17. Huang, M., Gao, X., Li, J. Y., Ren, H. Y., Chen, J. B.
and Xu, S. Z. 2010. Polymorphisms in MC4R gene and correlations with economic traits in cattle. Mol.
Biol. Rep. 37:3941-3944.
18. Jiao, Y., Zan, L. S., Liu, Y. F., Wang, H. B. and Guo, B. L. 2010. A novel polymorphism of the MYPN gene and its association with meat quality traits in Bos
taurus. Genet. & Mol. Res. 9(3):1751-1758.
19. Jiao, Y., Zan, L. S., Liu, Y. F. and Wang, H. B. 2011.
Molecular characterization, polymorphism of the ACOX1 gene and association with ultrasound traits in Bos taurus. Genet. Mol. Res. 10(3):1948-1957.
20. Kim, B. S. 2009. Association between the polymorphism of the nephroblastoma overexpressd and exostosin-1 genes within BTA 14 QTL region and economic traits in Hanwoo. M.Sc. Dissertation Kangwon National University, Chuncheon, Korea 21. Kim, D. H., Lee, Y. S., Oh, Y. S., Choi, C. B. and Yeo,
J. S. 2008. Discovery of candidate SNP related to meat quality using the BcSNPdb and Cattle QTLdb in Hanwoo J. Anim. Sci. Technol. 50(6):775-782.
22. Kim, N. K., Kim, G. S., Jung, Y. S., Moon, H. J., Cho, Y. M. and Yoon, D. H. 2009. Association study between polymorphisms of Inositol 1,4,5-triphosphate Receptor Type 1 (IP3R1) gene and carcass traits in Korean Cattle (Hanwoo). J. Anim. Sci. Technol.
51(4):289-294.
23. Kim, S. W., Lee, J. H., Kim, J. H., Won, Y. S., Kim, N.
S. and Kim, K. S. 2010. Effect of the fatty acid synthase gene for beef quantity traits in Hanwoo Breeding Stock. J. Anim. Sci. & Technol. 52(1):9-16.
24. Kim, Y., Ryu, J., Kim, J. B., Kim, C. Y. and Lee, C.
2011. Genome-wide association study reveals five nucleotide sequence variants for carcass traits in beef cattle. Ani. Genet. 32:361-365.
25. Lee, J. A. 2010. Identification of a SNP in Hanwoo CAPN1 and CAST gene with its effect on economic traits. M.Sc. Thesis. Hankyong National University, Anseong, Korea.
26. Lee, J. Y. and Kim, D. C. 2008. Main SNP Identification of Hanwoo carcass weight with multifactor dimensionality reduction (MDR) method.
Kor. J. Appl. Statis. 21(1):53-63.
27. Lee, S. H., van der Werf, J. H. J., Lee, S. H., Park, E.
W., Oh, S. J., Gibson, J. P. and Thompson, J. M.
2010. Genetic polymorphisms of the bovine Fatty acid binding protein 4 gene are significantly associated with marbling and carcass weight in Hanwoo (Korean Cattle). Anim. Genet. 41:442-444.
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
28. Liu, H., Tian, W., Zan, L., Wang, H. and Cui, H.
2010. Mutations of MC4R gene and its association with economic traits in Qinchuan cattle. Mol. Bio.
Rep. 37(1):535-40.
29. Li, X., Ekerljung, M., Lundstrom, K. and Lunden, A.
2013. Association of polymorphisms at DGAT1, leptin, SCD1, CAPN1 and CAST genes with color, marbling and water holding capacity in meat from beef cattle populations in Sweden. Meat Sci. 94:
153-158.
30. Marques, E., Nkrumah, J. D., Sherman, E. L. and Moore, S. S. 2009. Polymorphisms in positional candidate genes on BTA14 and BTA26 affect carcass quality in beef cattle. J. Anim. Sci. 87(8):2475-2484.
31. Michal, J. J., Zhang, Z. W., Gaskins, C. T. and Jiang, Z. 2006. The bovine fatty acid binding protein 4 gene is significantly associated with marbling and subcutaneous fat depth in Wagyu x Limousin F2 crosses. Anim. Genet. 37:400-402.
32. Morsci, N. S., Schnabel, R. D. and Taylor, J. F. 2008.
Association analysis of adiponectin and somatostatin polymorphisms on BTA1 with growth and carcass traits in Angus cattle. Anim. Genet. 37(6):554-62.
33. Nkrumah, J. D., Li, C., Yu, J., Hansen, C., Keisler, D.
H. and Moore, S. S. 2005. Polymorphisms in the bovine leptin promoter associated with serum leptin concentration, growth, feed intake, feeding behavior, and measures of carcass merit. J. Anim. Sci.
83(1):20-28.
34. Park, J. A., Kang, H. K., Chae, E. J., Seo, K. S., Kim, S. H., Yun, C. H. and Moon, Y. S. 2007. Analysis of the ADSF/resistin gene polymorphism associated with carcass traits in Hanwoo J. Anim. Sci. Technol.
49(5):577-584.
35. Park, S. R., Hong, M. W., Kim, H., Lee, S. K., Lee, Y.
S., Kim, J. W., Song, Y. H., Kim, K. B., Oh, J. D., Lee, H. K., Choi, J. W. and Lee, S. J. 2012. Association between a SNP of Stearoyl-CoA Desaturase-1 (SCD1) gene and economic traits using PCR-RFLP in Hanwoo. Ann. Anim. Resour. Sci. 23(1):1-7.
36. Rempel, L. A., Casas, E., Shackelford, S. D. and Wheeler, T. L. 2011. Relationship of polymorphisms
within metabolic genes and carcass traits in crossbred beef cattle. J. Anim. Sci. 90:1311-1316.
37. Rincon, G., Farber, E. A., Farber, C. R., Nkrumah, J.
D. and Medrano, J. F. 2009. Polymorphisms in the STAT6 gene and their association with carcass traits in feedlot cattle. Anim. Genet. 40(6):878-882.
38. Sasaki, S., Yamada, T., Sukegawa, S., Miyake, T., Fujita, T., Morita, M., Ohta, T., Takahagi, T., Murakami, H., Morimatsu F. and Sasaki, Y. 2009.
Association of a single nucleotide polymorphism in akirin 2 gene with marbling in Japanese Black beef cattle. BMC. Res. Notes. 2:131.
39. Schoonover, C. O. and Stratton, P. O. 1957 A photographic grid used to measure rib eye areas. J.
Anim. Sci. 16:957-960.
40. Seong, J. Y., Suh, D. S., Park, K. D., Lee, H. K., Kong, H. S. 2011. Identification and analysis of MC4R polymorphisms and their association with economic traits of Korean cattle (Hanwoo). Mol. Biol. Rep.
39:3597-3601.
41. Sherman, E. L., Nkrumah, J. D., Murdoch, B. M., Li, C. Wang, Z. Fu, A. and Moore, S. S. 2008.
Polymorphisms and haplotypes in the bovine neuropeptide Y, growth hormone receptor, ghrelin, insulin-like growth factor 2, and uncoupling proteins 2 and 3 genes and their associations with measures of growth, performance, feed efficiency, and carcass merit in beef cattle. J. Anim. Sci. 86(1):1-16.
42. Shin S. C. 2006. The development of DNA markers related to carcass and meat quality traits in kroean cattle. M.Sc. Thesis. Sangji University, Wonju, Korea.
43. Shin, S. C. and Chung, E. R. 2007. Association of SNP marker in the leptin gene with carcass and meat quality traits in Korean cattle. Asian-Aust J. Anim.
Sci. 20(1):1-6.
44. Shin, S. C. and Chung, E. R. 2007a. SNP detection of carboxypeptidase E gene and its association with meat quality and carcass traits in Korean cattle.
Asian-Aust. J. Anim. Sci. 20:328-333.
45. Shin, S. C. and Chung, E. R. 2007b. Identification of a novel SNP associated with meat qualtiy in C/EBPα gene of Korean cattle. Asian-Aust. J. Anim. Sci.
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM
www.earticle.net
20:466-470.
46. Shin, S. C. and Chung, E. R. 2007c. Association of SNP marker in the thyroglobulin gene with carcass and meat quality traits in Karean cattle. Asian-Aust.
J. Anim. Sci. 20:172-177.
47. Shin, H. S., Hong, M. W., Yun, D. W., Won, J. S., Kim, H., Song, Y. H. and Lee, S. J. 2008. Current status of SNP research in Korean cattle. Ann. Anim.
Resour. Sci. 19:1-10.
48. Stothard, P., Choi, J. W., Basu, U., Sumner-Thomson, J. M., Meng, Y., Liao, X. and Moore, S. S. 2011. Whole genome resequencing of black Angus and Holstein cattle for SNP and CNV discovery. BMC Geno.
12:559.
49. Ujan, J. A., Zan, L. S., Ujan, S. A., Adoligbe, C. and Wang, H. B. 2011. Back fat thickness and meat tenderness are associated with a 526 T->A mutation in the exon 1 promoter region of the MyF-5 gene in Chinese Bos taurus. Genet. Mole. Res. 10(7):3070-3079.
50. Van Tassell, C. P., Smith, T. P. L., Matukumalli, L. K., Taylor, J. F., Schnabel, R. D., Lawley, C. T., Haudenschild, C. D., Moore, S. S., Warren, W. C. and Sonstegard, T. S. 2008. SNP discovery and allele frequency estimation by deep sequencing of reducedrepresentation libraries. Nat. Methods.
5:247-252.
51. Wang, Z. and Moult, J. 2001. SNPs, Protein structure, and disease, Hum. Mutat. 17:263-270.
52. Wibowo, T. A., Michal, J. J. and Jiang, Z. 2007.
Corticotropin releasing hormone is a promising candidate gene for marbling and subcutaneous fat depth in beef cattle. Geno. 50(10):939-45.
53. Yamada, T., Itoh, M., Nishimura, S., Taniguchi, Y., Miyake, T., Sasaki, S., Yoshioka, S., Fujita, T., Shiga, K., Morita, M. and Sasaki, Y. 2008. Association of single nucleotide polymorphisms in the endothelial differentiation sphingolipidG-protein-coupled receptor 1 gene with marbling in Japanese black beef cattle.
Anim. Genet. 40:209-216.
54. Yamada, T., Sasaki, S., Sukegawa, S., Miyake, T., Fujita, T., Kose, H., Morita, M., Takahagi, Y., Murakami, H., Morimatsu, F. and Sasaki, Y. 2009a.
Novel SNP in 5' flanking region of EDG1 associated with marbling in Japanese Black beef cattle. J. Anim.
Sci. 80(4):486-9.
55. Yamada, T., Sasaki, S., Sukegawa, S., Miyake, T., Fujit, T., Kose, H., Morita, M., Takahagi, Y., Murakami, H., Morimatsu, F. and Sasaki, Y. 2009b. Association of a single nucleotide polymorphism in ribosomal protein L27a gene with marbling in Japanese Black beef cattle. J. Anim. Sci. 80(6):631-5.
56. Yamada, T., Sasaki, S., Sukegawa, S., Yoshioka, S., Takahagi, Y., Morita, M., Murakami, H., Morimatsu, F., Fujita, T., Miyake, T. and Sasaki, Y. 2009c.
Association of a single nucleotide polymorphism in titin gene with marbling in Japanese Black beef cattle.
BMC. Res. Notes. 2:78. online. Available:http:
//www.biomedcentral.com/1756-0500/2/78.
57. Yuan, Z. R. and Xu, S. Z. 2011. Novel SNPs of the bovine CACNA2D1 gene and their association with carcass and meat quality traits. Mol. Biol. Rep.
38:365-370.
(투고일: 2013.05.10. 수정일: 2013.06.13. 판정일: 2013.06.14.)
[Provider:earticle] Download by IP 118.70.52.165 at Monday, December 20, 2021 8:02 PM