Investigation of Microbial Communities in Sulculus diversicolor supertexta Through 16S rRNA Sequencing and Antibacterial Monitoring of Harmful Strains
Min-Seon Kim
1, Seung-Jong Lee
1* and Moon-Soo Heo
21
Jeju Fisheries Research Institute, National Institute of Fisheries Science, Jeju 63068, Korea
2
Major of Aquatic Life Medicine, Faculty of Marine Biomedical Sciences, Jeju National University, Jeju 63243, Korea Received August 7, 2018 /Revised October 12, 2018 /Accepted October 22, 2018
This study investigated the muscles, intestines, and gonads of Sulculus diversicolor supertexta to exam- ine the diversity of microbial communities within examples collected from the Jeju Coast. Using differ- ent media, initial pure isolation in MA, 1% BHIA, and 1% TSA indicated that the muscles, intestines, and gonads supported more communities, respectively. In analysis of relative similarity with 16s rRNA sequencing, 190 pure colonies were isolated, and further analysis with NBLAST identified 71 species, 39 genera, 25 families, and five phyla. Homogeny with the reference strain was 91-100%.
Microbial communities in S. supertexta consisted of gamma and alpha Proteobacteria (48%), Actinobac- teria (32.5%), Firmicutes (16.9%), Deinococcus-Thermus (1.3%), and Bacteroides (1.3%). In all tissue, Psychrobacter cibarius in Moraxellaceae was dominant. Alteromonadaceae, Enterobacteriaceae, Pasturellaceae, Moraxellaceae, Rhodobacteraceae, Geminicoccaceae, Dietziaceae, Intrasporangiaceae, Microbacteriaceae, Micrococ- caceae, Micromonosporaceae, Streptomycetaceae, Aerococcaceae, Bacillaceae, Paenibacillaceae, Planococcaceae, and Staphylcoccaceae were commonly isolated across all tissues, and Flavobacteriaceae, Corynebacteriaceae, Yesiniaceae, Vibrionaceae, Hahellaceae, Pseudomonadaceae were also identified from the intestines. In mi- crobial monitoring of four harmful bacteria, Streptomyces albus (96%) showed antibacterial activity against all four strains, and Agrococcus baldri (99%) and Psychrobacter nivimaris (99%) presented against E. Coli and E. aerogens. In addition, some strains with low homogeny were isolated and further experi- ments are therefore required, for example to refine the antimicrobial substances including new strain investigations. These additional experiments would aim to establish generic resources for the micro- bial communities in S. Supertexta and provide basic data for applied microbiological research.
Key words : Marine bacteria, microbial communities, Sulculus diversicolor supertexta
*Corresponding author
*Tel : +82-64-750-4370, Fax : +82-64-743-5884
*E-mail : sjlee1225@korea.kr
This is an Open-Access article distributed under the terms of the Creative Commons Attribution Non-Commercial License (http://creativecommons.org/licenses/by-nc/3.0) which permits unrestricted non-commercial use, distribution, and reproduction in any medium, provided the original work is properly cited.
Journal of Life Science 2018 Vol. 28. No. 12. 1477~1488 DOI : https://doi.org/10.5352/JLS.2018.28.12.1477
서 론
제주도 대부분의 해역에서 기후변화로 인한 아열대 현상이 제주 연안 어장 생태계에 변화를 일으키고 있다. 수온이 상승 하고 아열대성 해조류가 정착화되면서 전복과의 주 먹이인 해조류의 서식면적이 감소하며, 자원량을 감소시킨다[21]. 제 주도 연안에서 감소되는 대표적인 해양생물이 오분자기(Sul- culus diversicolor supertexta)이다. 오분자기는 연체동물문 (Mollusca), 복족강(Gastropoda), 전복과(Haliotidae), 오분자기속 (Sulculus)에 속하며, 난대성 연체동물로 주 산란기는 8~11월 로, 주로 미역, 다시마, 감태 등 갈조류를 먹이로 하고 있다.
외양성으로 파도의 영향이 많은 암초지대에서 서식하며 제주
연안과 일부 일본 해안에 분포한다. 최근에는 수온상승으로 인해 오분자기의 서식지가 점차 북상하여 남해안에서도 발견 되고 있다[18].
오분자기를 비롯한 전복과에 속하는 해양 동물들의 국내, 외 연구는 형태학적 특성, 자원 변동, 생리학적 특성 및 양식연 구에 대하여 보고되어 왔으나[18, 26], 여전히 미생물 관련 연 구는 미약한 실정이다.
미생물 군집에 대한 다양성 조사는 해당 생물을 비롯 여러 환경요소와 생태계의 관계를 이해하는데 매우 중요한 기초연 구이며, 생물자원의 질병으로부터의 예방, 개체 보호, 양식 기 술 개발에 있어 중요한 정보 자원으로써 필수로 수행되어야 하는 연구로 그 중요성이 대두되고 있다[40].
그러므로, 오분자기의 자원량 급감에 대한 여러 가지 원인 을 이해하기 위해선 기본적인 오분자기 내 미생물의 공생관계 의 확인과 이해가 필요하다.
해양 유래 미생물은 온도, 염분, pH, 해류 및 강수량 등 다양 한 특성으로 육상과는 다른 환경조건으로 서식환경에 대한 적응 능력이 뛰어나며, 일부 특이한 대사과정으로 잠재력이 무한하며 연구 가치가 높다[13].
본 연구는 오분자기 내 미생물 군집 다양성 조사, 미생물
유전학적 자원 확보 및 인체 유해 질병 세균에 대한 항균 실험 을 수행하며, 해양 유래 미생물의 응용 등 다양한 연구 방면에 기초자료를 제공하고자 하였다.
재료 및 방법
오분자기 채집
본 실험에 사용된 오분자기는 2018년 4월 제주 서부 지역 한림 한수리 연안에서 당일 채집된 시료로 한수리 어촌계를 통해 확보했다. 크기는 평균 각고 56.15±2.51 mm, 평균 각장 38.60±2.23 mm, 평균 무게 28.9±1.86 g로 총 3개체이며, 채취 후 냉장보관하여 운송 후 바로 실험에 사용하였다.
오분자기 내 미생물 분리 및 배양
오분자기 내 미생물의 다양성을 조사하기 위해 멸균된 해부 도구로 패각을 제거하고 부위별 미생물의 종 다양성을 알아보 기 위해 근육, 내장, 생식소 부위를 각각 1 g씩 척출한 후 인공 해수로 세척하였다. Homogenizer로 분쇄 후 voltexting하여 균질화 시킨 뒤 10
-1~10
-5배로 단계 희석을 하였다. 그 뒤 일반 증균 배지인 Brain Heart Infusion Agar, Tryptone Soy Agar, 최소 영양배지 R2A Agar에 오분자기의 서식환경을 고려하여 Sodium chloride를 1%씩 첨가하였고, 일반 해양 미생물 증식 배지인 Marine agar에 100 μl씩 적하하여 25℃에서 최대 14일 간 Incubator (JSGI-100T, JSR, Korea)에서 정체 배양하였다.
그 후 각각 평판 배지상에 자라난 균의 집락의 크기, 모양, 색깔 등 형태학적 특성을 관찰하여 동일한 배지에 계대배양하 여 순수 분리 하였다. 오분자기의 부위별 분리된 colony 수는 CFU/g-
1단위로 환산하였다. 분리된 균주는 24%(v/v) Gly- cerol에 현탁한 뒤 -85℃ Deep Preezer에 냉동 보관하여 추후 실험에 사용하였다.
분자생물학적 동정(16S rRNA 염기서열 계통분석) 오분자기의 근육, 내장, 생식소로부터 분리된 균주의 DNA 염기서열에 기초한 분자생물학적 동정을 위해 Genomic DNA Extraction kit (Bioneer, Korea)를 사용하여 제시된 protocol을 따라 다음과 같이 수행하였다. 각 균주의 배양액 1 ml을 12,000 rpm에서 3분간 원심분리 후 분리된 균체에 PBS buffer를 넣어 섞어준 뒤 Binding buffer를 놓고 새로운 e-tube에 옮겨 원심분 리하였다. 그 후 Washing buffer 1,2를 각각 순서대로 넣고 원심분리 후 건조한 뒤 Elution buffer를 넣고 반응 뒤 원심분 리 후 최종적으로 정제된 Genomic DNA를 추출하였다. 추출 된 DNA로부터 16S rRNA 유전자 증폭을 위해 27 Forward primer (5’-AGAGTTTGATCMTGGCTCAG-3’)와 1492 Reverse primer (5’-TACGGYTACCTTGTTACGACTT-3’)을 사용하였 다. PCR (US/MYGein32, MJ RESEARCH, USA)은 다음과 같 이 수행하였다. 95℃에서 4분간 초기 변성 단계, 94℃에서 1분
간 변성 단계를 수행하였고 55℃에서 1분간 결합 단계, 72℃에 서 1분간 합성 단계를 총 30회 반복한 뒤 72℃에서 5분간 최종 합성하였다. 증폭된 PCR 산물은 0.5 μg/ml ethidium bromide (0.5 ug/ml)가 들어있는 1.5% Agarose gel에서 전기영동을 실 시하여 band를 확인하였다.
16S rRNA염기서열 분석 및 계통도 작성
증폭된 16S rRNA 염기서열 분석은 ㈜제노텍(Daejeon, Korea)에 의뢰하였다. 그 뒤 도출된 염기서열을 미국 국립생 물정보센터 NCBI (National Center for Biotechnology Infor- mation)의 BLAST (Basic Local Alignment Search Tool) pro- gram Genbank의 database를 이용하여 등록된 표준 미생물과 각 분리된 균주의 염기서열의 유사도를 확인하였다. 그 뒤 ClustalW software (www.clustal.org)를 사용 Multiple align- ment를 수행한 뒤 Mega 6.0 program (69.36.184.213/mega.
php)를 사용하여 매뉴얼을 참고하여 Neighbor joinin phylo- genetic tree를 작성하였다. 계통수의 신뢰도 확보를 위해 bootstrap을 1,000회 반복 실행하였다.
인체 유해 질병 균주에 대한 항균 스크리닝
분리된 균주로부터 항균작용물질의 생산 여부를 알아보기 위하여 인체 유해 질병 세균을 대상으로 항균성 실험을 실시 하였다. 실험에 사용된 인체 유해 질병 세균은 미생물 보존센 터(Korean Culture Center of Microorganisms), 생물자원센터 (Korean Collection for Type Cultures)으로부터 분양받았다.
실험에 사용된 인체 유해 세균은 총 4균주로 그람 양성 2종 (Gram positive : Streptococcus mutans KCCM 40105, Enter- ococcus faecalis KCTC 5290), 그람 음성 2종(Gram negative : Escherichia coli KCTC 1682, Enterobacter aerogenes KCTC 2190) 이었으며, 분양받은 균주는 각각 알맞은 배지와 온도에서 24 시간 배양하였다.
항균 스크리닝은 교차획선법(Cross-streak method)으로, 이전 연구 실험을 참고하여 다음과 같이 수행하였다[37]. 이전 오분자기로부터 분리된 193균주의 배양액을 0.5 Mc Farland 로 탁도를 맞춘 뒤 Mueller Hinton Agar (MHA, Difco, USA) 에 접종한 후 25℃에서 48시간 배양하였다. 그 뒤 인체 유해 병원 균주 배양액 또한 동일하게 0.5 Mc Farland로 탁도를 맞춘 뒤 분리 균주와 교차되게 획선 도말하여 최대 48시간 동안 배양하였다. 성장이 저해된 Clear zone은 48시간 후에 확인되었으며, 대조구는 인체 유해 세균의 성장도를 평가하기 위해 실험 대상 균주를 접종하지 않았다.
결과 및 고찰
오분자기의 부위별 미생물 군락 계수
오분자기의 근육(Muscle), 내장(Intestine), 생식소(Gonads)
각 부위별 미생물의 다양성을 알아보고자 각 부위의 sample을
Table. 1. Microbial communities isolated from each organ of Sulculus diversicolor supertexta
Sample No. Each organ. Medium
*MA *1% TSA *1% BHIA *1% R2A **mean counts
1
Muscle Intestine
Gonads
1.9×10
51.8×10
31.0×10
26.5×10
42.2×10
42.7×10
31.7×10
52.3×10
42.1×10
32.4×10
31.95×10
21.7×10
21.06×10
51.17×10
41.2×10
3Mean counts 6.9×10
42.0×10
46.5×10
49.2×10
23.8×10
32
Muscle Intestine
Gonads
2.6×10
52.0×10
31.2×10
24.8×10
41.8×10
41.3×10
31.9×10
53.1×10
41.5×10
32.0×10
32.2×10
21.2×10
21.25×10
51.28×10
47.6×10
2Mean counts 8.7×10
42.2×10
47.4×10
47.8×10
24.5×10
43
Muscle Intestine
Gonads
2.4×10
51.7×10
31.6×10
25.1×10
48.4×10
31.84×10
32.1×10
52.2×10
41.8×10
33.3×10
31.4×10
21.15×10
21.1×10
58.0×10
39.7×10
2Mean counts 8.5×10
42.0×10
47.8×10
41.18×10
34.6×10
4* MA(Marine agar), 1% TSA, 1% BHIA, 1% R2A (Tryptone soy agar, Brain heart infusion agar and R2A agar, each supplemented with 1% Sodium Chloride.) ** unit : CFU/g
-1(colony forming unit)
재료 및 방법에 설명한 바와 같이 얻은 후 평판 배지에 접종하 여 배양하였다. 평판 배지상 자라난 colony의 모양, 색깔 등 형태학적 특징을 구분하여 순수 분리하였으며, 각 부위별 총 균수는 다음과 같이 나타났다(Table 1). 부위별 CFU 값은 근육 이 1.1×10
5CFI/g
-1으로 가장 많이 분리되었으며 순서대로 장 1.0×10
4CFU/g
-1, 생식소 9.7×10
2CFU/g
-1로 나타났다. 배지상 colony가 가장 많이 자라난 배지는 MA배지로 나타났다. 이러 한 결과는 이전 보고된 대부분의 해양 유래 생물로부터 분리 한 미생물들의 최다 배양, 최적 배양의 결과와 같다[20, 22, 27]. 하지만 근육으로부터 분리한 미생물은 사용한 모든 배지 에서 고루 잘 자라났으나 장내 분리 미생물은 해양 미생물 전용 배지로 무기염이 주성분인 MA보다 다양한 고영양분이 들어있는 1% TSA, 1% BHIA에서 더 높은 colony 개수를 나타 내었다. 이 결과는 종속영양 세균으로서 다양한 영양분이 요 구되는 장내세균의 특성에 따른 결과라 사료된다.
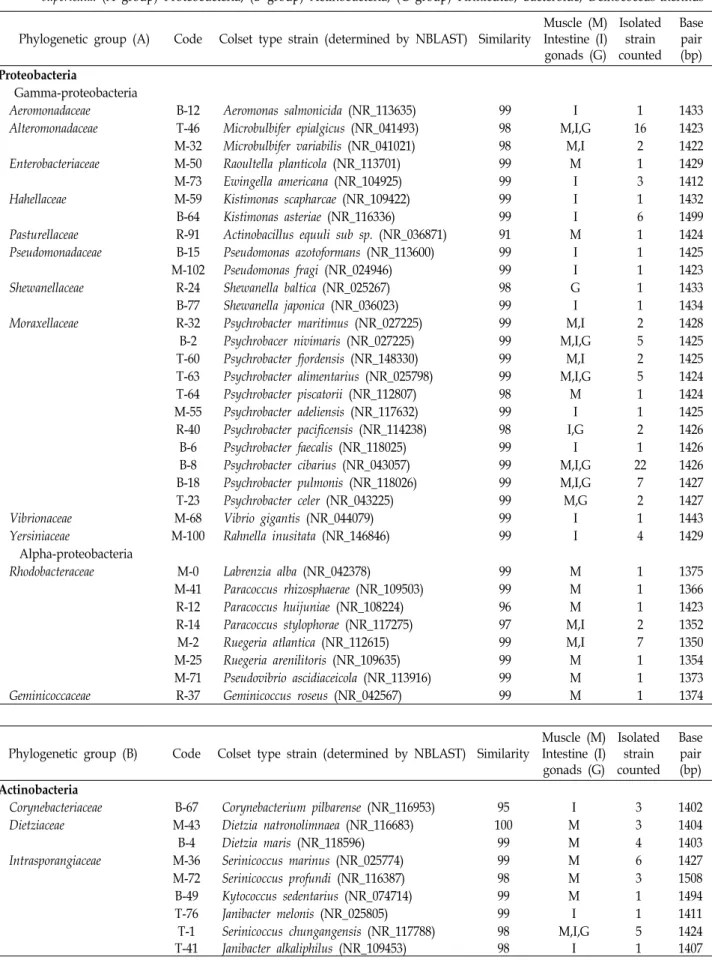
오분자기 내 부위별 미생물 염기서열 및 계통학적 분석 비교 오분자기의 근육, 내장, 생식소 각각의 부위로부터 얻은 190 colony의 16S rRNA Sequence는 각 부위마다 다양한 종으로 나타났다. 도출된 염기서열을 바탕으로 NBLAST program을 분석한 결과 크게 5문 25과 39속 71종으로 나타났다(Table 2).
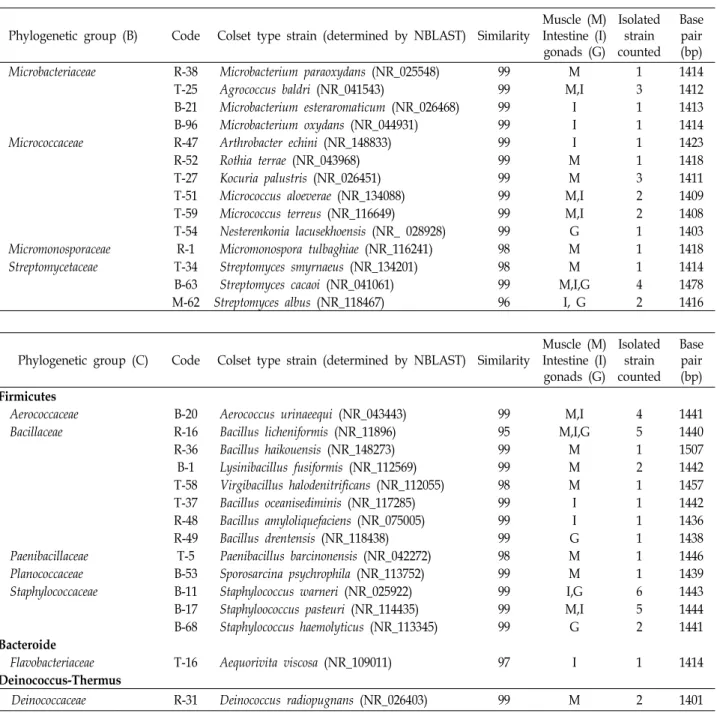
표준 균주와 가장 근연 속으로 나타난 분리 균주 속의 서열 은 Fig. 1에 계통수로 나타내었다.
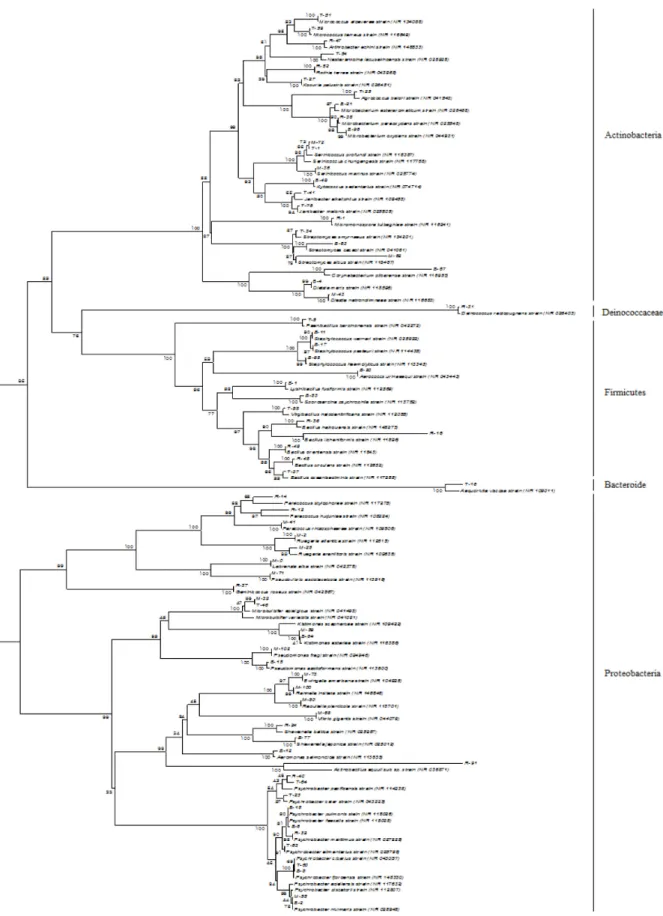
5문(Pylum) 중 Probacteria (Gamma-proteobacteria, Alpha- proteobacteria) 문이 48%로 가장 높게 나타났으며, Actino- bacteria 32.5%, Frimicutes 16.9%, Bacteroide 1.3%, Deinococ- cus-Thermus 1.3%로 각각 나타났으며, 부위별 우점과는 Fig.
2에 나타내었다.
오분자기의 근육으로부터 분리된 균주는 총 44종으로 전체 균주 중 57%를 차지하였다. Protebacteria 문이 25%로 우점 하였다. Gamma-proteobacteria 문은 4과 4속 12종으로 나타났 으며 91-99%의 상동성을 나타냈다. Moraxellaceae 과의 Psyc- hrobacter cibarius가 가장 높은 우점균으로 나타났다. P. cibarius 는 주로 다양한 해양 환경에서 발견된다. 해면과 같은 저서생 물 및 갑각류로부터 분리되었다[41, 44]. 본 실험에서도 내장, 생식소 모든 부위에서 고르게 분리되었다. Actinobacillus equu- li sub sp.는 91%의 낮은 상동성을 나타내었으며, 고래와 같은 바다 포유류 및 대서양 연어(Salmo Salar L.)로부터 분리된 바 있다[2, 10]. 주로 육상생물의 병원성 세균으로 보고되었으나, 현재 수중생물에 대한 병원성은 보고된 바 없다.
Alteromonadaceae 과의 Microbulbifer sp.은 Cellulose, Algi- nate 및 Chitin 등 다당류 분해능을 갖고 있으며[38], M. epi- agicus (98%), M. variabilis (98%)는 해파리(Cotylorhiza tuber- culata), 보라성게(Heliocidaris crassispina)에서도 분리되었다[6, 29]. 계통수 작성 결과 분 리균주 M-32, T-46 strains이 서로 각각의 표준 균주보다 더 가깝게 나타났다.
Alpha-proteobacteria는 2과 5속 8종으로 나타났으며 96-99
%의 상동성으로 나타났다. Rhodobacteraceae 과의 Rugeria at- lantica (99%)가 가장 높은 우점균으로 나타났으며, Rugeria sp 주로 해양 퇴적층에서 분리되며, 이 전복의 장에서도 발견되 었다[15, 48].
96~99%의 상동성으로 나타난 Paracoccus 속은 산호(Stylo-
phora pistillata)와 같은 저서생물, 독가시치(Siganus fuscescens)
의 위장 등 다양한 수중생물로부터 분리되었으며, 전복의 장
에서도 분리되었다[33, 46, 48]. Pseudovibrio ascidiaceicola (99%)
는 이전 조류(Delisea pulchra)에서 분리되었으며 넓은 스펙트
Table 2. Analysis of 16S rRNA sequence of microorganisms isolated from muscle, intestines and gonads of Sulculus diversicolor supertexta. (A group) Proteobacteria, (B group) Actinobacteria, (C group) Firmicutes, Bacteroide, Deinococcus-thermus Phylogenetic group (A) Code Colset type strain (determined by NBLAST) Similarity
Muscle (M) Intestine (I) gonads (G)
Isolated strain counted
Base pair (bp) Proteobacteria
Gamma-proteobacteria
Aeromonadaceae B-12 Aeromonas salmonicida (NR_113635) 99 I 1 1433
Alteromonadaceae T-46 Microbulbifer epialgicus (NR_041493) 98 M,I,G 16 1423
M-32 Microbulbifer variabilis (NR_041021) 98 M,I 2 1422
Enterobacteriaceae M-50 Raoultella planticola (NR_113701) 99 M 1 1429
M-73 Ewingella americana (NR_104925) 99 I 3 1412
Hahellaceae M-59 Kistimonas scapharcae (NR_109422) 99 I 1 1432
B-64 Kistimonas asteriae (NR_116336) 99 I 6 1499
Pasturellaceae R-91 Actinobacillus equuli sub sp. (NR_036871) 91 M 1 1424
Pseudomonadaceae B-15 Pseudomonas azotoformans (NR_113600) 99 I 1 1425
M-102 Pseudomonas fragi (NR_024946) 99 I 1 1423
Shewanellaceae R-24 Shewanella baltica (NR_025267) 98 G 1 1433
B-77 Shewanella japonica (NR_036023) 99 I 1 1434
Moraxellaceae R-32 Psychrobacter maritimus (NR_027225) 99 M,I 2 1428
B-2 Psychrobacer nivimaris (NR_027225) 99 M,I,G 5 1425
T-60 Psychrobacter fjordensis (NR_148330) 99 M,I 2 1425
T-63 Psychrobacter alimentarius (NR_025798) 99 M,I,G 5 1424
T-64 Psychrobacter piscatorii (NR_112807) 98 M 1 1424
M-55 Psychrobacter adeliensis (NR_117632) 99 I 1 1425
R-40 Psychrobacter pacificensis (NR_114238) 98 I,G 2 1426
B-6 Psychrobacter faecalis (NR_118025) 99 I 1 1426
B-8 Psychrobacter cibarius (NR_043057) 99 M,I,G 22 1426
B-18 Psychrobacter pulmonis (NR_118026) 99 M,I,G 7 1427
T-23 Psychrobacter celer (NR_043225) 99 M,G 2 1427
Vibrionaceae M-68 Vibrio gigantis (NR_044079) 99 I 1 1443
Yersiniaceae M-100 Rahnella inusitata (NR_146846) 99 I 4 1429
Alpha-proteobacteria
Rhodobacteraceae M-0 Labrenzia alba (NR_042378) 99 M 1 1375
M-41 Paracoccus rhizosphaerae (NR_109503) 99 M 1 1366
R-12 Paracoccus huijuniae (NR_108224) 96 M 1 1423
R-14 Paracoccus stylophorae (NR_117275) 97 M,I 2 1352
M-2 Ruegeria atlantica (NR_112615) 99 M,I 7 1350
M-25 Ruegeria arenilitoris (NR_109635) 99 M 1 1354
M-71 Pseudovibrio ascidiaceicola (NR_113916) 99 M 1 1373
Geminicoccaceae R-37 Geminicoccus roseus (NR_042567) 99 M 1 1374
Phylogenetic group (B) Code Colset type strain (determined by NBLAST) Similarity
Muscle (M) Intestine (I) gonads (G)
Isolated strain counted
Base pair (bp) Actinobacteria
Corynebacteriaceae B-67 Corynebacterium pilbarense (NR_116953) 95 I 3 1402
Dietziaceae M-43 Dietzia natronolimnaea (NR_116683) 100 M 3 1404
B-4 Dietzia maris (NR_118596) 99 M 4 1403
Intrasporangiaceae M-36 Serinicoccus marinus (NR_025774) 99 M 6 1427
M-72 Serinicoccus profundi (NR_116387) 98 M 3 1508
B-49 Kytococcus sedentarius (NR_074714) 99 M 1 1494
T-76 Janibacter melonis (NR_025805) 99 I 1 1411
T-1 Serinicoccus chungangensis (NR_117788) 98 M,I,G 5 1424
T-41 Janibacter alkaliphilus (NR_109453) 98 I 1 1407
Table 2. Continued
Phylogenetic group (B) Code Colset type strain (determined by NBLAST) Similarity
Muscle (M) Intestine (I) gonads (G)
Isolated strain counted
Base pair (bp)
Microbacteriaceae R-38 Microbacterium paraoxydans (NR_025548) 99 M 1 1414
T-25 Agrococcus baldri (NR_041543) 99 M,I 3 1412
B-21 Microbacterium esteraromaticum (NR_026468) 99 I 1 1413
B-96 Microbacterium oxydans (NR_044931) 99 I 1 1414
Micrococcaceae R-47 Arthrobacter echini (NR_148833) 99 I 1 1423
R-52 Rothia terrae (NR_043968) 99 M 1 1418
T-27 Kocuria palustris (NR_026451) 99 M 3 1411
T-51 Micrococcus aloeverae (NR_134088) 99 M,I 2 1409
T-59 Micrococcus terreus (NR_116649) 99 M,I 2 1408
T-54 Nesterenkonia lacusekhoensis (NR_ 028928) 99 G 1 1403
Micromonosporaceae R-1 Micromonospora tulbaghiae (NR_116241) 98 M 1 1418
Streptomycetaceae T-34 Streptomyces smyrnaeus (NR_134201) 98 M 1 1414
B-63 Streptomyces cacaoi (NR_041061) 99 M,I,G 4 1478
M-62 Streptomyces albus (NR_118467) 96 I, G 2 1416
Phylogenetic group (C) Code Colset type strain (determined by NBLAST) Similarity
Muscle (M) Intestine (I) gonads (G)
Isolated strain counted
Base pair (bp) Firmicutes
Aerococcaceae B-20 Aerococcus urinaeequi (NR_043443) 99 M,I 4 1441
Bacillaceae R-16 Bacillus licheniformis (NR_11896) 95 M,I,G 5 1440
R-36 Bacillus haikouensis (NR_148273) 99 M 1 1507
B-1 Lysinibacillus fusiformis (NR_112569) 99 M 2 1442
T-58 Virgibacillus halodenitrificans (NR_112055) 98 M 1 1457
T-37 Bacillus oceanisediminis (NR_117285) 99 I 1 1442
R-48 Bacillus amyloliquefaciens (NR_075005) 99 I 1 1436
R-49 Bacillus drentensis (NR_118438) 99 G 1 1438
Paenibacillaceae T-5 Paenibacillus barcinonensis (NR_042272) 98 M 1 1446
Planococcaceae B-53 Sporosarcina psychrophila (NR_113752) 99 M 1 1439
Staphylococcaceae B-11 Staphylococcus warneri (NR_025922) 99 I,G 6 1443
B-17 Staphyloococcus pasteuri (NR_114435) 99 M,I 5 1444
B-68 Staphylococcus haemolyticus (NR_113345) 99 G 2 1441
Bacteroide
Flavobacteriaceae T-16 Aequorivita viscosa (NR_109011) 97 I 1 1414
Deinococcus-Thermus
Deinococcaceae R-31 Deinococcus radiopugnans (NR_026403) 99 M 2 1401
럼의 항균활성을 갖는 Tropodithietic acid (TDA)를 생산한다 보고되었다[41].
Actinobacteria 문 6과 11속 15종으로 나타났으며 98-100%
의 상동성을 보였다. Intrasporangiaceae 과의 Serinicoccus mar- inus (99%)가 우점을 차지하였다. 완전한 해양 유래 방선균으 로 해수, 해양 퇴적물에서 주로 발견되며, 수중생물로부터 분 리 보고는 아직 없다[51]. Micrococcus aloeverae (99%)는 해면 (Candidaspongia flabellate and Rhopaloeides odorabile)에서 분리 된 보고가 있다[3]. Streptomyces cacaoi (99%)의 경우 해면, 해조 류(Thalassia hemprichii), 해양 퇴적층 다양하게 분리되었으며 살균제의 항생물질인 Polyoxin을 생산한다[19, 50].
Firmicutes 문은 5과 7속 8종으로 나타났으며 95-99%의 상
동성을 나타냈으며, 각 Aerococcus urinaeequi (99%), Bacillus
licheniformis (95%), Staphylococcus pasteuri (99%)가 우점을 차
지했다. B. licheniformis (95%)의 경우 모든 부위에서 분리되었
으며, 표준 균주와 낮은 상동성을 나타내었다. 일반적으로 위
장기관과 관련이 있으며, 병원성을 갖고 있으나 위험도는 현
저히 낮다. 반면 알칼리성 환경에서도 생존 가능한 protease를
생성한다[34]. 이는 병원성을 갖고 있으나 다양한 방면으로도
응용이 가능한 미생물의 특징을 나타낸다. 이 또한 낮은 상동
성으로 새로운 메커니즘의 물질 개발 가능성을 지닌다고 사료
된다. Paenibacillus barcinonensis (98%)의 경우 ‘ Xylanase’라는
Fig. 1. Neighbour-joining phylogenetic tree based on 16S rRNA gene sequences showing the position of microorganisms strains from each organs (muscle, intestine, gonads) of Sulculus diversicolor supertexta. Bootstrap percentage (from 1,000 replications)
>50% are shown at brach points.
A B
C
Fig. 2. Distribution of each organs bacteria in Sulculus diversicolor supertexta. (a; muscle, b; Intestine, c; gonads.)
다당류 분해 효소 생성능을 갖고 있어, 효소 산업 활용가치의 가능성을 갖는다[35].
Deinococcus-Thermus 문은 1과 1속 1종으로 나타났다.
Deinococcus radiopugans (99%)는 수중생물에서 보고는 비교적 적으나, 해산어인 병치매가리(Parastromateus niger), 제주 연안 에 서식하는 옥돔(Branchostegus japonicas)의 내장에서도 분리 된 바 있다[25, 45].
오분자기의 장으로부터 분리된 균주는 총 41종으로 전체 균주 중 23.2%를 차지하였다. Proteobacteria 문 Gamma-pro- teobacteria가 총 9과 9속 19종으로 나타났으며 근육, 생식소 보다 비교적 더 다양한 균주가 분리되었다. 이 중 Psychrobacter
cibarius (99%)가 가장 높은 우점도를 보였으며, 이는 이전 대서 양 연어(Salmo salar)의 장내세균 우점균의 결과와 같게 나타났 다[42]. 옥돔(B. japonicas), 대서양 대구(Gadus morhna L.)의 장 내세균군에서도 일부 분리되었으며, 남중국해의 해면(Stelletta tenui, Halichondria rugose and Dysidea avara)에서도 발견됐다 [25, 43, 54]. 대부분의 다양한 해양생물로부터 P. cibarius를 비 롯한 P. faecalis, P. pulmonis , P. celer 등 대개, Psychrobacter sp. 속의 많은 종이 분리된 결과를 보았을 때, Psychrobacter sp.는 해양수중생물의 분리되는 주요 우점 미생물 군 중 하나 로 사료된다[52, 55].
Shewanella japonica (99%)는 홍합(Prototaca jedoensis)로 분리
됐으며, alginate 분해능을 갖고 있는 것으로 나타났다[17].
Shewanella sp.은 이전 전복(Halio tis discus hannai)의 장으로부 터 분리되었다[23].
분리 균주 중 Kistimonas sp. 2종은 장내세균에서 단독으로 분리되었으며 키조개(Atrina pectinate)의 창자, 불가사리 (Asterias amurensis)에서 분리되었다[7, 28].
Vibrio sp.는 이전 연구에서 전복류의 장내세균 중 주요 우 점을 차지하는 종으로 나타났다[12, 14, 48]. 본 실험에서 분리 된 V. gigantis (99%)의 경우 이전 전복(Haliotis giganteo)의 장 내로부터 분리된 적이 있다. Vibrio sp.의 일부는 alginate를 분 해하는 것으로 나타났으며, V. gigantis 또한 분해능을 갖고 있는 것으로 나타났다[8].
Actinobacteria 문은 총 5과 8속 12종으로 분리됐으며, Seri- nicoccus chungangensis (98%)가 우점균으로 나타났다. M-72, T-1 Strain의 경우 각각 표준 균주와 S. profundi (98%), S. chun- gangensis (98%)의 상동성을 나타냈으나 계통수 작성 결과 표 준 균주보다 서로 더 가까운 상동성을 나타냈다. Janibacter al- kaliphilus (98%) 균주는 중국 산호 Anthogorgia로부터 분리되 었으며[31], 표준 균주와 낮은 상동성을 나타냈다. Agrococcus baldri (99%)는 제주 연안에 건조된 바닷말에서 분리된 바 있다 [30].
Microbacterium oxydans (99%)는 최근 한국 부산 연안에서 다시마(Laminaria japonica)로부터 분리되었으며, Laminarin 생성능을 나타냈다. 이 다당류는 인체의 혈관 개선에 매우 좋 은 성능을 갖고 있는 것으로 나타나[24], 응용 미생물로서의 잠재력이 높다 사료된다.
Firmicutes 문은 3과 3속 6종으로 분리됐으며 Staphylococ- cus warneri (99%)가 우점균으로 나타났다. 육상생물의 병원 인자로 알려져 있지만, S. warneri, S. pasteuri는 아열대 해양 어류로부터 빈번하게 발견되나 해양생물에 대한 병원성은 보 고되지 않았다[4].
Bacillus amyloliquefaciens (99%)는, 북방 전복(Haliotis discus hannai)의 성장과 Vibrio sp. 질병예방으로서 Probiotic 효과가 입증된 바 있다[39]. 그 외 Areolate grouper (Epinephelus areo- latus), 옥돔(B. japonicas)의 장내로부터 분리된 바 있다[1, 25].
Bacteroide 문은 1과 1속 1종으로 Aequorivita viscosa (97%) 의 낮은 상동성을 나타내었다. 식물성 플랑크톤, 회색 산호 (Orbicella faveoata), 해초(Cystoseria compressa) 등 조간대로부 터 분리됐다[32, 53].
생식소에서는 다른 부위에 비해 적은 종의 세균 군이 분리 되었다. 우점균은 근육, 장과 동일하게 Proteobacteria에서 나 타났으며, Acitnobacteria 문 중 Micrococcaceae 과의 Nesteren- konia lacusekhoensis (99%), S. haemolyticus는 중국 남부의 태평 양 전복에서도 동일하게 분리됐다[47].
전복류를 비롯한 해양무척추동물의 세균 군집의 연구결과 는 proteobacteria의 Gamma-proteobacteria 문이 우점하였으
며, Vibrio, Pseudomonas, Flavobacterium, Micrococcus, Aeromo- nas가 상당 수로 나타났다[11, 12, 14, 47, 48]. 본 실험에서도 대부분이 동일하게 나타났다. 하지만 Microbulbifer sp., Janibacter sp., Deinococcus sp., 등 이전 전복류에서 보고되지 않은 종들이 분리되었으며 이와 같은 결과는 생활 서식지 환경 등 여러 요인에 따라 미생물 군집은 다양한 차이를 나타내는 것을 알 수 있었다.
Rugeria arenilitoris, Dietzia natronolimnaea의 경우 주로 열대 성 토양과 온난 해수 및 아열대 생물에서 분리되는 고온성 박테리아로[15, 49] 온난화에 따른 제주 연안의 수온 변화가 미생물에 미치는 영향 중 하나의 사례로 생각할 수 있다.
본 실험에서 분리된 오분자기 유래 균주들은 이전 연구결과 와 비교하였을 때, 조류, 해초, 해면, 퇴적층에서 분리된 사례 가 많게 나타난 것을 알 수 있다[30, 32, 38, 41, 46, 50, 53, 54]. 이런 결과로 미루어 볼 때 바위, 암초, 조류가 많은 곳에서 부착생활을 하는 오분자기의 서식 특성과 큰 관련이 있는 것 으로 사료된다.
오분자기와 같은 전복류 H. disucus hannai를 비롯 많은 이 전의 전복 내 미생물 군집 조사 결과, 분리한 박테리아 중 대부 분이 alginate 분해능이 있는 것으로 확인되었다[14, 47]. 본 실험에서도 alginate 분해능이 밝혀진 균주(Microbulbifer sp., Microbacterium sp., Vibrio sp., shewanella sp.,)가 여러 분리되 었다. 조류는 오분자기의 주 먹이이며, 알긴산과 같은 다당류 로 구성된 세포벽을 갖고 있다. 이런 균주들은 오분자기의 조 류 소화능에 관여하는 것으로 사료되며, 이런 결과는 먹이 습 성이 오분자기의 장을 비롯한 미생물 군집 구성에 큰 영향을 미칠 수 있음을 암시한다.
16S rRNA sequence 분석 결과 표준 균주와 낮은 상동성을 나타낸 균주(C. pilbarense 95%, B. lichenformis 95%, S. albus 96%, P. huijuniae 96%, P. stylophorae 97%) 들을 확인할 수 있었 다. 대부분이 다양한 해양 환경으로부터 분리되지만, 잦은 환 경 변화에 적응 능력이 높은 해양미생물의 특성을 염두에 둔 다면 신 균주의 개발, 새로운 메커니즘의 천연물질 개발 가능 성을 생각해 볼 수 있다.
이처럼, 미생물 군집 조사는 숙주의 생활사 또는 주변 생태 계의 변화를 이해하는데 많은 도움이 되며, 균주들의 대사산 물로부터 새로운 물질 개발 가능성의 모니터링이 가능하다.
미생물 군집 실험은 크게 배양법과 비배양법으로 나뉜다. 본 실험에서 실행한 배양법은 분리할 수 있는 종이 전체 중 극히 일부이지만, 아직 밝혀지지 않은 나머지 수많은 균주들의 배 양학적 특성, 생화학 특성을 조사하기 위해 가장 기초적인 연 구로 생각하여 수행하였다.
분리된 균주의 인체 유해 질병 균주에 대한 항균활성
총 4가지의 인체 유해 질병 균주에 대한 항균실험의 결과
분리균 71균주 중 3균주가 항균활성을 나타냈으며 균주별로
A B C
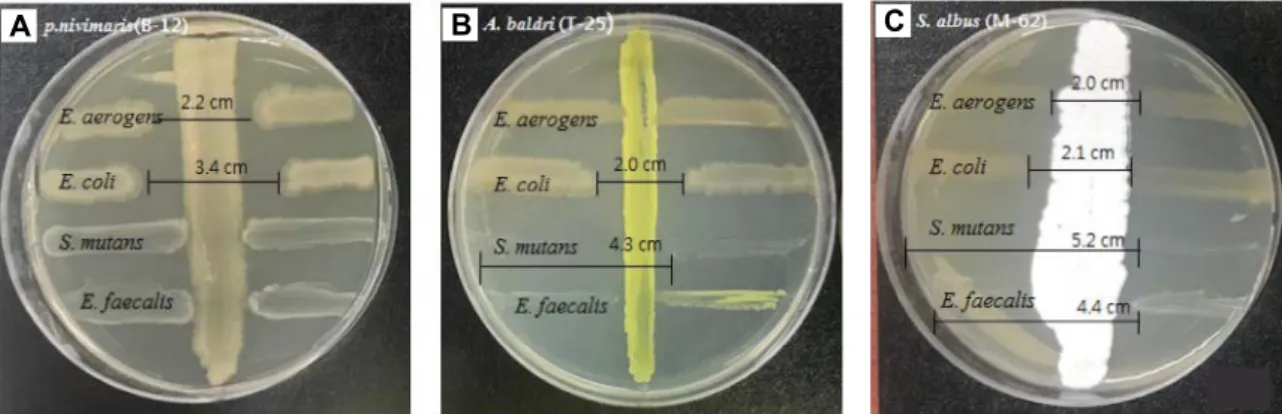
Fig. 3. Antimicrobial monitoring of human harmful disease by cross- streak method of Microorganisms isolated from Sulculus diversi- color supertexta (harmful disease of human body: gram negative; Enterobacter aerogens KCTC 2190, Escherichia coli KCTC 1682, gram postitive: Streptococcus mutans KCCM 40105, Enterococcus faecalis KCTC 5290), (tested organisms; A;P. nivimaris (B-12), b; A. bibardi (T-25), S. albus (M-62)
각각 다르게 나타났다(Fig. 3).
해양 유래 방선균인 S. albus (M-62)는 해면과 해양 퇴적층 에서 분리됐으며, Pseudodisaccharide, Aminoglycoside라는 항균물질을 생산하며 항균, 항종양 능력이 보고되었다. 호기 성의 그람 음성 막대균에 대하여 항균작용을 갖지만 일부 그 람 양성 균주에도 항균능을 갖는다[5, 16]. 본 실험에서는 그람 음성 균주에 비하여 S. mutans, E. faecalis 두 양성 균주의 성장 을 확실히 억제한 것으로 나타났다.
A. Baldri (T-25)는 이전 해양 퇴적층으로부터 분리되었으며 Listeria monocytogens에 대한 항균 작용이 보고되었다[9]. 본 실험에서는 E. erogenes를 제외한 그람 음성, 양성 균주에 대하 여 억제능을 보였으나 특히 S. mutans, E. faecalis 그람양성균주 에 대한 효과가 더 높게 나타났다.
P. nivimaris (B-12) 균주는 그람 음성 균주에 대하여 억제 효과를 보였다. 이전 산호에서 분리된 P. nivimaris는 Candida albicans, Staphylococcus epidermidis, Bacillus subtilis와 같은 그 람 양성균에 대하여 항균능을 보이고 E. coli와 같은 그람 음성 균주에는 항균능을 나타내지 않았다[36]. 하지만 본 실험에서 는 E. coli, E. aerogens 그람 음성 균주에 한정하여 항균능이 나타났다. 이런 항균능의 차이는 아무리 같은 종이라도 각 분 리 지역 환경의 특성에 따라 만들어지는 대사산물이 다양한 항균활성을 나타내는 것을 알 수 있으며, 이에 따른 추가적인 생화학적 특성 검토가 필요하다고 사료된다.
본 실험에서는 항균활성을 갖는 균주가 다소 적게 분리되었 지만 각 분리 균주 마다 한 균주에 국한된 항균능이 아닌 다소 넓은 범위의 항균능을 나타내었다. 그리고 기존의 연구와는 다른 항균작용기전을 갖는 균주를 확인할 수 있어 새로운 항 균물질의 가능성도 확인할 수 있었다. 특히 그람양성에 대하 여 확실한 억제능을 보인 S. albus (M-62) 경우 기존 표준 균주 와 96%의 낮은 상동성을 나타내므로 새로운 천연물질 개발을 위한 잠재력을 갖는 후보 균주로 가능성을 나타내었다.
감사의 글
이 논문은 2018년도 국립수산과학원 수산과학연구사업
「제주주변 연근해어업 및 환경생태 조사(R2018031)」의 지 원으로 수행된 연구입니다.
References