Genome 분석에서의 Microarray 응용 Genome 분석에서의 Microarray 응용Genome 분석에서의 Microarray 응용
김 유 리((주)엘앤씨바이오 차장)
DNA Microarray (DNA chip)란 genome상의 변화를 반영할 수 있도록 디자인된 특정 nucleotide sequence (probe)를 합성하여 수 십 만개 이상의 probe를 고집 적으로 spotting해 놓은 것을 말한다. 초기에는 합성 된 oligo를 실험실에서 arrayer (or spotter)를 이용하여 직접 spotting하여 실험에 이용하는 homebrew chip이 많이 사용되었으나 상용화된chip으로 빠르게 대체되 고 있다.
Microarray 기술이 유전체 분석에 이용하게 되었을 때는 RNA level에서 gene expression pattern의 변화를 분석하기 위한 expression chip으로부터 시작되었다.
몇 년이 지난 지금 microarray의 응용 범위는 gene expression 뿐 아니라 DNA copy number change, Protein-DNA interaction, Splicing, Methylation등 다양 한 genomics 연구의 흐름에 맞추어 빠른 속도로 확대 되고 있다.
연연구의 목적에 따라 probe sequence를 디자인 하는 방법도 다양화 되었고, target region도세분화 되었다.
DNA level로 microarray의 응용범위가 확대되면서 genome–wide detection이 연구 목적의 중요한 부분을 차지하게 되었다. 이는 한번의 실험으로 분석 할 수 있는 probe의 숫자가 많을수록 즉, genome coverage가
높을수록 실험의 효율성과 경 제성을 높일 수 있으므로 보다 많은 probe를 spotting 하기 위한 기술이 개발되고 있다. 현재는 십 만개 단위의 probe를 spotting 할 수 있으나 곧 백만개의 probe 를 spottiong 할 예정임을 microarray 제조사들이 밝히고 있다.
Microarray의 응용범위와 제 품의 수는 증가 나날이 증가하 고 있다. 그러나 microarray는 그 특성상 대량의 data를 한번
에 얻게 되므로 이 중에서 연구의 목적이었던 핵심 정보를 찾아내는 data mining에 대한 기본 지식이 요 구되는 분야이다. 따라서 연구의 세부목적을 면밀히 검토하여 세우고 연구의 기본적인 목적에 맞는 probe set을 가지고 있는지, genome coverage는 어떠한지, 어느 정도의 density를 필요로 하는지 등을 고려하여 최적의 microarray platform를 선택하는 것이 유의한 microarray 실험 결과를 얻기 위한 출발점이라고 할 수 있다.
1) Gene Expression microarray
Gene expression pattern의 변화 양상을 분석하기 위 한 microarray이다. Sample로부터total RNA를 추출한 후 cDNA를 합성하여 two color나 one color로 labeling 하여 array에 hybridization하는 방법을 사용한다. Two color system은 cy3 와cy5로 각각 labeling 한 대조군 과 실험군을 섞어 하나의 array에 hybridization 시킨
그림 1) Human breast cancer cell line에서 miRNA expression microarray 를 이용한 실험 결과 (by Agilent Technologies Inc. JAPAN, Analyzed with GeneSpring GX)
다. One color system은 하나의 dye를 사용하여 대조 군 하나와 여러 개의 실험군을 각각 별도의 array에 hyb한다. Sample의 숫자가 많고, 대조군의 양이 충분 하지 못한 경우 one color system이 적합하다.
유전자 염기서열이 이미 밝혀진 다양한 종(species) 의 transcripts 자료를 기반으로 많은 수의 array가 개 발되어 판매되고 있다. 가장 최근의 application으로 는 sequence homology가 높은 miRNA의 expression pattern을 100ng의 total RNA를 sample로 하여 효과적 으로 파악 할 수 있는 miRNA expression microarray (Agilent Technologies Inc.)가 있다(그림1).
2) Array CGH
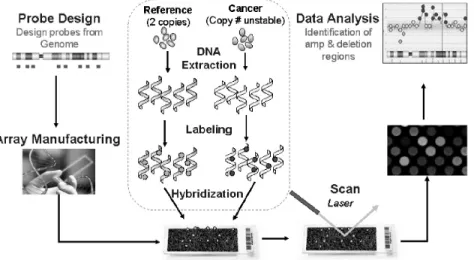
Chromosome상의 숫적 변화를 detection하는 cytological 방법으로는 FISH, Karyotyping등이 있으나 이들 방법들 은 resolution이 낮고 low throughput이라는 실험상의 한계가 있다. 이러한 한계를 극복하고자 도입된 방법이 array CGH (Comparative Genomic Hybridization) 이다.
DNA copy number의 변화를 측정, 정량 하기 위해 정상 군과 실험군의 genomic DNA를 서로 다른 dye로 표지하 여 비교 분석하는 것이다. 초기에는 BAC clone이나 cDNA를 probe로 사용하였으나 최근 oligo probe를 이용
그림 2) array CGH workflow
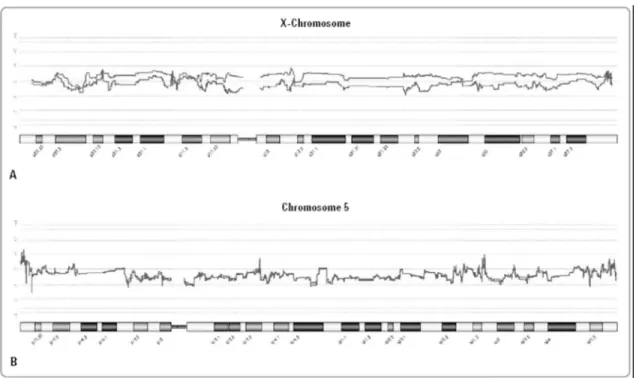
그림 3) array CGH를 이용한 Copy number deletion detection(Analyzed with CGH Analytics, Agilent Technologies Inc.)
한 논문들이 발표되고 있다. Oligo –based aCGH는 short oligo (~25-30mer)와 long oligo (50-70mer)로 나뉘 며, probe 길이에 따라 적용되는 application에 차이가 있다.
Array CGH는 주로 Cancer, 유전성 질환 등에서 보여 지는 DNA copy number change를 파악하는 것이 주
목적이므로 genic region, intergenic region, telomere region, miRNA region을 포함하고, 일정한 간격으로 probe를 제작하는 tiling array보다는 변화 가능성에 비 례적인 probe 디자인이 보다 효율적이라 할 수 있다.
Array CGH는 대조군과 실험군의 genomic DNA를 사용한다 (그림 2). Intact한 genomic DNA를 확보하는
그림 4) Chip-on-chip workfolw
것이 정확한 분석을 위한 기본 조건이다. 그러나, 주된 연구분야인 임상 sample의 상태는 보관형태나 기간에 따라 degradation 일어난 genomic DNA를 실험에 이용해 야 하는 경우가 발생한다. 따라서 degradation된 임상 sample을 활용할 수 있는 protocol을 제공하는 CGH platform을 선택하는 것이 중요하다.
3) ChIP-on-chip (Chromatin immuno precipitation on chip)
Micorarray는 gene expression pattern을 파악하는 것 에서 발전하여 이에 관여하는 DNA binding protein의 behavior를 파악하고자 하는 데에도 사용될 수 있다.
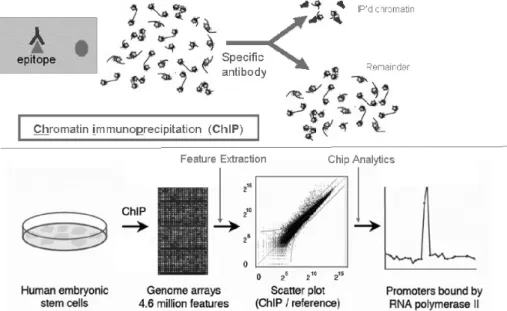
ChIP-on-chip은 location analysis라고도 불리어지 며, Transcription factor나 histone과 같은 DNA binding protein의 chromosome상의 binding 위치를 분석할 수 있는 방법이다.
Target protein이 DNA에 binding된 상태의 cell을 고정 하여 DNA를 작게 잘라 target protein이 결합된 DNA fragment만을 antibody를 이용해 분리한다 (Chromatin immuno precipitation, chIP). 이를 amplification한 후
array에 hybridization 시킨다 (그림 4 참조)
ChIP-on-chip microarray는 Yeast 에서 Histone modification pattern을 genome wide로 screening 하거나 (Pokholok et al., 2005), Human Stem cell 에서 TF의 co-regulation을 밝혀내는데 사용 되었다 (Boyer et al., 2005).
Chip-on-chip 실험에서 가장 중요한 것은 target protein에 specific한 affinity를 갖는 antibody를 확보하 는 것과 target protein이 chIP 단계에서 높은 affinity를 가지고 DNA에 binding 함으로서 reference와 확연히 구별되는 binding intensity를 갖는 chIP product를 얻 는 것이다. 이러한 관점에서 다양한 chIP 방법들 중에 서 가장 효율적인 방법을 찾아내는 것이 선행되어야 한다. 아울러 Microarray는 target region (ex. promoter, encoding region)에서의 변화를 정확히 detection할 수 있도록 디자인된 probe와 검증된 protocol 및 분석 프 로그램으로 실험상의 변수를 최대한 줄일 수 있는 platform을 선택하는 것 또한 중요하다.
4) Methylation array
DNA methylation 연구는 DNA 서열을 바꾸지 않고
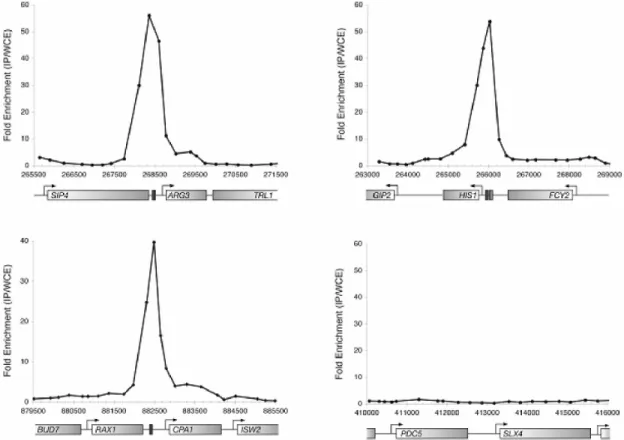
그림 5) Data example:GCN4 binding for known (unknown) targets in Yeast (Pokholok et al., Cell 2005 Aug 26;122(4):517-27)
그림 6) Affinity-based isolation of methylation DNA
gene expression이 조절되는 Epigenetics의 관점에서 출발한다.
DNA methylation은 CpG island에서 주로 관찰되며 고등 Eukaryote에서는 주로 발생, 분화 과정 조절, chromatin 구조 및 안정성, gene silencing, genomic imprinting, X-inactivation, cancer에 관여하는 것으로 알려져 있다. Methylated CpG island를 많이 갖고 있는
"Hypermethlyated gene"은 expression이 감소 또는 중지 되는 반면, promoter CpG islands는 "Hypomethylated"의 경향성을 보이며 대체로 repression 되지 않는다.
Methylation microarray는 methylation이 된 부분을 선택적으로 분리하여 2-color로labeling한 후 CpG island에서 디자인된 probe가 spotting 되어있는 array 에 hybridization 시키는 과정으로 진행된다(Agilent
그림 7) Comparison of methylated DNA (mDIP-enriched) between male HEL299 and female IMR90 embryonic lung fibroblast cell lines.
CpG island Microarray의 경우) (그림 6). Methylation 된 DNA fragment 분리하는 대표적인 방법에는 affinity method (mDIP)와 Restriction enzyme based method등이 있다.
References
Pokholok D. K. et al., Genome-wide Map of Nucleosome Acetylation and Methylation in Yeast Cell 122, 517–
527 (2005)
Boyer L.A. et al., Core Transcriptional Regulatory Circuitry in Human Embryonic Stem Cells Cell 122, 947–956 (2005)