학 술 논 문
105
MRI 영상 유도 수술 로봇을 위한 개선된 군집 분석 방법을 이용한 뇌종양 영역 검출 개발
김대관 · 차경래 · 승성민 · 정세미 · 최종균 · 노지형 · 박충환 · 송태하
대구경북첨단의료산업진흥재단 첨단의료기기개발지원센터
Development of Brain Tumor Detection using Improved Clustering Method on MRI-compatible Robotic Assisted Surgery
DaeGwan Kim, KyoungRae Cha, SungMin Seung, Semi Jeong, JongKyun Choi, JiHyoung Roh, ChungHwan Park and Tae-Ha Song
Medical Device Development Center, Daegu-Gyeongbuk Medical Innovation Foundation (Manuscript received 2 January 2019 ; revised 13 June 2019 ; accepted 18 June 2019)
Abstract: Brain tumor surgery may be difficult, but it is also incredibly important. The technological improvements for traditional brain tumor surgeries have always been a focus to improve the precision of surgery and release the potential of the technology in this important area of the body. The need for precision during brain tumor surgery has led to an increase in Robotic-assisted surgeries (RAS). One of the challenges to the widespread acceptance of RAS in the neurosurgery is to recognize invisible tumor accurately. Therefore, it is important to detect brain tumor size and location because surgeon tries to remove as much tumor as possible. In this paper, we proposed brain tumor detection procedures for MRI (Magnetic Resonance Imaging) system. A method of automatic brain tumor detection is needed to accurately target the location of the lesion during brain tumor surgery and to report the location and size of the lesion. In the qualitative assessment, the proposed method showed better results than those obtained with other brain tumor detection methods. Comparisons among all assessment criteria indicated that the proposed method was significantly superior to the threshold method with respect to all assessment criteria. The proposed method was effective for detecting brain tumor.
Key words: Medical imaging, MRI, K-Means, Robotic-assisted surgery, Image processing, Brain tumor
I. 서 론
현대 뇌종양 수술은 뇌의 손상을 최소화 하면서 뇌종양을 최대한 제거 하는 방향으로 많은 발전을 거듭해 왔다[1]. 몸의 가장 중요한 부위 중에 하나인 뇌를 수술 하기 위해서 뇌 수술 영역의 의료 기술은 미세 수술법을 통해 뇌 종양 제거에
대한 정확도를 높이는데 초점을 두었다[1]. 현대에서 의료 로봇과 수술 보조 장치, 영상 분석 분야 등의 최신 기술들의 도입은 수술 부담이 큰 뇌, 척추 등의 부위에 안전하고 정 교한 수술이 가능하게끔 하였으며, 특히 수술 보조 로봇 (Robotic-Assisted Surgery) 은 미세 수술법의 정확도를 향상 시키는 중요한 역할을 하게 되었다. 특히, 미세 수술을 위한 수술 보조 로봇에서 환자의 상태를 촬영 하는 자기공명영상 (Magnetic Resonance Imaging, MRI), 컴퓨터단층촬영 (Computed Tomography, CT) or 양전자단쵤영(Positron Emission Tomography, PET) 와 같은 영상 기기가 중요한 부분을 차지 하고 있다[2]. 의료영상기기에서 얻은 영상 정 보를 기반으로 수술 전에는 병변의 위치 및 상태를 파악하 여 수술 계획을 세우게 되며, 수술 중에는 수술 전에 작성 Corresponding Author : KyoungRae Cha
Medical Device Development Center, Daegu-Gyeongbuk Medical Innovation Foundation, 80 Cheombok-ro, Dong-gu, Daegu, Republic of Korea
Tel: +82-10-5658-5029 E-mail: orotham@dgmif.re.kr
본 연구는 산업통상자원부 산업핵심기술개발사업(과제번호: 10080609)
의 지원으로 수행되었습니다.
106
한 계획 및 다양한 센서를 활용하여 의료진이 볼 수 없는 위치 및 병변의 변화를 예측하여 안전하게 수술을 수행 할 수 있게 한다[2]. 뇌종양 제거 수술에서도 MRI 시스템에서 얻어진 데이터의 처리가 중요한 역할을 하는데, 성공적인 뇌 수술을 위한 중요한 목표 중에 하나인 종양 조직을 정확하 게 확인하고, 수술 위치 및 도구를 판단하는데 이용되기 때 문이다[2]. 뇌종양 판단과 분석은 숙련된 전문의 에게도 시 간 소모가 크며 어려운 작업이기 때문에, 다양한 분야의 기 술을 적용하여 뇌 종양 검출 기술에 대한 연구가 활발히 이 루어지고 있다. 특히, 최근에는 딥 러닝 기법을 이용한 연구 가 많은 연구자들 사이에 주목을 받고 있다[3]. 의료 분야 에서는 기본적으로 영상을 통해 진단이 많이 이루어지기 때 문에 영상에서 특징을 추출하는데 특화된 합성곱 신경망 (Convolutional Neural Network, CNN) 이 가장 많이 활 용되고 있는 추세이다[3]. 빠르고 정확하게 뇌 종양을 검출 을 위해 CNN을 적용한 몇 가지 연구들[4-7]은 좋은 성능 의 알고리즘을 보여 주기도 하지만, 검출을 위해 많은 데이 터와 준비 작업이 필요하기 때문에 이 부분의 개선을 위한 지속적인 연구가 필요로 한다. 뇌 종양 검출 방법에는 현재 CNN 이외에도 전통적인 방법인 히스토그램(Histogram) 분석, 임계 처리(Thresholding), K-평균 알고리즘(K-means algorithms) 등을 이용한 연구도 과거에서부터 지속적으로 이루어 지고 있으며, 이러한 영상 처리 방법들은 다양한 개선 연구를 통해 성능을 향상을 보여 주고 있다[8-15].
본 연구에서는 MRI 뇌 종양 검출을 위한 방법으로 K-means 을 개선한 방법을 제안한다.
현재 뇌종양 검출에 이용되는 대표적인 알고리즘으로는 CNN, 모폴로지 연산(Morphological Computation), 군집 분석(Clustering analysis), 히스토그램 분석(Histogram analysis), 특징 추출(Feature extraction)와 웨이블릿(Wavelet) 방법들이 대표적 이다[16-20]. 이러한 방법들은 좋은 성능과 결 과를 보여 주지만, 영상 분석을 위한 복잡한 준비 단계와 알고 리즘으로 인해 실시간 적용에 어려움이 발생한다. 우리는 실시 간 영상 처리를 위해 논문에서 뇌 종양 검출에 많이 쓰이는 K- means 방법을 뇌 종양 수술을 위한 수술 보조 로봇에 적용 하 기 위해 몇 가지를 수정하였다. 본 논문에서 연구된 뇌 종양 검 출 알고리즘은 두 단계로 나누어 개발 되었으며, 첫번째 단계 는 뇌 종양 검출을 위한 최적의 군집 개수 파악, 두번째 단계는 유클리드 거리 함수를 이용한 실시간 뇌 종양 검출 단계로 이 루어져 있다. 이러한 단계별 알고리즘 적용은 수술 중간에 촬 영된 영상에 대해 실시간 뇌 종양 검출이 가능하게 하였으며, 다른 알고리즘과 비교 하였을 때 좋은 결과를 보여 주었다.
본 논문은 각 단계별 사용된 군집 결정 및 종양 검출 방법 에 대해 설명하며, 결론 부분에는 본 시스템에서 사용된 알고 리즘의 성능을 비교 평가하기 위한 실험 결과를 보여 준다. 마 지막으로 결론 부분에서는 본 논문에서 소개된 알고리즘을 적 용한 전체 시스템 및 향후 연구 방향에 대해 기술한다.
II. 연구 데이터 및 방법
1. 연구 데이터
본 연구에서는 the Brain Tumor Image Segmentation
그림 1. BRATS 2015 데이터(MRI, Image size: 512×512 pixels, Voxel size: 1×1×1 mm).
Fig. 1. Sample MRI from BRATS dataset [15].
107 2015 (BRATS 2015) 데이터 베이스에서 획득한 영상을 사
용하였다[21]. BRAT 2015데이터는 T1강조영상, T1 조영 증강영상, T2 강조영상와 FLAIR (fluid attenuated inversion recovery) 의 4가지 영상 기법으로 획득한 고악성도신경교종 (high-grade glioma, HGG) and 저악성도신경교종(low- grade glioma, LGG) 2 가지 타입의 뇌종양으로 구성되어 있다.
본 논문에서 사용된 데이터는 FLAIR 영상 기법으로 획득한 HGG 의 30개의 영상으로, 각 영상은 512 × 512 픽셀과 1 mm×1 mm×1 mm 복셀로 되어 있다(그림 1). 본 연구는 CPU i7-2600, 메모리 8GB, 비디오 카드 GeForce GTX 560TI, OPENCV 3.0 과 Visual studio 2015을 사용하는 컴퓨터 시스템에서 진행되었다.
2. 연구 방법
실시간으로 뇌 종양 영역을 정확하게 구별하여 개발 진행 중인 MRI유도기반의 수술 보조 로봇 시스템(그림 2)에 적 용하기 위해 본 논문에서는 수정된 K-means 방식을 이용 한 군집방식으로 개발된 영상처리 시스템을 제안한다. 본 연 구에서 K-means 방식을 적용한 이유는 크게 2가지로 나누 어 진다. 첫번째는 K-means 방식은 주어진 자료에 대한 사 전 뇌 분할 부분에서 널리 쓰이는 방식으로 그 정확성이 여 러 논문을 통해 입증되어 있는 방법이다[9,24]. 두번째는 간 단한 알고리즘으로 인해 짧은 계산 시간 때문에 실시간 뇌 영역 종양 검출 연구에 적합하다는 것이다. 하지만, K-means 방 식의 단점으로 초기 군집 수 결정이 중요한 역할을 하기 때 문에 군집 수가 원 데이터 구조에 적합하지 않으면 좋은 결 과를 얻을 수 없다는 점이다. 초기 군집 개수 선정에 따라 어떻게 설정하느냐에 매우 다른 결과를 얻을 수 있다[25].
따라서, 본 논문에서는 기존의 방법인 사전에 임의로 군집 개수를 입력하여 영상을 분할한 방법을 수정하여, 제안된 영 상 처리 시스템을 개발하게 되었다.
MRI- 영상유도기반의 수술 보조 로봇 시스템은 사전 수술
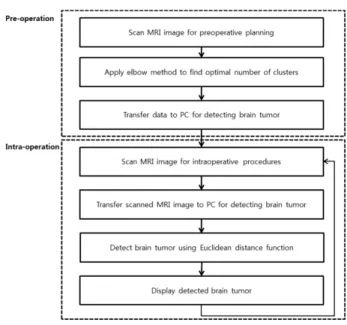
계획 부분과 수술 진행 부분으로 나누어져 운영 되기 때문에, 본 연구에서 제안된 영상 처리 시스템 또한 수술 보조 로봇 시스템에 맞추어 수술 계획과 수술 진행, 2가지 단계로 나 누어져 있다(그림 3). 그림 4은 본 논문에서 제안하는 영상 처리 시스템의 구동 방식에 대한 다이어그램을 보여 준다.
시술 전(pre-operation) 준비 단계에서는 엘보우 기법을 이 용한 군집 개수 결정 알고리즘을 사용하여 군집 변경 내의
그림 2. 개발 진행 중인MRI 유도 수술 보조 로봇 시스템.Fig. 2. MRI-compatible robotic assisted surgery system.
그림 3. 제안된 영상 처리 시스템 구상도.
Fig. 3. Proposed brain tumor detection system from MRI- compatible robotic assisted surgery.
그림 4. 제안된 영상 처리 시스템의 다이어그램.
Fig. 4. Block diagram of proposed brain tumor detection system from MRI-compatible robotic assisted surgery.
108
동질성과 이질성을 측정하여 최적의 군집 개수를 결정하고, 결 정된 군집 개수에 따라 각 군집의 중심 값을 찾아 낸다. 군 집의 중심 값은 시술 중 영상에 적용되기 위해 실시간 MR 영상처리 시스템으로 전송되어, 시술 중에 출력되는 MR 영 상 데이터 와 준비 단계에서 전송된 군집의 중심 값 사이의 유사도를 유클리드 거리 방법을 적용 한 종양 검출 알고리 즘을 사용하여 목표가 되는 군집 영역만을 검출한다. 검출 된 군집은 후 처리(post-processing) 과정을 거쳐서 최종으로 종 양의 위치와 크기를 화면에 표시하게 된다.
3. 군집 개수 결정을 위한 알고리즘
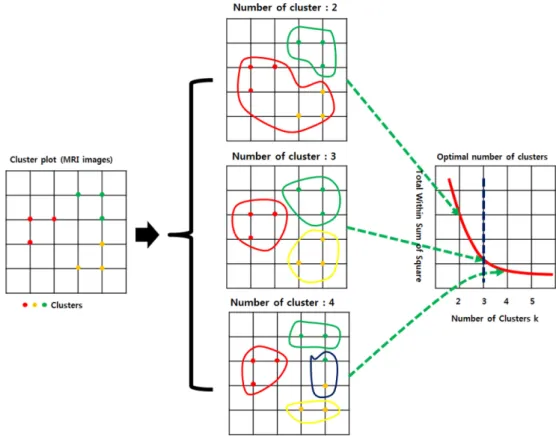
본 장에서는 사용된 군집 개수 결정에 사용되는 방법과 그 알고리즘에 대해 알아보기로 한다. 실시간으로 주어지는 MR 영상을 K개의 동질적인 집단으로 분류하기 위해서 군 집 개수와 목표 군집을 선택해야 하는데, 군집 개수 결정은 군집의 형성 및 군집 형성에 걸리는 시간에 대해 중요한 요 인이 된다. 본 논문에서는 초기 클러스터 수 와 위치 설정을 하 는 단계를 시술 전 계획 단계라 지정하였다. 일반적으로 수 술 로봇 시스템은 시술 전 단계에서 환자의 영상 및 다양한 정보의 분석을 통해 환자의 적합한 상태에 따라 적합한 수 술 계획을 수립하게 되는데, 수술 계획 단계에 주어진 일정 량의 영상 데이터를 토대로 엘보우 기법(elbow method)을 통해 영상 데이터들이 해 있는 군집의 개수와 군집의 중심
점을 결정하였다. 본 논문에서 서술하는 엘보우 기법은 군 집 내 오차제곱합(Sum of Squares Error, SSE)의 값이 최 소가 되도록 클러스터의 중심을 결정해 나가는 방법이다.
SSE 를 수학적으로 나타내어 보면, K는 클러스터 수, x는 샘플 데이터, c i 는 i번째 군집의 중심 데이터로써,
식 (1)
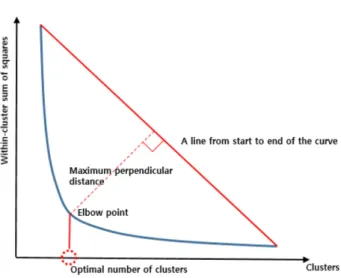
으로 식 (1)처럼 표현 할 수 있다. 본 논문에서 엘보우 기 법 적용에 대해 예시를 들어보면, 만약 군집의 개수를 2로 두고 계산한 SSE 값과 군집의 개수를 4로 두고 계산한 SSE 값을 비교 분석 하였을 때, 군집의 개수를 4로 두고 계산한 SSE 이 값이 더 적다면, 2개의 군집보다 4개의 군집이 더 적합하다는 결론이 나오게 된다. 이런 식으로 군집의 개수를 늘 려가면서 계산한 SSE에 대해 그림 5와 같이 그래프로 나 타내어 보면 SSE의 값이 점점 줄어 들다가 어느 순간 줄 어드는 비율이 급격하게 작아지는 부분이 발생하는데, 그 부 분이 구하려는 최적의 군집 개수가 된다. 그림 6은 감소세 가 정체되는 지점을 찾는 방법을 보여 주고 있다. 처음과 마 지막 데이터 간의 직선에서 수직 거리를 측정하여 거리가 가장 큰 값을 가진 군집 수를 최적의 군집 수로 결정하였다. 시 술 전 단계에서는 군집 결정을 위한 알고리즘을 적용하기 전, 전 처리 단계로써 배경을 제거 한 후(그림 7(a)) 알고리
SSE dist x c ( , i ) 2
x c ∑ ∈
ii 1 =
∑ K
=
그림 5. 엘보우 방법을 이용한 군집 수 결정 방법.
Fig. 5. Elbow method to determine the optimal number of clusters.
109 즘을 적용한다. 알고리즘 적용 후, 결과 화면은 그림 7(b)에
서 확인 할 수 있다.
4. 뇌 종양 검출 알고리즘
이번 장에서는 수술 진행 단계에서 들어오는 MRI에서 실 시간으로 종양을 분할하는 방법에 대해 알아보기로 한다. 본
단계에서는 개발된 알고리즘은 군집 분석에서의 군집과 입 력된 영상 데이터간의 유사성 계산을 통해 실시간으로 주어 지는 영상데이터에서 종양을 분할하는 역할을 하고 있다. 수 술 중에 실시간으로 새롭게 들어오는 영상 데이터가 속해 있는 군집은 시술 전 계획 단계에서 구해진 군집 개수와 중 심점 정보를 가지고 결정 짓게 된다. 영상 데이터와 군집간 의 거리 값이 작을수록 서로 유사성이 높다는 것을 의미한 다. 그림 7는 새로운 데이터가 유클리드 거리 함수를 이용 하여 자신의 군집을 결정하는 방법을 설명하고 있다.
본 논문에서 이용한 유클리드 거리 d(x, y)는 식 (2)와 같 이 정의된다.
식 (2)
여기서 x, y는 영상 내의 픽셀 좌표를 나타낸다.
본 논문에서는 각각의 영상 데이터와 각 군집간의 중심점 과의 거리를 계산하여, 유사성을 측정하는 것으로 영상 데 이터를 군집화 하였다. 여기서의 유사성은 유클리드 거리 값 이 작아 진다는 것을 의미한다. 현재 영상 데이터와 각 군 집간의 거리를 계산하여 가장 작은 거리를 가진 군집이 영 상 데이터가 속해 있는 군집이라 판단하게 된다. 그림 8에
d x y ( ) , ( x i – y i ) 2
i 1 =
∑ n
=
그림 6. 엘보우 방법을 이용한 군집 수 결정 방법.Fig. 6. Elbow method to determine the optimal number of clusters.
그림 7. (a) 배경 제거 후 MRI 영상, (b) 군집 결정 알고리즘 적용 후 군집 별로 화면 출력.
Fig. 7. (a) Background subtraction (b) Result image.
그림 8. 유클리드 거리 함수를 이용한 종양 군집 검출 방법.
Fig. 8. Euclidean distance function for brain tumor detection.
110
서 보듯이, 이진화 작업을 통해 종양이 속해 있는 군집만 하 얗게 표현하여 출력하게 된다.
5. 평가 측정 방법
본 연구에서는 정량적 평가의 측정 방법으로 영상 분할 평가에 쓰이는 대표적인 지표인, 정확도(Accuracy), 정밀도 (Precision), 재현율(Recall), 다이스 계수(DICE Similarity Coefficient, DICE) 를 이용 하였다[25].
본 논문에서 정확도의 정의는 제안한 뇌 종양 분할 방법과 일반적으로 쓰이는 이진화 방법을 이용해 종양을 분할한 방 법들이 실측 자료(Ground truth)와 얼마나 가까운 지를 나 타내는 기준으로써 정의 되었으며, 식 (3)으로 아래와 같이 나타낸다.
Accuracy = 식 (3)
으로 정의 된다. 여기서 TP은 실제 와 비교해서 예측이 참인 값(True Positive), TN은 실제와 비교해서 예측이 거짓인 값(True Negative), FN은 실제가 거짓인데 예측도 거짓인 값(False Negative), FP은 실제는 거짓인데 참으로 예측된 값(False Positive)을 뜻한다.
정밀도는 예측된 값 중에 실제로 참인 값의 비율로써 식 (4)처럼
Precision = 식 (4)
으로 정의 된다
식 (5)의 검출율은 참으로 예측한 값에서 실제 참이 맞는
비율이 얼마인지를 뜻하는 것으로써
Recall = 식 (5)
으로 정의 된다
다이스 계수는 본 논문에서 제시된 실측 자료 와 제안된 검출 방법 사이의 결과를 직접 비교하여 그 유사도를 나타 내는 것으로 정의는 식 (6)과 같다.
DICE = 식 (6)
III. 연구 결과
1. 시각적 평가
본 논문에서는 BRATS 2015에서 제공한 데이터 중에 30 명의 실험용 FLAIR 영상 데이터를 사용하였으며, 전체 뇌 부분에 대하여 각 복셀을 대상으로 실험을 수행 하였다. 제안 방법을 통한 뇌종양 검출에 대한 시각적 및 정성적 평가를 위해 딥 러닝을 이용한CNN 방법, Snakes로 알려진 능동 윤곽 모델(Active contour models), 그리고 수동 분할인 이 진화 방법과 ground truth와의 비교 분석을 통해 평가를 실시하였다. 여기서 사용된 CNN 방법은 의료 영상 분석을 위해 개발된 NiftyNet[26]을 이용하여 학습된 모델로 평가를 진행하였다. 그림 9에서 볼 수 있듯이, 전체적으로 제안된 방법과 CNN을 적용한 방법은 실측 자료와 비교 했을 때 큰 차이를 보여 주고 있지 않고 있다는 걸 알 수 있다. 종 양의 위치 및 크기 부분에서는 미미한 차이점만 보여 주고 있다는 것을 비교 그림에서 알 수가 있다. 수동 분할과 Snakes TP TN +
TP FN FP TN + + + ---
TP TP FP + ---
TP TP FN + ---
2TP FP FN + 2TP + ---
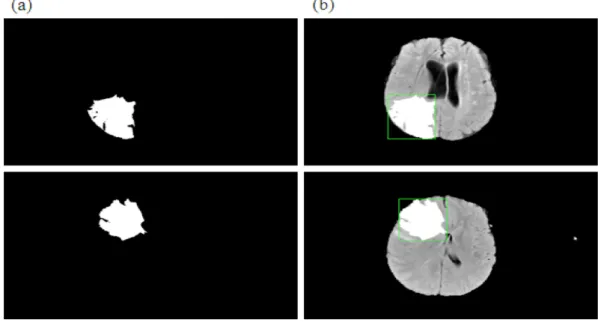
그림 9. 유클리드 거리 함수를 이용한 군집 결정 결과 (a). 종양 검출 binary 작업, (b) 최종 출력 화면.
Fig. 9. Brain MRI outputs (a) Binary images, (b) Tumor segmentation images.
111 을 적용한 방법과의 비교를 통해서 제안된 방법의 이 좀 더
우수하다는 것을 알 수가 있는데, 수동 분할과 Snakes인 경우 위치는 실제 종양의 하고 일치 하지만 윤곽 부분이나 크기 부분에서 제안된 방법과 뚜렷한 차이를 보여 준다. 전반적 으로 제안된 방법이 이진화 방법과 Snakes 보다 우수하게 나 타나고 있는 것을 알 수 있다. 그러나 제안된 종양 검출 방 법도 전체적인 종양 부위는 양호하게 분할 하였으나, 전두 엽 및 후두엽 부근의 백진 부위에 그룹화해야 할 일부 화소를 놓치고 있다. 이러한 결과는 제안된 방법이 CNN 보다 검
출력 평가에서 떨어지는 이유라고 볼 수 있다.
2. 정량적 분석
시각적인 분석으로는 정량적으로 종양 검출 능력이 얼마 나 되는지를 정확히 알 수 없기 때문에 이를 판단하기 위해 정량적 분석 방법을 적용하였다.
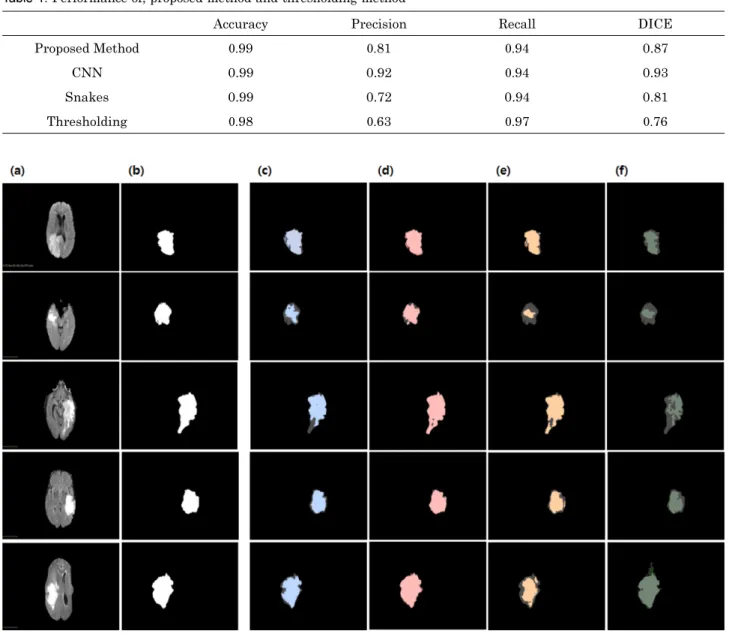
표 1의 정량적 분석 결과는 30개의 데이터에 대한 각 지표 별 평균 값을 비교 한 것으로, 제안 방법 과 비교 방법들의 정확도와 재현율은 각각 0.98, 0.90 이상으로 4가지 방법들
표 1. 정량적 분석 결과
Table 1. Performance of; proposed method and thresholding method
Accuracy Precision Recall DICE
Proposed Method 0.99 0.81 0.94 0.87
CNN 0.99 0.92 0.94 0.93
Snakes 0.99 0.72 0.94 0.81
Thresholding 0.98 0.63 0.97 0.76
그림 10. 뇌 종양 검출 결과 분석을 위한 Ground truth, 와4가지 방법과 비교. (a) MRI 입력 영상 (b) Ground truth (c) Ground truth 와 제안된 방법 간의 비교 (d) Ground truth 와 CNN 간의 비교 e) Ground truth와 snakes 간의 비교 (f) Ground truth와 이진화 간의 비교.
Fig. 10. Visual comparison between ground truth, proposed method, and thresholding method. (a)Examples of MRI images (b) Ground truth (c) Results from a slice comparing the ground truth and the proposed method (d) Results from a slice comparing the ground truth and the CNN (e) Results from a slice comparing the ground truth and the Snakes (f) Results from a slice comparing the ground truth and the thresholding method.
112
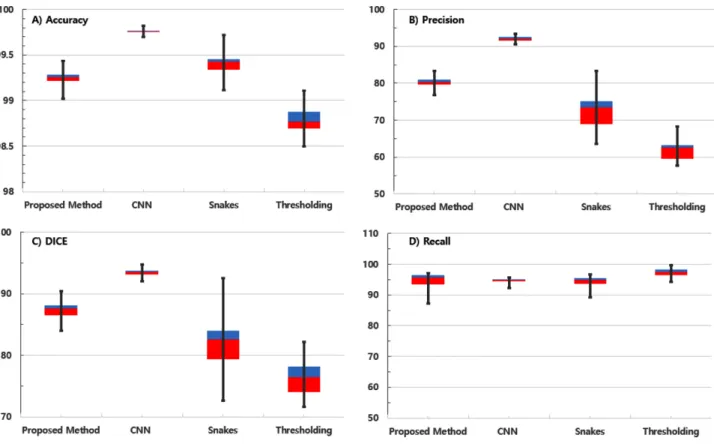
간의 큰 차이가 없지만, 정밀도와 다이스 지표에서는 명확 한 차이를 보여 준다. 정밀도는 CNN 방법이 0.9 이상으로 가장 높은 지표 값을 보여 주었으며, 제안된 방법이 0.8 이 상, Snakes와 이진화 방법은 0.8 이하에 그치고 있다. 특 히, 다이스 지표는 영상 분할(segmentation) 평가에서 유 사도를 측정 할 때 대표적으로 활용 되는 것으로, CNN 방 법은 0.93, 제안된 방법은 0.87인에 반해 나머지 두 방법은 각각 0.81, 0.76에 그치고 있다. 또한, 그림 11의 상자 수 염 그래프를 통해, 각 항목별로 각각의 방법들의 세부적인 수치를 비교해 놓았다. 상자 수염 그래프는 30개의 데이터 에 대해 제안된 알고리즘의 성능 특성을 잘 반영하여 보여 주는 그래프로써, 성능 지표에 대해 특성 분포를 분석하여 보여준다. 데이터 마다 균일한 성능이 나오는지를 평가하기 위해 상자 수염 그래프를 지표 별로 작성하였다. 본 논문에서 제안된 방법과 CNN방법은 Snakes 와 이진화 방법보다 중 앙값이 박스의 중심에 있으며 높낮이가 좁다는 사실에서 데 이터의 분포가 일정하다는 것을 알 수 있다. 이것은 CNN과 제 안된 방법의 성능이 데이터에 상관없이 성능이 일정하게 나 온다는 것을 알려 주는 부분이다. 특히 DICE 지표에 대한 박스 수염 그래프는 CNN 과 제안된 방법이 다른 두 방법과 큰 차이를 보여 주고 있다. 이러한 차이는 특정한 데이터에서 종 양 부위와 정상 부위 간의 인식을 위한 영상에서의 실제 종양
을 정확하게 검출 하지 못했다는 것을 뜻한다. 표 1과 그림 11 에서 보여준 결과 값에서는 CNN 방법이 제안된 방법보다 4 가지 지표에서 전반적으로 우수한 성능을 보여 주고 있다.
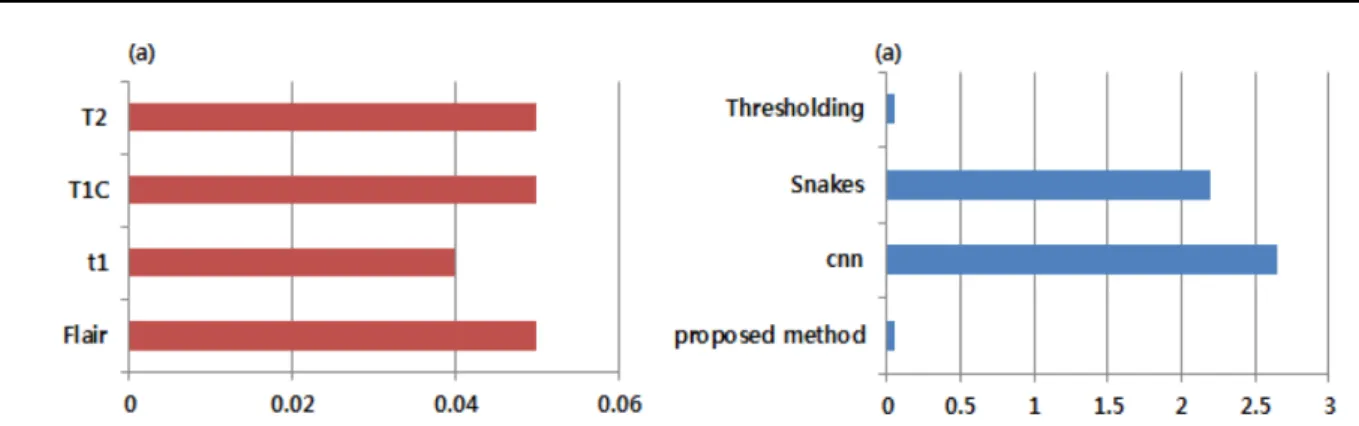
추가적으로, 실시간 종양 검출 시스템의 적합성을 알아 보 기 위해, 처리 시간(Computation time)을 측정하였다. 2.1 에서 소개한 동일한 시스템에서 측정을 실시하였다. 본 연 구에서는 제안된 방법의 이종 데이터 간의 처리 속도를 알 아 보기 위해 다양한 모달리티에서의 데이터 처리 시간을 측정하였으며, 4가지 방법의 처리 시간을 측정 결과하여 결 과를 비교 분석해보았다. 여기서의 처리 시간은 수술 중에 촬영한 1 slice의 MRI 이미지를 전송 받아 처리 후, 뇌 종 양을 화면에 출력하는데 까지 걸리는 시간을 뜻한다. 그림 11(a) 에서 보듯이 제안된 알고리즘을 적용한 이종 데이터에 서의 처리 시간은 0.04 sec에서 0.05 sec로써 데이터간의 차 이가 있어도 일정하게 처리 속도가 유지 되는 것을 알 수 있다. 그림 11(b)는 4가지 방법에서 처리 속도를 측정 하였 는데, 제안된 방법과 수동 분할 방법은 0.05 sec이며, CNN 과 Snakes 방법은 2 sec를 넘기는 처리 속도를 보여 주고 있다. 검출 능력에 대한 지표에서는 CNN방법이 다른 3가 지의 방법보다 우수한 성능을 보여 주었지만, 실시간 처리 시스템에서 중요한 속도 측면에서는 가장 떨어지는 성능을 보여 주고 있다.
그림 11. 상자 수염 그래프를 이용한 정량적 분석 결과.
Fig. 11. Performance comparison of proposed method and thresholding method.
113
IV. 고 찰
뇌 종양 수술 시에 정확한 종양 조직의 위치 및 크기를 판단 하여 실시간으로 수술 보조 로봇에게 종양의 경로 및 목표 정보를 전달한다면, 기존의 수술 도구로는 의료진이 접 근하기 어려운 영역에 수술 보조 로봇이 접근하여 의사의 양손을 대신하여 수술을 진행할 수가 있게 된다. 이에 따라 정확한 뇌 종양 영역을 검출해서 의사 및 로봇에 전달해 주는 것이 수술 보조 로봇을 적용한 뇌수술에서 중요한 부분이며, 만 약 종양 부위를 잘못 판단하여 수술을 진행 한다면, 환자에 게 치명적인 결과를 줄 수 있을 수도 있기 때문이다[2]. 이 러한 이유로 인해 본 논문에서는 실시간 뇌종양 검출을 위 한 개량된 K-means 알고리즘을 사용하여 MRI에서 뇌종 양을 자동 분할하는 방법을 제한하였으며, BRATS 2015에 서 공개한 데이터를 기반으로 제안 방법의 구현 가능성에 대해서 검증하였다. 기존의 K-means 방식의 알고리즘은 여러 가지 장점에도 불구하고 실제 의료 영상 시스템 적용되기에 는 제약이 있었다. 특히, 실시간 클러스터 개수를 정하는 부 분은 K-means 방식이 가지고 있는 가장 큰 문제점 중에 하 나였다. 본 연구에서는 이런 문제점을 고려하여 실시간 영 상 처리 시스템에 적합한 처리 시간과 정확성을 동시에 확 보하기 위해 시스템에 특성에 맞추어 k-means 방식을 수 정하여 새로운 방법을 제시 하였다. MRI영상 유도 수술 보 조 로봇 시스템에 적용하기 위해 본 논문에서 제안된 영상 처리 시스템은 두 단계로 나누어져 개발되었다. 수술 전 준 비 단계에 엘보우 방법을 이용하여 최적의 클러스터 수를 정한 후, 수술 진행 중에 전송되는 영상에 수술 준비 단계 에서 전송 받은 클러스터 수 및 클러스터 중심 값을 유클리 드 거리 함수에 적용하여, 뇌 종양의 위치 및 크기를 화면 에 표시하게 된다. 그림 8, 9에서 보듯이 시각적 및 정량적 평가 부분에서 CNN이 다른 방법과 비교해서 다이스 지표 를 비롯한 5가지 평가지표에서 높은 성능을 낸다는 것을 볼 수 있다. 각각의 방법들은 재현율 및 정확도에서는 비슷한
값을 보여 주고 있지만, 정확도 및 다이스 지표에서는 CNN 이 가장 높은 성능을 보여 주고 있다. 정확도가 모든 지표 들이 높게 나온 것은 전송되는 MRI 에서 배경이 실제 검출 종 양 보다 차지 하는 부분이 많다 보니 TN에 대한 값이 높아 정 확도 와 재현율이 높게 나오게 되었으며, 실제 종양 검출 능 력을 알 수 있는 TP 에 대한 지표인 정밀도 와 다이스 계 수에서는 4가지 방법들이 명확한 차이를 보여 주고 있다.
TP 에 대해서는 CNN이 높은 성능을 보여 주고 있지만, 속 도를 비교한 부분에서는 그림 10에서 보듯이 CNN 보다 제 안된 방법이 우수하다는 것을 보여 주고 있다. 본 연구는 앞 에서 소개한 수술 보조 로봇 시스템에 적용할 실시간 종양 검출 알고리즘 개발이 목표로 써, 우수한 검출 능력만큼 처 리 속도도 중요한 부분이다. 적합한 영상 처리 알고리즘 개 발은 검출 능력과 처리 속도를 모두 만족하는 것이 필요로 하다.
결과적으로, 우리는 연구 과정에서 나온 결과를 종합하여, 제 안된 방법이 검출 능력과 처리 속도를 종합하였을 때 더 적 합하다고 판단 하였다. 제안된 방법의 검출 능력은 CNN 보 다 다이스 계수 기준7% 정도 성능이 떨어지지만, 속도 측면에 서는 CNN과 비교하여 실시간 적용에 적합하다(그림 12).
또한, 수술 보조 로봇 시스템 구성 측면에서CNN 같은 경 우에는 방대한 영상 데이터를 수집하여 학습을 통해 수술 중 검출을 위한 모델을 만들어야 하므로, 준비 기간과 데이터 수집에 문제가 발생하며 그에 따라 시스템 구성의 추가를 고려해야 한다. 하지만, 본 연구에서 제안된 방법은 비교적 간단한 알고리즘 이용하여 종양을 검출하기 때문에 복잡한 준비 단계나 수술 중에 종양 부분을 검출 하기 위해 기다리 지 않아도 된다는 장점이 있다. 추가로, 시술 계획 시에 촬 영한 MRI로 실시간 종양 검출에 필요한 파라미터를 구할 수가 있다. 따라서 본 연구의 결과에 의해 수술 보조 로봇 시스템에 적합한 영상 처리 알고리즘은 제안된 방법이라고 판단 되었다. 하지만, 정량적 평가에서 보여 준 특정한 데이 터에서의 큰 편차는 분명 개선되어야 할 부분이다. 일정한
그림 12. 뇌 종양 검출 시간 비교 (a) 데이터 타입 별 제안된 방법 검출 시간, (b) 소개된 4가지 방법의 검출 시간.Fig. 12. Computation time for MRI tumor detected images (a) 4 type MRI data, (b) 4 methods.
114
지표 성능 유지와 전체 지표의 성능을 향상을 동시에 하기 위해서는 후처리 또는 파라미터의 상세 조정 등을 통해 완 화 될 수 있는지에 대해 계속적인 연구를 수행 할 필요가 있다. 본 연구에서 소개된 방법을 개선하기 위해, 후속으로 2 가지 대한 연구를 진행 할 예정이다. 첫 번째는 처리 속도 를 향상한 개선된 CNN방법을 적용 하는 부분과 두 번째는 처리 속도를 유지하면서 제안된 방법의 검출 능력을 향상 시킬 지에 대한 두가지 방식의 후속 연구를 통해 얻어진 결 과로 비교 분석 할 것이다.
이와 더불어, 향후 개선된 알고리즘을 적용한 수술 보조 로봇 시스템을 확장하여 좀 더 다양한 뇌종양 MRI와 다른 부위에 기반한 확대 연구들이 이루어 지도록 할 것을 목표 로 하고 있으며, 현재 개발 중인 수술 보조 로봇 시스템에 실제 제안된 알고리즘을 탑재하여 성능 평가를 통한 연구 결과를 비교하여 시스템 고도화 작업에 활용할 계획이다.
참고문헌
[1] A. R. Asthagiri, N. Pouratian, J. Sherman, G. Ahmed, and M.
E. Shaffrey, “Advances in brain tumor surgery,” Neurol Clin., vol. 25, pp. 975-1003, 2017.
[2] J. A. Smith, J. Jivraj, R. Wong, and V. Yang, “30 years of neurosurgical robots: review and trends for manipulators and associated navigational systems,” Annals of Biomedical Engineering, vol. 44, no. 4, pp. 836-846, 2016.
[3] G. J. S. Litjens, T. Kooi, B. E. Benjnordi, A. A. A. Setio, F.
Ciompi, M. Ghafoorian, J. A. W. M. van der Laak, B. v. Gin- neken, and C. I. Sánchez, “A survey on deep learning in medical image analysis,” Medical Image Analysis, vol. 42, pp. 60-88, 2017.
[4] S. Hussain, S. M. Anwar, and M. Majid, “Segmentation of glioma tumors in brain using deep convolutional neural net- work,” Neurocomputing, vol. 282, pp. 248-261, 2018.
[5] X. Zhao, Y. Wu, G. Song, Z. Li, Y. Zhang, and Y. Fan, “A deep learning model integrating FCNNs and CRFs for brain tumor segmentation,” Medical Image Analysis, vol. 43, pp.
98-111, 2018.
[6] M. Havaei, A. Davy, D. Warde-Farley, A. Biard, A. Courvi- lle, Y. Bengio, C. Pal, P. Jodoin, and H. Larochelle, “Brain tumor segmentation with deep neural networks,” Medical Image Analysis, vol. 35, pp. 18-31, 2017.
[7] S. Pereira, A. Pinto, V. Alves, and C. A. Silva, “Brain tumor segmentation using convolutional neural networks in mri images,” IEEE Transactions on Medical Imaging, vol. 35, no. 5, pp. 1240-1251, 2016.
[8] T. Kalaiselvi and P. Nagaraja, “An automatic segmentation of brain tumor from mri scans through wavelet transforma- tions,” International Journal of Image, Graphics and Signal Processing, vol. 8, no. 11, pp. 59-65, 2016.
[9] S. Gupta, M. Agrawal, and S. Kumar, “An enhanced brain tumor area detection and segmentation techniques in mri medical images using modified K-means algorithm,” Inter- national Journal of Computer Applications, vol. 143, no. 13, pp. 46-50, 2016.
[10] Uma-E-Hani, S. Naz, and I. A. Hameed, “Automated tech- niques for brain tumor segmentation and detection: A review study,” in Proc. 2017 International Conference on Behavioral, Economic, Socio-cultural Computing (BESC), Krakow, 2017, pp. 1-6.
[11] U. Ilhan and A. Ilhan, “Brain tumor segmentation based on a new threshold approach,” Procedia Computer Science, vol.
120, pp. 580-587, 2017.
[12] Virupakshappa and B. Amarapur, “Cognition-based MRI brain tumor segmentation technique using modified level set method,” Cognition, Technology & Work, Feb. 2018.
[13] N. B. Bahadure, A. K. Ray, and H. P. Thethi, “Image analysis for MRI based brain tumor detection and feature extraction using biologically inspired BWT and SVM,” International Journal of Biomedical Imaging, vol. 2017, pp. 1-12, 2017.
[14] Y. K. Dubey, M. M. Mushrif, and K. Mitra, “Segmentation of brain MR images using rough set based intuitionistic fuzzy clustering,” Biocybernetics and Biomedical Engineering, vol. 36, no. 2, pp. 413-426, 2016.
[15] S. Banerjee, S. Mitra, and B. Uma Shankar, “Single seed delineation of brain tumor using multi-thresholding,” Infor- mation Sciences, vol. 330, pp. 88-103, 2016.
[16] D. Kharbanda and G. K. Verma, “Multi-level 3D wavelet analysis: application to brain tumor classification,” In Proc.
2016 International Conference on Micro-Electronics and Telecommunication Engineering (ICMETE), GHAZIABAD, India, 2016, pp. 379-384.
[17] T. M. Devi, G. Ramani, and S. X. Arockiaraj, “MR brain tumor classification and segmentation via wavelets,” In Proc.
2018 International Conference on Wireless Communications, Signal Processing and Networking (WiSPNET), Chennai, India, 2018, pp. 1-4.
[18] B. Devkota, A. Alsadoon, P. W. C. Prasad, A. K. Singh, and A. Elchouemi, “Image segmentation for early stage brain tumor detection using mathematical morphological reconstruction,”
Procedia Computer Science, vol. 125, pp. 115-123, 2018.
[19] Y. Ai, F. Miao, Q. Hu, and W. Li, “Multi-feature guided brain tumor segmentation based on magnetic resonance images,”
IEICE Transactions on Information and Systems, vol. E98.D, no. 12, pp. 2250-2256, 2015.
[20] J. G. and H. Inbarani H., “Hybrid tolerance rough set-firefly based supervised feature selection for MRI brain tumor image classification,” Applied Soft Computing, vol. 46, pp. 639- 651, 2016.
[21] B. H. Menze et al., “The multimodal brain tumor image seg- mentation benchmark (BRATS),” IEEE Transactions on Medical Imaging, vol. 34, no. 10, pp. 1993-2024, 2015.
[22] S. J. Nanda, I. Gulati, R. Chauhan, R. Modi, and U. Dhaked,
“A K-means-galactic swarm optimization-based clustering algorithm with otsu’s entropy for brain tumor detection,”
Applied Artificial Intelligence, pp. 1-19, 2018.
[23] K. Thiruvenkadam, K. Nagarajan, and S. Padmannaban,
“Automatic brain tissues segmentation based on self initial- izing K-means clustering technique,” International Journal of Intelligent Systems and Applications, vol. 9, no. 11, pp. 52- 61, 2017.
[24] C. Saha and M. F. Hossain, “MRI brain tumor images clas- sification using K-means clustering, NSCT and SVM,” in 2017 4th IEEE Uttar Pradesh Section International Conference on Electrical, Computer and Electronics (UPCON), Mathura,
115
2017, pp. 329-333.[25] M. Angulakshmi and G. G. Lakshmi Priya, “Automated brain tumour segmentation techniques- A review,” International Journal of Imaging Systems and Technology, vol. 27, no. 1, pp. 66-77, 2017.
[26] E. Gibson, W. Li, C. Sudre, L. Fidon, D. I. Shakir, G. Wang,
Z. Eaton-Rosen R. Gray, T. Doel, Y. Hu, T. Whyntie, P. Nachev, M. Modat, D. C. Barratt, S. Ourselin, M. J. Cardoso, and T.
Vercauteren, “NiftyNet: a deep-learning platform for medical imaging,” Computer Methods and Programs in Biomedicine, vol. 158, pp. 113-122, 2018.