대한소화기학회지 2007;49:209-224
위축 위염과 장형화생에서 마이크로어레이를 이용한 유전자 발현
건국대학교 의과대학 외과학교실, 내과학교실
*
, 가정의학교실†, 병리학교실‡, 해부학교실§, 의료생명대학 생명공학과∥,부산성모병원 소화기내과¶
김경래·오수연·박웅채·왕준호*·이재동*·권혁중
†·김상윤
‡·박승화
§·최동국
∥·김찬길
∥·최성호
¶Gene Expression Profiling using Oligonucleotide Microarray in Atrophic Gastritis and Intestinal Metaplasia
Kyong Rae Kim, M.D., Soo Youn Oh, M.D., Ung Chae Park, M.D., J oon Ho Wang, M.D.
*
, J ae Dong L ee, M.D.*
, Hyuk J ung Kweon, M.D.†, Sang Yoon Kim, M.D.‡, Seung Hwa Park,Ph.D.§, Dong Kug Choi, Ph.D.∥, Chan Gil Kim, Ph.D.∥, and Seong Ho Choi, M.D.¶
Departments of General Surgery, Internal Medicine
*
, Family Medicine†, Pathology‡, and Anatomy§, Konkuk University College of Medicine; Department of Biotechnology, College of Biomedical and Health Science∥, Chungju;Department of Internal Medicine, Busan St. Mary’s Hospital¶, Busan, Korea
Background/Aims: The atrophic gastritis with intestinal metaplasia of gastric mucosa has been considered to be the major factor of carcinogenesis in the stomach. However, the key molecules are still poorly understood. To elucidate the molecular genetic basis, we report the results of our initial microarray data to analyze the genome pattern in patients with atrophic gastritis and intestinal metaplasia of the stomach. Methods: We used oligonu- cleotide microarray technique to evaluate the gene expression profiles in atrophic gastritis with intestinal metaplasia, in comparison with those of normal mucosa. For the identification of differentially expressed genes, Significance Analysis of Microarrays (SAM) package method was used. The results were analyzed using global normalization, intensity dependent normalization, and box plot normalization. Results: Eight genes including FABP, REG, OR6C1, MEP1, SLC6A1, SI, Mucin 1, and RAB23 in mucosa of atrophic gastritis and intestinal metaplasia were up-regulated by more than 10 times as compared with normal gastric mucosa. Only one gene, LOC44119 was down-regulated by more than 10 times of the expression as compared with normal gastric mucosa. In respect to the expression of known genes related to gastric carcinogenesis, 8 genes including FN1, SRMS, TP53, TP53IMP2, TP53I3, FGFR4, TGFB1, and TGFA showed up- and down-regulations more than 2 folds in expression pattern. Conclusions: We could identify a total genome pattern in patient with atrophic gastritis and intestinal metaplasia using oligonucleotide microarray. We believe that the current results will serve as a fundamental bioinformative basis for clinical applications in diagnosis and treatment of gastric cancer and precancerous lesion in the future. (Korean J Gastroenterol 2007;49:209-224)
Key Words: Gastric cancer; Atrophic gastritis; Intestinal metaplasia; Oligonucleotide microarray; Gene expression
Correspondence to: Kyong Rae Kim, M.D.
Department of General Surgery, Konkuk University School of Medicine, 620-5, Kyohyun-dong, Chungju 380-704, Korea Tel: +82-43-840-8826, 8240, Fax: +82-43-848-0865 E-mail: kkongr@kku.ac.kr
접수: 2006년 10월 23일, 승인: 2007년 2월 17일
연락처: 김경래, 380-704, 충북 충주시 교현 2동 620-5번지 건국대학교의료원 충주병원 외과
Tel: (043) 840-8826, 8240, Fax: (043) 848-0865 E-mail: kkongr@kku.ac.kr
*
이 논문은 2004년도 건국대학교 학술진흥연구비 지원에 의한논문임.
210 대한소화기학회지: 제49권 제4호, 2007
서 론
위암 발병의 원인이나 기전은 아직까지 정확하게 밝혀지 지 않았으나, 위암 발병 요인과 방어 요인에 대한 많은 연구 가 진행되어 왔다. 발병 요인 중에서 만성 위축 위염은 암으 로 발전되는 위험도가 높은 일종의 암전구 병변으로 위암 발병까지 16-24년의 장기간이 필요하다.1 위점막의 장형화 생은 위암종 중에서 장형 위암종 전 단계 병변으로 조직, 조 직화학, 전자현미경, 그리고 효소 특징에 의해 여러 아형으 로 분류 연구되고 있다.2-4 그러나 정상 점막과 장형화생의 유전자 변화와 차이점에 대한 연구는 아직까지 미미하다.
이번 연구에서는 위암 발생의 중요 단계인 장형화생을 보 이는 만성 위축 위염과 정상 점막과의 유전자 비교를 기존 의 southern blot, northern blot, DNA sequencing 같은 유전자 정보를 개별적으로 확인하던 방법에서 진일보하여, 적은 양 의 시료로 대량의 정보를 얻을 수 있는 올리고뉴클레오티드 마이크로어레이(oligonucleotide microarray) 방법을 이용하여 조사하였다. 국내에서는 아직까지 위암의 중요 발생 요인인 장형화생을 보이는 만성 위축 위염의 올리고뉴클레오티드 마이크로어레이 실험 결과에 대한 보고가 없다. 이에 저자 들은 올리고뉴클레오티드 마이크로어레이를 이용한 대량 유전자 검색으로 다양한 유전자 변화를 확인하고 위암 발생 과의 연관성을 조사하여 임상적으로 활용할 수 있는 기본 자료 축적을 위해 이번 연구를 시도하였다.
대상 및 방법
이번 연구에서는 위내시경 검사를 받았던 환자 중에서 만 성 위축 위염을 의심하여 위 점막의 조직검사를 시행한 후 만성 위축 위염과 장형화생으로 진단받은 24개 검체(정상 점막 및 폴립 조직 12쌍)를 대상으로 하였다. 조직의 절반은 진단을 위한 병리조직 검사에 이용하고, 나머지 절반은 RNA 파손을 막기 위해 RNA laterⓇ (Ambion Inc., Austin, TX)에 담근 후 4oC 이하에서 냉장 보관한 다음날 액체 질소 용기에 급속 냉동한 후 실험실로 운반하였다. Agilent 2100 Bioanalyzer (Agilent Technologies, Palo Alto, CA, USA) 분석 을 통하여 RNA 상태가 양호하고 병리 조직학적으로 만성 위축 위염과 장평화생으로 확인한 3명의 환자에서 추출한 3 쌍의 검체를 대상으로 마이크로어레이 분석 및 GeneSpring 프로그램을 이용한 클러스터링 분석을 수행하였다. 실험에 사용된 전 RNA 표본에서 대조군으로 정상 점막에서 추출 한 RNA 표본을 사용했으며, RNA 추출 후 정성검사를 시행 하였다. 역전사(reverse transcription) 과정을 통해 fluorescent dCTP가 같이 구성된 cDNA를 합성하였으며 올리고뉴클레 오티드 마이크로어레이 칩에 혼성화(hybridization)를 실시하
였다. 이번 실험에 사용된 올리고뉴클레오티드 마이크로어 레이 칩은 Operon Human Whole 35K Oligo chip (Genocheck, Ansan, Korea)으로 이것은 Operon사에서 보유하고 있는 human Whole Genome Oligo subsets V4.0 중 Exon sequence oligo (31,387개), transcript sequence oligo (3,396개)와 chip의 정성 확인과 정상화를 위하여 대조군(1,877개)이 점적되어 있다.
대조 유전자를 포함하여 총 36,912개의 spot이 포함되어 있 으며 48개의 구획에 769개씩 분산되어 구성되어 있다. 이 중에는 29,134개의 기능이 알려진 유전자와 기능이 밝혀지 지 않은 transcript, EST sequence 등 5,901개가 포함되었다 (Fig. 1).
1. 형광 DNA 표지와 혼성화
전체 RNA는 Trizol 시약(Gibco RBL, Grand island, NY)을 이용하여 추출하였다. 전체 RNA 30μg에 역전사 혼합물 [400 U Superscript RNase H-reverse transcriptase (Gibco RBL, USA), oligo (dT)18, 0.5 mM dATP, dTTP , 0.2 mM dCTP, 0.1 mM Cy3 또는 Cy5로 표지된 dCTP (NEN Life Science, Boston, MA)]을 첨가하여 총 30μL의 형광 표지된 cDNA 탐 색자를 합성하였다. 역전사 반응 후 5 μL의 반응 정지액(0.5 M NaOH/50 mM EDTA)을 넣고, 65oC에서 10분간 처리하였 다. Cy3, Cy5로 형광 표지된 cDNA는 에탄올 침전을 통해 농축한 후 12μL의 혼성화 용액(2X Denhardt’s solution, 4.5%
SDS, 1X SSC, 1 mM EDTA, 0.25 M Na2HPO4, 0.05 mg/mL yeast tRNA)을 이용하여 용해 후 합성된 두 개의 cDNA를 똑같은 양으로 섞어서 95oC에서 2분간 변성시킨 후 45oC에 Fig. 1. Contents of Operon Human Whole 35K oligo chip. This oligonucleotide microarray chip is comprised of 31,387 exon se- quence oligos, 3,396 transcript sequence oligos, and 1,877 control oligos for confirmation of quantity and normalization.
김경래 외 10인. 위축 위염과 장형화생에서 마이크로어레이를 이용한 유전자 발현 211
서 20분간 처리하였다. cDNA 혼합물을 3,000개의 DNA가 집적되어 있는 슬라이드 글라스(GenoCheck, Korea)로 넣은 후, 카버 슬립(22×22 mm)을 덮고 혼합물이 글라스에 잘 분 포되도록 하였다. 슬라이드를 반응 chamber에 넣은 후 62oC 에 14시간 동안 혼성화시켰다. 상온에서 2X SSC, 0.1% SDS 용액을 이용하여 2분간 먼저 세척한 후, 1X SSC 용액에서 3 분간, 그리고, 0.2X SSC 용액에서 2분간 세척하였다.
2. 스캐닝과 이미지 분석
혼성화가 끝난 후 슬라이드의 스캐닝은 GenePix 4000B scanner (Axon, Union City, CA)를 사용하여 수행하였다. 각 spot에서 방사되는 Cy3와 Cy5의 형광정도(fluorescence in- tensity)를 수치화하였다. 스캐너 분석용 소프트웨어(GenePix pro 4.1) 프로그램을 이용해서 전환된 수치 값은 마이크로소
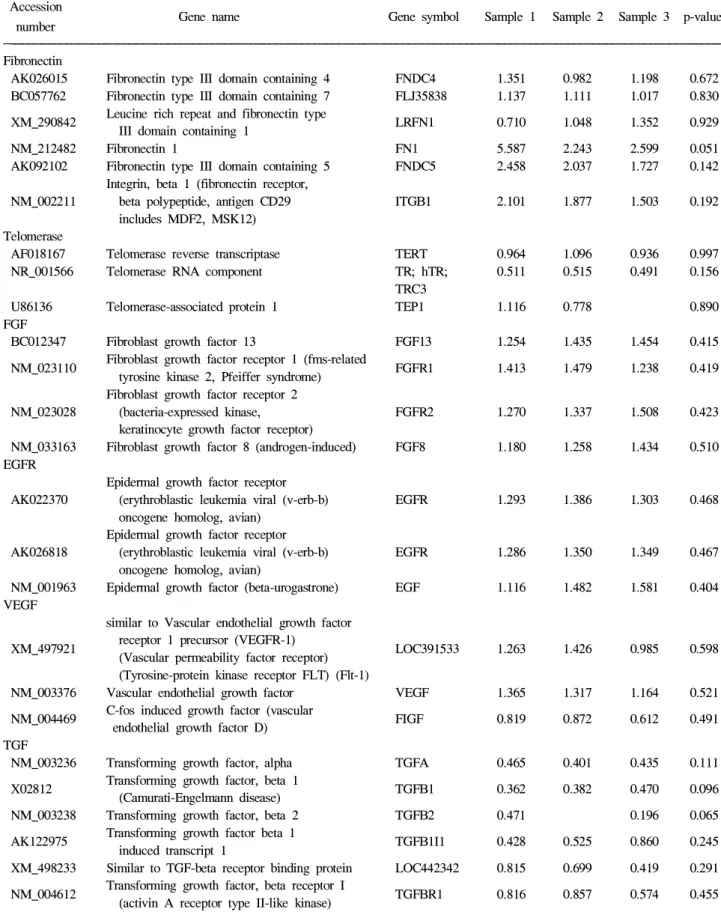
프트 엑셀 프로그램 등을 사용하여 정도기준정상화를 실시 하였다(Fig. 2). 이미지 분석은 GenePix Pro 4.1와 GeneSpring (Silicon genetics, San Jose, CA)의 분석 프로그램를 사용하였 으며 개별 spot의 분석은 GenePix에 내장된 알고리듬을 이 용하여 수행하였다.
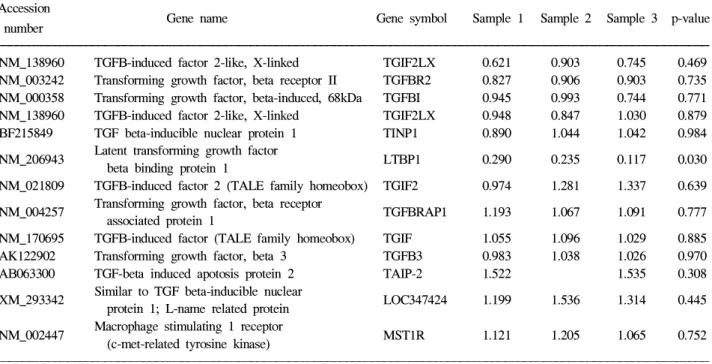
실험 재료와 표시시약의 특성, 실험에서의 변이를 보정하 고 결과의 신뢰성을 위해 정상화를 실시하였다. Global normalization과 정도 의존 정상화 및 각 구역에 따른 box plot normalization도 실시하여 올리고 칩에 사용된 총 48개 pin들이 각각의 특성과 상태에 따른 차이와 혼성화 과정에 서 생길 수 있는 국소적 배경으로 인한 부정확한 정도비 등 의 차이를 줄였다. 얻어진 모든 유전자들의 분포를 histo- gram으로 표시하였으며(Fig. 3), 정상화가 끝난 결과의 분포 도와 비교 검토하여 실험 결과의 정확성을 확인하였다.
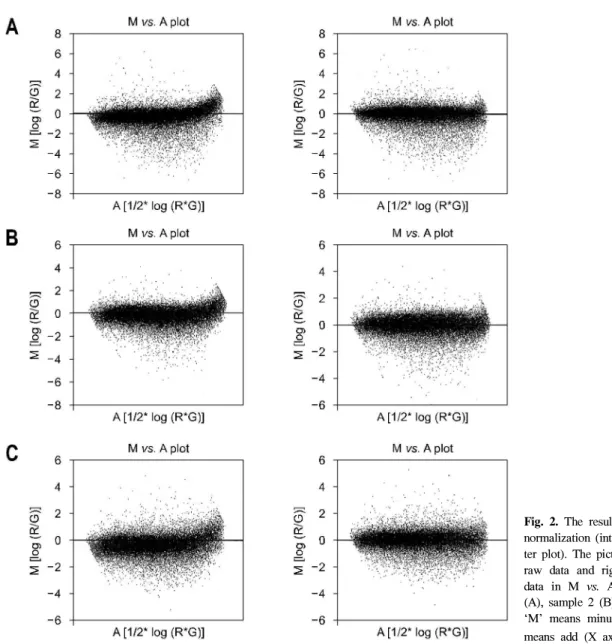
Fig. 2. The results of intensity-based normalization (intensity M vs. A scat- ter plot). The pictures show left-sided raw data and right-sided normalized data in M vs. A plot of sample 1 (A), sample 2 (B), and sample 3 (C).
‘M’ means minus (Y axis) and ‘A’
means add (X axis).
212 The Korean Journal of Gastroenterology: Vol. 49, No. 4, 2007
3. 통계 분석
결과의 통계학적인 정확성을 확인하기 위해 SAM (Stan- ford Univ., Stanford, CA) 프로그램을 이용하였으며 실험 결 과는 distant cutoff가 2.0 배수의 변화를 보일 때 유의하였고
차단 상관 계수는 0.95였다. t-test는 실험군과 대조군 사이의 통계 차이를 결정하기 위해 사용하였으며 다면 비교의 경 우, 유의수준은 Bonferroni 조정법을 따랐다.
결 과
1. 올리고뉴클레오티드 마이크로어레이칩의 발현 양상
첫 번째 표본의 정상점막 표본에 대비한 유전자 발현 변 이에서, 총 3,160개의 유전자들이 마이크로어레이에서 유의 한 수준인 2배 이상 발현 변이를 나타내었다. 그중 CYP- 3A4, CYP4F3, ACADS, SDHB, ALOXE3, P4HB, TXNL5, UQ- CRFS1 등의 에너지 경로에 관여하는 유전자들과 NPHS, TRO, ITGB1, CDH17, DSG2, TSPAN2, CD9 등의 유착 분자 (adhesion molecule)에 관여하는 유전자 등을 포함하여 584개 의 유전자들이 2배 이상의 과발현을 나타내었다. 한편, DLK1, ZNF262, NUMB, IP3, DLX3, TLX3, TLL1 등의 발생 에 관여하는 유전자들과 ITGA6, ITGAV, ITGB6, CDH8, CDH26, ADAM2, ADAM15, DGCR2, DGCR6 등의 유착 분 자에 관여하는 유전자를 포함하여 2,576개의 유전자들이 마 이크로어레이에서 유의한 수준인 2배 이상 저발현을 보였다.
두 번째 표본의 경우, 총 2,404개의 유전자들이 유의한 수 준인 2배 이상 발현 변이를 나타내었다. 그 중에 SENP6, CPA5, CPB2, RPS10, RPL31, RPL26L1, HBS1L, PPIC, TM4SF3, GCNT1, DET1 등의 protein fate에 관여하는 유전 자들을 포함하여 444개의 유전자들이 2배 이상의 과발현을 나타내었다. 또한, FOXN4, FOXD1, EAF1, MAF, MAFG, ELK4, TBP, TBL1Y, SMAD7, ALF 등의 transcription factor activity에 관여하는 유전자를 포함하여 1,960개의 유전자들 이 유의한 수준인 2배 이상의 저발현을 나타내었다.
세 번째 표본의 경우, 유전자 발현 변이에서 총 2,456개의 유전자들이 마이크로어레이에서 유의한 수준인 2배 이상 이상 발현 변이를 나타내었다. 그중에 PPP1CC, SIAT7F, GCNT3, GALNT7, GPI, GK, SI, NEU1, MGAT4B 등의 탄수 화물 대사에 관여하는 유전자들을 포함하여 707개의 유전 자들이 2배 이상의 과발현을 나타내었다. 그 외에 SNX25, ASB3, SHB, SNX22, SNX24, SHANK2, PLCB3, SRMS, PRKCG, INPP5D 등의 intracellular signal cascade에 관여하는 유전자들을 포함하여 1,749개의 유전자들이 저발현을 보였다.
2. 공통 유전자 발현 양상
세 시료에 대한 분석 결과, 상호 상관관계는 0.693, 0.680, 0.698의 correlation coefficient를 나타내어 유사한 발현 양상 을 보였다. 또한 전체 clustered image를 통해 상호 발현양상 을 확인하였다(Fig. 4). 세 실험에서 공통적으로 2배 이상 과 Fig. 3. Normalized data intensity histographs. The left-sided graphs
mean raw data and right-sided mean normalized data in each figure [sample 1 (A), sample 2 (B), and sample 3 (C)].
Kim KR, et al. Gene Expression Profiling using Oligonucleotide Microarray in Atrophic Gastritis and Intestinal Metaplasia 213
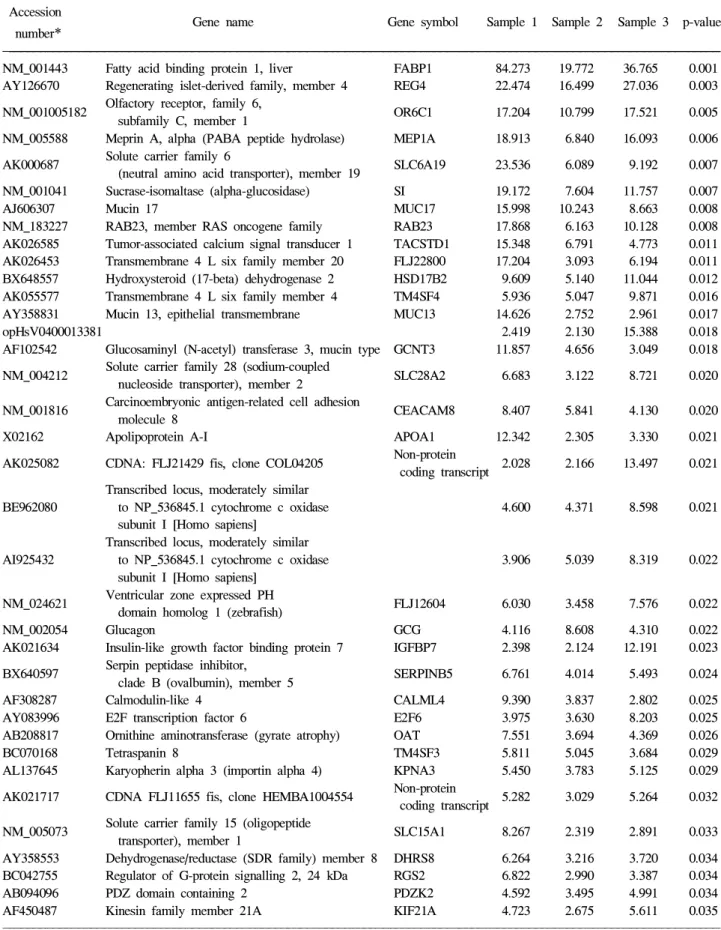
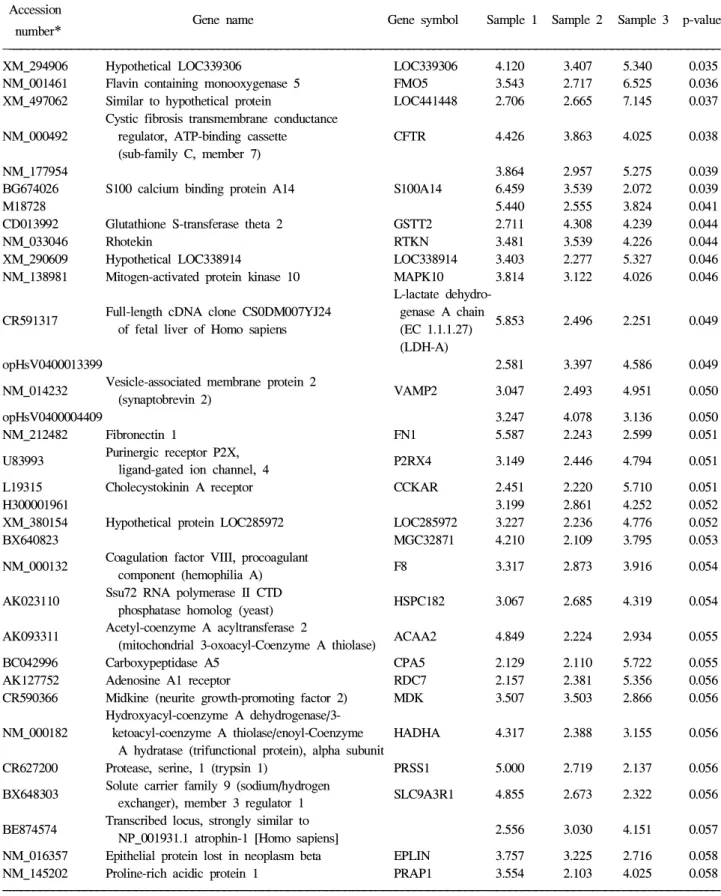
발현을 보인 유전자는 ACAA2, ADAM11, APOA1, CALML4, CCL3, GCG, MAPK10, S100A14를 포함하여 139개였으며 (Table 1), 공통적으로 2배 이상 저발현을 나타낸 유전자는 CCR5, CCR8, CDKL1, CDC42BPG, DEFA4, DEFB119, DDX31, FOXD1, ITGAD, MYO1F를 포함하여 350개였다(저 발현 결과는 Table에서 생략하였음). 세 표본의 공통된 특징 적인 유전자를 SAM을 통하여 확인하였다. 이 중 10배 이상 뚜렷하게 과발현된 유전자는 모두 8개로 FABP (fatty acid binding protein 1, liver), REG (regenerating islet-derived fa- mily, member), OR6C1 (olfactory receptor, family 6, subfamily C, member 1), MEP1 (meprin A, alpha: PABA peptide hydrolase), SLC6A1 (solute carrier family 6: neutral amino acid transporter, member 19), SI (sucrase-isomaltase: alpha-gluco- sidase), mucin 1, RAB23 (RAB23, member RAS oncogene family)였다. 10배 이하로 뚜렷하게 발현이 저하된 유전자는 similar to hypothetical protein FLJ1030 (LOC44119) 하나였다.
3. 위암 발생과 관련된 유전자 발현 양상
장형화생 점막에서 위암 발생과 관련된 대표적인 유전자 변화를 확인하였다(Table 2). 또한 공통적으로 2배 이상의 증감을 보이는 유전자 중 위암 발생과 관련된 FN1, SRMS, TP53, TP53IMP2, TP53I3, FGFR4, TGFB1, TGFA 등 모두 8 개 유전자 발현을 확인하였다(Table 3). 종양 원유전자 중 위암발생과 관련된 c-met는 유의하지 않았으나 전반적으 로 과발현되는 양상을 보였다. 성장억제 인자에서 TGFA, TGFB1, FGF 모두 정상 점막에 비해 오히려 저발현되었으 며 p53의 경우도 tumor protein p53 inducible nuclear protein 2, tumor protein p53와 tumor protein p53 inducible protein 3 모두 저발현을 보였다. Telomerase의 경우 telomerase RNA component 유전자에서만 세 경우 모두 일정하게 저발현되었 다.
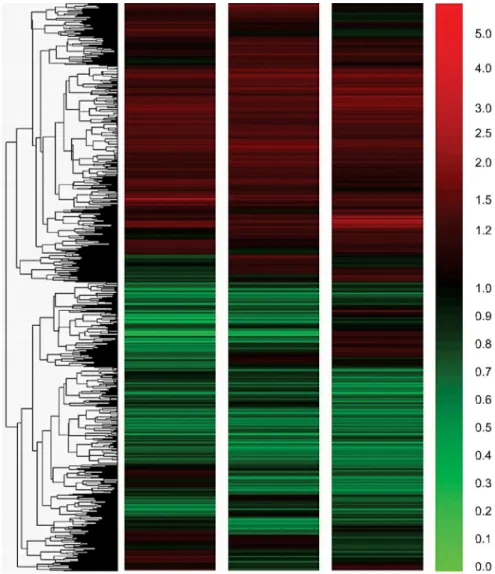
Fig. 4. Profiling of gene expression patterns by hierarchical clustering. Hi- erarchical clustering is an unsuper- vised method for the organization and display of microarray data. Each raw represents a different gene and each column represents a different sample on the microarray. The red and green colors correspond to high (up-regula- ted) and low (down-regulated) expres- sions respectively.
Table 1. Commonly Up-regulated Gene Expression in Gastric Mucosa of Atrophic Gastritis and Intestinal Metaplasia Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
*
NM_001443 Fatty acid binding protein 1, liver FABP1 84.273 19.772 36.765 0.001 AY126670 Regenerating islet-derived family, member 4 REG4 22.474 16.499 27.036 0.003
Olfactory receptor, family 6,
NM_001005182 OR6C1 17.204 10.799 17.521 0.005
subfamily C, member 1
NM_005588 Meprin A, alpha (PABA peptide hydrolase) MEP1A 18.913 6.840 16.093 0.006 Solute carrier family 6
AK000687 SLC6A19 23.536 6.089 9.192 0.007
(neutral amino acid transporter), member 19
NM_001041 Sucrase-isomaltase (alpha-glucosidase) SI 19.172 7.604 11.757 0.007
AJ606307 Mucin 17 MUC17 15.998 10.243 8.663 0.008
NM_183227 RAB23, member RAS oncogene family RAB23 17.868 6.163 10.128 0.008 AK026585 Tumor-associated calcium signal transducer 1 TACSTD1 15.348 6.791 4.773 0.011 AK026453 Transmembrane 4 L six family member 20 FLJ22800 17.204 3.093 6.194 0.011 BX648557 Hydroxysteroid (17-beta) dehydrogenase 2 HSD17B2 9.609 5.140 11.044 0.012 AK055577 Transmembrane 4 L six family member 4 TM4SF4 5.936 5.047 9.871 0.016 AY358831 Mucin 13, epithelial transmembrane MUC13 14.626 2.752 2.961 0.017
opHsV0400013381 2.419 2.130 15.388 0.018
AF102542 Glucosaminyl (N-acetyl) transferase 3, mucin type GCNT3 11.857 4.656 3.049 0.018 Solute carrier family 28 (sodium-coupled
NM_004212 SLC28A2 6.683 3.122 8.721 0.020
nucleoside transporter), member 2
Carcinoembryonic antigen-related cell adhesion
NM_001816 CEACAM8 8.407 5.841 4.130 0.020
molecule 8
X02162 Apolipoprotein A-I APOA1 12.342 2.305 3.330 0.021
Non-protein
AK025082 CDNA: FLJ21429 fis, clone COL04205 2.028 2.166 13.497 0.021 coding transcript
Transcribed locus, moderately similar
BE962080 to NP_536845.1 cytochrome c oxidase 4.600 4.371 8.598 0.021 subunit I [Homo sapiens]
Transcribed locus, moderately similar
AI925432 to NP_536845.1 cytochrome c oxidase 3.906 5.039 8.319 0.022 subunit I [Homo sapiens]
Ventricular zone expressed PH
NM_024621 FLJ12604 6.030 3.458 7.576 0.022
domain homolog 1 (zebrafish)
NM_002054 Glucagon GCG 4.116 8.608 4.310 0.022
AK021634 Insulin-like growth factor binding protein 7 IGFBP7 2.398 2.124 12.191 0.023 Serpin peptidase inhibitor,
BX640597 SERPINB5 6.761 4.014 5.493 0.024
clade B (ovalbumin), member 5
AF308287 Calmodulin-like 4 CALML4 9.390 3.837 2.802 0.025
AY083996 E2F transcription factor 6 E2F6 3.975 3.630 8.203 0.025 AB208817 Ornithine aminotransferase (gyrate atrophy) OAT 7.551 3.694 4.369 0.026
BC070168 Tetraspanin 8 TM4SF3 5.811 5.045 3.684 0.029
AL137645 Karyopherin alpha 3 (importin alpha 4) KPNA3 5.450 3.783 5.125 0.029 Non-protein
AK021717 CDNA FLJ11655 fis, clone HEMBA1004554 5.282 3.029 5.264 0.032 coding transcript
Solute carrier family 15 (oligopeptide
NM_005073 SLC15A1 8.267 2.319 2.891 0.033
transporter), member 1
AY358553 Dehydrogenase/reductase (SDR family) member 8 DHRS8 6.264 3.216 3.720 0.034 BC042755 Regulator of G-protein signalling 2, 24 kDa RGS2 6.822 2.990 3.387 0.034 AB094096 PDZ domain containing 2 PDZK2 4.592 3.495 4.991 0.034 AF450487 Kinesin family member 21A KIF21A 4.723 2.675 5.611 0.035
* Locus link accession number.
214 대한소화기학회지: 제49권 제4호, 2007
Table 1. Continued Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
*
XM_294906 Hypothetical LOC339306 LOC339306 4.120 3.407 5.340 0.035 NM_001461 Flavin containing monooxygenase 5 FMO5 3.543 2.717 6.525 0.036 XM_497062 Similar to hypothetical protein LOC441448 2.706 2.665 7.145 0.037
Cystic fibrosis transmembrane conductance
NM_000492 regulator, ATP-binding cassette CFTR 4.426 3.863 4.025 0.038 (sub-family C, member 7)
NM_177954 3.864 2.957 5.275 0.039
BG674026 S100 calcium binding protein A14 S100A14 6.459 3.539 2.072 0.039
M18728 5.440 2.555 3.824 0.041
CD013992 Glutathione S-transferase theta 2 GSTT2 2.711 4.308 4.239 0.044
NM_033046 Rhotekin RTKN 3.481 3.539 4.226 0.044
XM_290609 Hypothetical LOC338914 LOC338914 3.403 2.277 5.327 0.046 NM_138981 Mitogen-activated protein kinase 10 MAPK10 3.814 3.122 4.026 0.046
L-lactate dehydro- Full-length cDNA clone CS0DM007YJ24 genase A chain
CR591317 5.853 2.496 2.251 0.049
of fetal liver of Homo sapiens (EC 1.1.1.27) (LDH-A)
opHsV0400013399 2.581 3.397 4.586 0.049
Vesicle-associated membrane protein 2
NM_014232 VAMP2 3.047 2.493 4.951 0.050
(synaptobrevin 2)
opHsV0400004409 3.247 4.078 3.136 0.050
NM_212482 Fibronectin 1 FN1 5.587 2.243 2.599 0.051
Purinergic receptor P2X,
U83993 P2RX4 3.149 2.446 4.794 0.051
ligand-gated ion channel, 4
L19315 Cholecystokinin A receptor CCKAR 2.451 2.220 5.710 0.051
H300001961 3.199 2.861 4.252 0.052
XM_380154 Hypothetical protein LOC285972 LOC285972 3.227 2.236 4.776 0.052
BX640823 MGC32871 4.210 2.109 3.795 0.053
Coagulation factor VIII, procoagulant
NM_000132 F8 3.317 2.873 3.916 0.054
component (hemophilia A) Ssu72 RNA polymerase II CTD
AK023110 HSPC182 3.067 2.685 4.319 0.054
phosphatase homolog (yeast) Acetyl-coenzyme A acyltransferase 2
AK093311 ACAA2 4.849 2.224 2.934 0.055
(mitochondrial 3-oxoacyl-Coenzyme A thiolase)
BC042996 Carboxypeptidase A5 CPA5 2.129 2.110 5.722 0.055
AK127752 Adenosine A1 receptor RDC7 2.157 2.381 5.356 0.056
CR590366 Midkine (neurite growth-promoting factor 2) MDK 3.507 3.503 2.866 0.056 Hydroxyacyl-coenzyme A dehydrogenase/3-
NM_000182 ketoacyl-coenzyme A thiolase/enoyl-Coenzyme HADHA 4.317 2.388 3.155 0.056 A hydratase (trifunctional protein), alpha subunit
CR627200 Protease, serine, 1 (trypsin 1) PRSS1 5.000 2.719 2.137 0.056 Solute carrier family 9 (sodium/hydrogen
BX648303 SLC9A3R1 4.855 2.673 2.322 0.056
exchanger), member 3 regulator 1 Transcribed locus, strongly similar to
BE874574 2.556 3.030 4.151 0.057
NP_001931.1 atrophin-1 [Homo sapiens]
NM_016357 Epithelial protein lost in neoplasm beta EPLIN 3.757 3.225 2.716 0.058 NM_145202 Proline-rich acidic protein 1 PRAP1 3.554 2.103 4.025 0.058
* Locus link accession number.
김경래 외 10인. 위축 위염과 장형화생에서 마이크로어레이를 이용한 유전자 발현 215
Table 1. Continued Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
*
Transcribed locus, weakly similar to NP_536846.1
BF037218 2.537 2.613 4.500 0.058
cytochrome c oxidase subunit II [Homo sapiens]
LIM domain containing preferred
NM_005578 LPP 3.683 2.685 3.053 0.061
translocation partner in lipoma
opHsV0400013404 2.822 2.970 3.589 0.061
AB011123 TRAF2 and NCK interacting kinase TNIK 3.294 2.944 3.138 0.061 PREDICTED: Homo sapiens similar to
XM_497935 ferritin heavy chain (ferritin H subunit) LOC442090 3.416 2.113 3.773 0.062 (LOC442090), mRNA
BE737246 Small nuclear ribonucleoprotein polypeptide G SNRPG 3.053 2.166 3.970 0.064 NM_000860 Hydroxyprostaglandin dehydrogenase 15-(NAD) HPGD 3.694 2.736 2.523 0.067
AJ318054 SFRS protein kinase 1 SRPK1 2.860 2.703 3.353 0.068
BC068450 Sialic acid acetylesterase CSE-C 3.370 2.152 3.324 0.069 N-acylsphingosine amidohydrolase
NM_014435 ASAHL 3.723 2.137 2.934 0.069
(acid ceramidase)-like
J04755 Ferritin, heavy polypeptide pseudogene 1 FTHP1 2.853 2.066 3.854 0.070 NM_006410 HIV-1 Tat interactive protein 2, 30 kDa HTATIP2 3.610 2.212 2.935 0.070
Full-length cDNA clone CS0DI020YI20
BM913228 of Placenta Cot 25-normalized 3.090 2.121 3.481 0.071 of Homo sapiens (human)
AL050256 Chromosome 22 open reading frame 24 C22orf24 2.900 2.270 3.472 0.072 XM_293937 Similar to RIKEN cDNA 0610012A05 LOC345711 3.766 2.222 2.654 0.072 NM_006163 Nuclear factor (erythroid-derived 2), 45 kDa NFE2 2.129 2.141 4.254 0.074 NM_018128 Hypothetical protein FLJ10534 FLJ10534 2.705 2.322 3.468 0.074 NM_001012993 Chromosome 9 open reading frame 152 C9orf152 3.077 2.862 2.556 0.074
Mannosyl (alpha-1,3-)-glycoprotein beta-1,
AK023137 MGAT4B 2.640 2.239 3.604 0.074
4-N-acetylglucosaminyltransferase, isozyme B
H200011983 2.337 2.347 3.785 0.075
BC065937 5’-nucleotidase, ecto (CD73) NT5E 3.068 2.181 3.213 0.075 Q8NAN8_
AK092379 CDNA FLJ35060 fis, clone OCBBF2018828 2.418 2.125 3.915 0.075 HUMAN
opHsV0400002510 3.500 2.866 2.083 0.075
Calcium/calmodulin-dependent protein
CR604926 CaMKIINalpha 2.956 2.818 2.638 0.076
kinase II inhibitor 1
NR_002201 Ferritin, heavy polypeptide-like 3 FTHL3 2.998 2.157 3.095 0.078 BC022429 Hypothetical protein LOC90624 LOC90624 2.449 2.788 3.013 0.078 BC021004 Basic transcription factor 3-like 4 MGC23908 2.304 2.044 3.872 0.079 NM_178351 Late cornified envelope 1C LCE1C 2.550 3.015 2.504 0.082 BX537697 Hypothetical gene supported by BX537697 DKFZp686D0853 3.101 2.456 2.489 0.082
Interferon induced transmembrane
NR_001590 dJ377H14.5 2.771 2.588 2.649 0.083
protein 4 pseudogene
opHsV0400005209 2.503 2.068 3.264 0.087
AB208789 Sterol carrier protein 2 SCP2 2.712 2.787 2.319 0.087 AK056630 Zinc finger with KRAB and SCAN domains 1 ZKSCAN1 2.218 2.385 3.204 0.088 AK122775 BTB (POZ) domain containing 12 BTBD12 3.535 2.022 2.077 0.092
Sulfotransferase family, cytosolic
AB209149 SULT1A1 3.024 2.433 2.159 0.092
1A, phenol-preferring, member 1
AK025644 peroxisomal biogenesis factor 3 TRG18 2.096 2.675 2.794 0.093 BG271949 CDNA FLJ16097 fis, clone SYNOV2001660 2.662 2.268 2.622 0.094
* Locus link accession number.
216 The Korean Journal of Gastroenterology: Vol. 49, No. 4, 2007
Kim KR, et al. Gene Expression Profiling using Oligonucleotide Microarray in Atrophic Gastritis and Intestinal Metaplasia 217
Telomerase reverse transcriptase (TERT)와 telomerase-associa- ted protein 1 (TEP 1)에서는 발현양상이 불규칙하였다.
장형화생과 밀접한 관계를 가지는 Helicobacter pylori (H.
pylori) 감염 때 발현 변화가 알려진 유전자들의 변화를 확인 하였다. 세포 사멸과 관련된 caspase-3의 경우 유의하지는 않 으나 일정한 발현 증가 소견을 보였고, MAPK 10과 trefoil Table 1. Continued
Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
*
AL162047 Nuclear receptor coactivator 4 NCOA4 3.113 2.050 2.320 0.095 NM_173482 Hypothetical protein FLJ40365 FLJ40365 2.481 2.491 2.508 0.095
MRNA; cDNA DKFZp566B193 (from Non-protein coding
AK057104 2.579 2.127 2.767 0.096
clone DKFZp566B193) transcript
AV708957 Interferon induced transmembrane protein 3 (1-8U) IFITM3 2.066 2.465 2.859 0.098
AY358990 Hephaestin HEPH 2.233 2.352 2.717 0.100
NM_001680 FXYD domain containing ion transport regulator 2 FXYD2 2.324 2.884 2.078 0.101 AK093129 Testis-specific leucine zipper protein nurit NURIT 2.178 2.334 2.714 0.102
BC014016 Deoxyhypusine synthase DHPS 2.187 2.150 2.734 0.107
opHsV0400004364 2.581 2.414 2.099 0.107
Chromobox homolog 8
AF174482 CBX8 2.832 2.010 2.189 0.109
(Pc class homolog, Drosophila)
NM_014748 Sorting nexin 17 SNX17 2.214 2.018 2.732 0.111
XM_499198 LOC441699 LOC441699 2.218 2.351 2.386 0.111
D28588 Sp2 transcription factor SP2 2.057 2.581 2.306 0.112
AJ406948 Keratin associated protein 9-4 KRTAP9-4 2.343 2.354 2.229 0.112 6-phosphofructo-2-kinase/fructose-2,
NM_004567 PFKFB4 2.479 2.253 2.162 0.113
6-biphosphatase 4
NM_002390 ADAM metallopeptidase domain 11 ADAM11 2.066 2.123 2.669 0.115 Suppression of tumorigenicity 13 (colon
NM_003932 ST13 2.698 2.021 2.127 0.115
carcinoma) (Hsp70 interacting protein) UDP glucuronosyltransferase
J05428 UGT2B7 2.489 2.273 2.026 0.117
2 family, polypeptide B7
NM_015898 Zinc finger and BTB domain containing 7A ZBTB7 2.194 2.021 2.510 0.119
BX648171 Tropomyosin 1 (alpha) TPM1 2.212 2.026 2.449 0.121
H300014750 2.527 2.106 2.060 0.121
BG697954 Late cornified envelope 1D LCE1D 2.082 2.314 2.203 0.125 NM_173542 Hypothetical protein LOC196463 LOC196463 2.281 2.000 2.227 0.128 XM_496387 Similar to tripartite motif-containing 43 LOC440610 2.197 2.035 2.259 0.129
Rap guanine nucleotide exchange
NM_016339 RAPGEFL1 2.016 2.355 2.125 0.129
factor (GEF)-like 1
LOC389833;
AK074146 Similar to hypothetical protein MGC27019 2.007 2.421 2.065 0.129 FLJ35473
UDP-N-acetyl-alpha-D-galactosamine:
BC047468 polypeptide N-acetylgalactosa- GALNT7 2.041 2.341 2.098 0.130 minyltransferase 7 (GalNAc-T7)
DOT1-like, histone H3 methyltransferase
AB058717 DOT1L 2.085 2.251 2.083 0.132
(S. cerevisiae)
BX537968 Hypothetical LOC51149 LOC51149 2.243 2.031 2.103 0.134
NM_014903 Neuron navigator 3 NAV3 2.140 2.202 2.035 0.134
CR591007 Chemokine (C-C motif) ligand 3 CCL3 2.021 2.121 2.187 0.137 NM_153818 Peroxisome biogenesis factor 10 PEX10 2.033 2.053 2.160 0.141 XM_499061 Hypothetical protein LOC340281 LOC340281 2.154 2.040 2.020 0.142
* Locus link accession number.
Table 2. The Expression of Genes Related to the Gastric Carcinogenesis in Gastric Mucosa of Atrophic Gastritis and Intestinal Metaplasia
Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
Fibronectin
AK026015 Fibronectin type III domain containing 4 FNDC4 1.351 0.982 1.198 0.672 BC057762 Fibronectin type III domain containing 7 FLJ35838 1.137 1.111 1.017 0.830
Leucine rich repeat and fibronectin type
XM_290842 LRFN1 0.710 1.048 1.352 0.929
III domain containing 1
NM_212482 Fibronectin 1 FN1 5.587 2.243 2.599 0.051
AK092102 Fibronectin type III domain containing 5 FNDC5 2.458 2.037 1.727 0.142 Integrin, beta 1 (fibronectin receptor,
NM_002211 beta polypeptide, antigen CD29 ITGB1 2.101 1.877 1.503 0.192 includes MDF2, MSK12)
Telomerase
AF018167 Telomerase reverse transcriptase TERT 0.964 1.096 0.936 0.997 NR_001566 Telomerase RNA component TR; hTR; 0.511 0.515 0.491 0.156
TRC3
U86136 Telomerase-associated protein 1 TEP1 1.116 0.778 0.890
FGF
BC012347 Fibroblast growth factor 13 FGF13 1.254 1.435 1.454 0.415 Fibroblast growth factor receptor 1 (fms-related
NM_023110 FGFR1 1.413 1.479 1.238 0.419
tyrosine kinase 2, Pfeiffer syndrome) Fibroblast growth factor receptor 2
NM_023028 (bacteria-expressed kinase, FGFR2 1.270 1.337 1.508 0.423 keratinocyte growth factor receptor)
NM_033163 Fibroblast growth factor 8 (androgen-induced) FGF8 1.180 1.258 1.434 0.510 EGFR
Epidermal growth factor receptor
AK022370 (erythroblastic leukemia viral (v-erb-b) EGFR 1.293 1.386 1.303 0.468 oncogene homolog, avian)
Epidermal growth factor receptor
AK026818 (erythroblastic leukemia viral (v-erb-b) EGFR 1.286 1.350 1.349 0.467 oncogene homolog, avian)
NM_001963 Epidermal growth factor (beta-urogastrone) EGF 1.116 1.482 1.581 0.404 VEGF
similar to Vascular endothelial growth factor receptor 1 precursor (VEGFR-1)
XM_497921 LOC391533 1.263 1.426 0.985 0.598
(Vascular permeability factor receptor) (Tyrosine-protein kinase receptor FLT) (Flt-1)
NM_003376 Vascular endothelial growth factor VEGF 1.365 1.317 1.164 0.521 C-fos induced growth factor (vascular
NM_004469 FIGF 0.819 0.872 0.612 0.491
endothelial growth factor D) TGF
NM_003236 Transforming growth factor, alpha TGFA 0.465 0.401 0.435 0.111 Transforming growth factor, beta 1
X02812 TGFB1 0.362 0.382 0.470 0.096
(Camurati-Engelmann disease)
NM_003238 Transforming growth factor, beta 2 TGFB2 0.471 0.196 0.065 Transforming growth factor beta 1
AK122975 TGFB1I1 0.428 0.525 0.860 0.245
induced transcript 1
XM_498233 Similar to TGF-beta receptor binding protein LOC442342 0.815 0.699 0.419 0.291 Transforming growth factor, beta receptor I
NM_004612 TGFBR1 0.816 0.857 0.574 0.455
(activin A receptor type II-like kinase) 218 대한소화기학회지: 제49권 제4호, 2007
김경래 외 10인. 위축 위염과 장형화생에서 마이크로어레이를 이용한 유전자 발현 219
factor 3의 경우 유의하게 과발현되었다. 이외의 유전자에서 는 특징적인 변화가 없었다(Table 4).
고 찰
위암 발병 요인 중 H. pylori, 만성위염, 장형화생 등이 많 은 관심을 끌고 있다. 만성 위축 위염과 관계된 장형성이상 은 위암, 특히 장형 위암의 발생과 관련이 있다.5,6장형화생 이란 정상적인 위점막 상피세포가 장상피와 닮은 형태인 장
특이적인 술잔세포(goblet cell), 흡수세포(absorptive cell), 솔변 연(brush border) 등을 가지는 장 표현형을 갖는 것을 말하며 임상에서는 비교적 흔한 병변으로 나이가 들수록 증가한다.
장형화생 빈도를 위암 환자와 대조군을 비교했을 때, 위암 에서 유의하게 높았으나 대조군에서도 꽤 높은 빈도로 발현 되므로, 전암 병변으로서의 활용을 위해 장형화생을 세분화 하여 연구한다. 대부분의 경우 대장형 sulphomucin을 가진 장형화생의 아형이 장형위암과 관련이 있다는데 의견이 일
치한다.3,4,7,8 국내 연구에 따르면 같은 연령군에서 제1, 2형
Table 3. Significant Changes of Expression (>2.0 Fold) of Genes Related to Gastric Carcinogenesis as Compared with Normal Mucosa Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
NM212482 Fibronectin 1 FN 1
*
5.5873 2.243 2.599 0.051Src-related kinase C-terminal regulatory
NM080823 SRMS† 0.2283 0.26 0.226 0.036
tysosine and N-terminal myristylation site
DQ186648 Tumor protein p53 (Li-Frumeni syndrome) TP53† 0.3526 0.261 0.135 0.039 AY422170 Tumor protein p53 inducible nuclear protein 2 TP53INP2† 0.4634 0.495 0.343 0.111 NM147283 Tumor protein p53 inducible protein 3 TP53I3† 0.4386 0.412 0.335 0.093 NM022963 Fibroblast growth factor receptor 4 FGFR4† 0.459 0.392 0.314 0.088
Transforming growth factor, beta 1
XD2812 TGFB1† 0.3616 0.382 0.47 0.096
(Camurati-Engelmann disease)
NM003236 Transforming growth factor, alpha TGFA† 0.4652 0.401 0.435 0.111
*
up-regulated genes.†down-regulated genes.
Table 2. Continued Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
NM_138960 TGFB-induced factor 2-like, X-linked TGIF2LX 0.621 0.903 0.745 0.469 NM_003242 Transforming growth factor, beta receptor II TGFBR2 0.827 0.906 0.903 0.735 NM_000358 Transforming growth factor, beta-induced, 68kDa TGFBI 0.945 0.993 0.744 0.771 NM_138960 TGFB-induced factor 2-like, X-linked TGIF2LX 0.948 0.847 1.030 0.879 BF215849 TGF beta-inducible nuclear protein 1 TINP1 0.890 1.044 1.042 0.984
Latent transforming growth factor
NM_206943 LTBP1 0.290 0.235 0.117 0.030
beta binding protein 1
NM_021809 TGFB-induced factor 2 (TALE family homeobox) TGIF2 0.974 1.281 1.337 0.639 Transforming growth factor, beta receptor
NM_004257 TGFBRAP1 1.193 1.067 1.091 0.777
associated protein 1
NM_170695 TGFB-induced factor (TALE family homeobox) TGIF 1.055 1.096 1.029 0.885 AK122902 Transforming growth factor, beta 3 TGFB3 0.983 1.038 1.026 0.970 AB063300 TGF-beta induced apotosis protein 2 TAIP-2 1.522 1.535 0.308
Similar to TGF beta-inducible nuclear
XM_293342 LOC347424 1.199 1.536 1.314 0.445
protein 1; L-name related protein Macrophage stimulating 1 receptor
NM_002447 MST1R 1.121 1.205 1.065 0.752
(c-met-related tyrosine kinase)
220 The Korean Journal of Gastroenterology: Vol. 49, No. 4, 2007
장형화생에서는 위암종 및 관상선종과 양성 질환 사이에 차 이가 없으나 제3형에서는 양성 질환보다 위암종과 관상선 종에서 더 높은 빈도를 보였다.9Silva 등10의 전향 추적 조사 결과에서도 제3형 장형화생의 위험성을 보여 준다. 만성 위 축 위염과 관계된 장형화생에서 관련 유전자에 대한 연구는 많지 않으나 MUC2, SI (sucrase-isomaltase: alpha-glucosidase), carbonic anhydrase I 정도를 들 수 있다. 이번 실험 결과에서 도 SI 및 Mucin 1의 뚜렷한 발현을 확인함으로써 기존의 장 형화생과 관련있다고 밝혀진 이러한 유전자 발현을 통해 그 관련성을 재확인할 수 있었다.
위암 발생의 중요 요인의 하나로 여겨지는 H. pylori 감염
은 위 위축이나 장형화생으로의 진행 초기과정에 관여한다.
물론 H. pylori 감염뿐 아니라 음식이나 담즙 역류 등 여러 가지 요소가 같이 작용을 하고,11임상적으로는 입증되지 않 았으나 지속적인 H. pylori 감염이 선 위축과 장형화생을 야 기하고 결국 위선암종으로 진행된다는 가설은 동물 실험을 통해 제기된 바 있다.12,13 또한 장형화생의 대부분이 H.
pylori 감염과 연관된 만성 염증 이후에 발생하며 감염과 관 련된 단백인 Src Homology phosphatase 2 (SHP-2)와 IL-Iβ 등이 세포 내 신호전달에 영향을 주어 결국 장 특이 전사인 자로 세포 발생, 증식과 분화 그리고 장상피층의 항상성 유 지에 관여하는 Cdx-2 유전자 발현에도 영향을 준다는 보고 Table 4. Expression Profiling of Typically Known Genes Associated with Infection of Helicobacter pylori in Gastric Mucosa of Atrophic Gastritis and Intestinal Metaplasia
Accession
Gene name Gene symbol Sample 1 Sample 2 Sample 3 p-value number
JUK/SAPK
AF136407 TRAF3 interacting protein 2 C6orf4 0.668 0.703 0.978 0.521 TRAF3 interacting protein 2
AF136407 C6orf4 1.023 1.329 1.334 0.592
Old name: adapter protein CIKS Trefoil factor 2
BI517365 Trefoil factor 2 (spasmolytic protein 1) TFF2 0.132 2.043 0.698 0.914 BU536516 Trefoil factor 3 (intestinal) TFF3 1.802 2.175 8.014 0.040
Trefoil factor 1 (breast cancer,
NM_003225 TFF1 0.315 4.787 1.143 0.141
estrogen-inducible sequence) Proline-rich protein
AK131382 Proline rich 3 PRR3 0.611 1.055 0.934 0.707
BG190758 Small proline rich protein 4 SPRR4 1.084 1.243 1.017 0.780 Caspase-3
NM_004346 Caspase 3, apoptosis-related cysteine peptidase CASP3 1.785 1.533 1.363 0.291 VAC A
BX647725 Vacuolar protein sorting 26 homolog A (yeast) VPS26 0.488 0.869 1.373 0.807 NM_032353 Vacuolar protein sorting 25 (yeast) MGC10540 0.705 0.903 1.089 0.783 AL834261 Vacuolar protein sorting 37C (yeast) VPS37C 0.447 1.627 0.835 0.939 CR608392 Vacuolar protein sorting 72 (yeast) TCFL1 1.029 0.980 0.734 0.818 AL834189 Vacuolar protein sorting 37A (yeast) FLJ32642 0.928 0.996 0.885 0.867 NM_017890 Vacuolar protein sorting 13B (yeast) COH1 0.596 1.095 1.271 0.975 MAPKK
NM_138980 Mitogen-activated protein kinase 10 MAPK10 1.081 1.166 1.160 0.743 Mitogen-activated protein kinase 8
NM_012324 MAPK8IP2 1.140 1.254 1.044 0.724
interacting protein 2
NM_002758 Mitogen-activated protein kinase kinase 6 MAP2K6 1.280 1.235 0.887 0.746 NM_002745 Mitogen-activated protein kinase 1 MAPK1 1.057 1.144 1.013 0.863 AK093838 Mitogen-activated protein kinase kinase 3 MAP2K3 1.145 0.968 1.085 0.873 NM_145162 Mitogen-activated protein kinase kinase 5 MAP2K5 1.281 1.026 0.885 0.877 NM_002755 Mitogen-activated protein kinase kinase 1 MAP2K1 0.939 1.185 0.977 0.935 BX537897 Mitogen-activated protein kinase 3 MAPK3 0.978 1.058 0.995 0.980
Mitogen-activated protein kinase kinase
NM_145686 MAP4K4 0.927 1.141 0.975 0.973
kinase kinase 4
NM_138981 Mitogen-activated protein kinase 10 MAPK10 3.814 3.122 4.026 0.046
Kim KR, et al. Gene Expression Profiling using Oligonucleotide Microarray in Atrophic Gastritis and Intestinal Metaplasia 221
도 있다.14 마우스를 이용한 동물실험에서는 Cdx-2 유전자 과발현 시 위장점막에서 장형화생이 발생하였고, 인간조직 을 대상으로 Cdx-2 유전자가 위점막의 장형화생에서 발현 되며 장형 위선암종 점막에서 발현이 증가하고, 미만형 위 선암종 점막에서는 발현이 감소함을 확인하여, 장형 위선암 종 발생에 중요한 역할을 한다고 보고하였다.15,16 H. pylori 감염과 위암과의 관계는 혈청 항 H. pylori 항체의 측정을 통한 역학 검토에서 확인하였으며,17여러 동물 실험을 통해 서나 임상관찰을 통해서도 확인되었다.6,18,19
이번 실험의 결과에서도 SHC (Src homology 2 domain containing) family, member (RaLP), Src homology 2 domain containing transforming protein (SHD), Src homology 2 domain containing adaptor protein B (SHB), SHC transforming protein 1 (SHC1), Src family associated phosphoprotein 1 (SCAP1) 등 Src homology 관련 유전자의 발현 양상을 확인하였으나 특 이 유전자 발현은 보이지 않았다. 인체 생검 조직을 대상으 로 세포독소관련 단백질(CagA)과 SHP-2와의 복합체 형성이 확인되었으나 이번 연구의 대상인 장형화생과 위암 발생 사 이에서는 복합체 형성이 확인되지 않았다.20 즉, CagA에 의 한 위점막 상피세포 내 변화는 위암발생가설인 정상 점막에 서 표층 위염, 위축 위염까지의 시기에 지속적으로 작용할 것으로 생각할 수 있다. cagA 유전자는 3’영역의 아미노산 반복배열 차이에 의해 몇 가지 종류로 나눌 수 있으며 지역 특색을 보인다. 또 다른 병원 유전자인 vacA 유전자는 몇몇 균주에서만 VacA 단백을 분비하며, CagA와는 다른 염증 반 응에 관여한다.
이외에 H. pylori가 위상피세포에 부착할 때 새로운 대립 형질이 발현되어 생기는 새로운 독성인자인 ice 유전자가 있으며, 그 아형인 iceA1의 소화성 궤양과의 관련성이 제기 되었으나 여러 연구에서 cagA, vacA 그리고 iceA 유전자형 과 임상질환과의 연관성은 없었고 인종 차이 비교에서도 임 상 차이를 보이지는 않았다.21-23 이번 연구의 경우 이런 cagA, vacA 그리고 iceA 유전자 발현은 확인하지 못하였다.
감염 후 점막은 상피세포의 손상으로 세포의 방어 능력 저 하, H. pylori에 의한 세포 독성 물질과 점막 내 사이토카인 유발, 그리고 이에 의해 활성화된 염증세포 침윤 단계를 거 치며, 특히 세포사멸현상은 여러 in vivo 및 in vitro 실험에 서 증명되고 손상기전의 하나로 여겨진다. 세포사멸과 관련 된 중요효소로서 caspase는 세포 내 전구효소로 존재하다가 다른 단백 분해효소나 자가 공정을 통해 활성화되며, 이후 PARP를 포함한 기질들을 분해하고 기능을 억제하여 그 형 태학적인 변화와 oligonucleotide DNA fragmentation에 관여 한다.24 진 등25도 위암세포주를 대상으로 H. pylori에 의한 caspase-3 활성화와 PARP 절단을 보고하였다. cagA, vacA 또 는 picB에 따른 세포사멸 영향에 대해서는 아직 결론이 나
지 않았다.
세포 내 신호전달물질 중 mitogen-activated protein kinase (MAPK)는 외부자극에 대한 세포의 성장, 사멸, 분화에 관 여하는 중요 조절인자의 하나이다. 그러나 이런 MAPK의 여러 반응이 다양한 자극과 세포형태에 따라 다르게 기능을 나타내므로 H. pylori 감염에 특이적이라고 말할 수는 없 다.26이번 실험 결과에서도 여러 종류의 MAPK가 불규칙한 형태의 유전자 발현 결과를 보여 감염에 비특이적이라는 내 용을 뒷받침하였으며 caspase-3의 경우 통계학적으로 뚜렷 하지는 않으나 모두 증가하여 위의 내용과 유사한 결과를 보였다(Table 4). 국내 연구에 의하면 H. pylori는 MAPK의 세 구성성분, ERK (extracellular signal-regulated kinase), JNK (c-Jun N-terminal kinases), p38 kinase 모두를 활성화시키며, p38 kinase 활성화는 부분적이지만 caspase-3 활성화와는 독 립적으로 H. pylori에 의한 세포사멸에 관여한다고 한다.25 H. pylori 감염에 의한 세포 방어 능력 저하에는 많은 요인 들이 관여하며 대표적으로 prostaglandin E2 receptor EP4, proline-rich protein 2A, trefoil factor-2를 들 수 있으며 prostaglandin E2 합성과 분비는 in vivo 및 in vitro 실험에서 증명되었다. Prostaglandin E2 는 바로 수용체 아형을 활성화 시킴으로써 세포사멸로부터 위점막 세포를 보호한다.27,28이 번 실험에서 small proline-rich protein 4의 경우 세 시료 모두 정상 점막에 비해 증가된 결과를 보였으나 trefoil factor-2 유 전자는 그 발현이 불규칙하였다. DNA 복제 완성에 필수적 인 telomerase의 경우 장형화생의 23%와 선종의 50%에서 활 성이 보고되고 있고, 대부분의 원발 위암에서 정상점막에 비해 그 활성이 높고 H. pylori 감염과도 관계가 있다.29 이번 실험에서는 telomerase RNA component 유전자에서만 모두 일정하게 저발현되었음을 확인하였다. 상피세포성장인자 수용체 및 성장인자인 c-erbB1/ EGF receptor, EGF, TGF-α 등이 위암 발생에 관여하며, EGF receptor와 c-erbB2, c-met 발현은 암 발생 초기 단계에, p53 유전자는 후기에 관여한 다고 알려져 있다. 이번 실험에서는 TGF- α, TGF-β1, FGF 모두 정상 점막에 비해 오히려 저발현되었으며 p53의 경우 도 tumor protein p53 inducible nuclear protein 2, tumor protein p53, tumor protein p53 inducible protein 3 모두 저발현되어 위암 발생과의 관계를 확인할 수 없었다. 단지 종양 원유전 자 중 위암 발생과 관련된 c-met의 과발현만을 확인하였다.
생물 정보학의 발전과 컴퓨터 사이언스와 같은 기능 유전 체 분석 기술의 접목으로 DNA 마이크로어레이 같은 극미 량의 시료로도 수만 개의 유전자를 한꺼번에 분석할 수 있 는 기술이 진보하였으며, 수많은 유전 정보들을 손쉽게 분 석하여 목표 질환에 대한 예측, 종류 판별, 치료 방법 평가 등을 통해 환자 개개인의 유전 소인에 따른 진단과 치료가 결정된다는 소위 “맞춤 의학”까지 바라보게 되었다.30,31 이
222 대한소화기학회지: 제49권 제4호, 2007
번 실험에 사용된 올리고뉴클레오티드 마이크로어레이는 기존의 cDNA 마이크로어레이와는 달리 20∼25개의 염기로 이루어진 수천 내지 수만 개의 DNA probe를 질소 셀룰로오 스막이 아닌 슬라이드 유리와 같은 기판 위에 고밀도로 정 렬하여 고정한 후 목표로 하는 세포와 비교 세포들로부터 추출된 mRNA를 역전사(reverse transcription)시켜 cDNA를 합성하여 어레이의 염기와 결합시키는 것이다. cDNA 마이 크로어레이 칩과는 달리 완전한 유전자(full-length open leading frame)대신에 보다 짧은 염기를 사용하기 때문에 집 적되는 유전자의 양이 더욱 커진다. 이런 마이크로어레이 기술은 한 번의 실험으로 동시에 많은 유전자들에 대한 발 현 변이 양상을 탐색할 수 있으며 또한 유전자의 진단, 돌연 변이, 의약품의 효과 확인 및 질병 진단 등에 널리 응용될 수 있어 새로운 차원의 HTS (high-throughput screening) 시스 템이라고 할 수 있다.
마이크로어레이가 여러 분야에 도입된 후 임상에서는 주 로 각종 암과 관련된 특이 유전자 확인과 질병의 새로운 분 류 등에 사용되었다. 주된 응용분야는 유전자 발현이며 genomic DNA 수준에서 염기 다형증 및 돌연변이연구에 이 용되기도 한다.32 위암과 관련하여서는 cDNA 마이크로어레 이를 사용하여 위암을 침윤군과 비침윤군으로 나누어 그 관 련 유전자를 조사하였으며 특히 matrix metalloproteinase 7, keratin 6B, thrombospondin 2, IGF binding protein 3 등이 유 의하게 증가되었다는 보고가 있다.33,34Hasegawa 등35은 장형 위암에서 림프절 전이와 관련된 12개의 유전자를 찾았고 최 종적으로 5개의 유전자가 림프절 전이와 관계있다고 하였 다. 향후 유전자 발현 패턴에 관한 연구와 함께, 위암의 조 직 분화도 및 형태학 분류를 고려한 세부군으로 나누어 마 이크로어레이를 이용하여 환자의 예후나 치료반응 예측 등 에 대한 연구가 계속 될 것이다. 국내에서는 이 등36이 조기 위암과 진행 위암을 대상으로 cDNA 마이크로어레이를 이 용하여 44개의 유전자 발현 증가와 92개 유전자 발현 감소 를 보고하였다. 이 실험에서는 유전자 검증을 위해 RT-PCR 을 시행하였다.
이상에서 위암을 대상으로 마이크로어레이를 이용한 유 전자 발현 보고와 H. pylori 감염에 관련된 보고는 많으나 위암발생의 전 단계 병변으로 알려진 장형화생을 보이는 위 축 위염을 대상으로 한 결과는 없다. 따라서 저자들은 장형 화생을 보이는 위축 위염에 대해 올리고뉴클레오티드 마이 크로어레이를 이용하여 유전자 전반의 변화를 살펴 정상 점 막 유전자와의 차이점을 확인하고 관련 유전자 탐색의 기초 를 만들고자 하였다. 이번 연구에서 과다 혹은 과소 발현되 었거나 공통적으로 뚜렷한 발현 변화를 보인 유전자들이 장 형화생을 보이는 위축 위염에서 특이성과 재현성이 있는가 에 대해서는 더 많은 실험이 필요할 것이다. 또한 각 유전자
별로 추후에 면역조직화학 연구, real time-PCR 등을 통한 특정 유전자 검증과정을 거쳐야 할 것이다. 이번 연구는 올 리고뉴클레오티드 마이크로어레이를 이용한 장형화생을 보 이는 위축 위염에 대한 일종의 선험 연구(pilot study)에 불과 하나 이후 이 결과들을 기초로 유전자 검증과정을 통해 암 화 진행에 중요한 유전자를 발굴하고, 이를 이용한 잠재암 환자에서 위암 발병 소인을 검출하는 선별 검사를 위한 kit 제작과 같은 여러 임상 응용도 가능하리라 생각한다.
요 약
목적: 장형화생을 동반한 위축 위염은 최근까지 위암발 생의 중요한 요인으로 여겨져 왔다. 그러나 아직 장형화생 과 위축 위염에 발현하는 주요 유전자에 관해서 거의 알려 진 것이 없다. 이번 연구는 기존의 유전자 탐색에 사용되었 던 어떤 방법보다도 많은 장점을 가지고 있는 올리고뉴클레 오티드 마이크로어레이를 이용하여 전반적인 유전자 변화 와 함께 위암과의 관련성을 알아보고자 하였다. 대상 및 방 법: 장형화생을 동반한 위축 위염환자의 위점막과 정상 점 막에서 올리고뉴클레오티드 마이크로어레이 기술을 이용하 여 대량의 유전자 정보를 얻었으며, 유전자들의 확인을 위 해 SAM (Significance Analysis of Microarrays) package 방법 을 사용하였다. Global normalization, intensity dependent normalization 그리고 box plot normalization를 사용하여 결과 를 분석하였다. 결과: FABP, REG, OR6C1, MEP1, SLC6A1, SI, Mucin 1, RAB23 등 8개 유전자는 정상점막에 비해 10배 이상 과발현되었고, LOC44119유전자 하나만이 10배 이상 저발현되었다. 알려진 위암의 발암과정에 관여된 유전자로 는 FN1, SRMS, TP53, TP53IMP2, TP53I3, FGFR4, TGFB1, TGFA 등 모두 8개에서 공통적으로 2배 이상의 발현 증감을 보였다. 결론: 저자들은 위암의 전단계병변인 장형화생을 동반한 위축 위염에서 암화와 관련한 유전자를 확인할 수 있었으며, 이번 연구 결과는 위암 진단 및 치료와 같은 임상 적용을 위한 분자생물학적인 특성을 이해하는 데에 도움이 될 것이다.
색인단어: 위암, 위축 위염, 장형화생, 올리고뉴클레오티드 마이크로어레이, 유전자 발현
참고문헌
1. Correa P. Human gastric carcinogenesis. A multistep and mul- tifactorial process-first American Cancer Society award lecture on cancer epidermiology and prevention. Cancer Res 1992:52:
6735-6740.
2. Ramesar KC, Sanders DS, Hopwood D. Limited value of type
김경래 외 10인. 위축 위염과 장형화생에서 마이크로어레이를 이용한 유전자 발현 223
III intestinal metaplasia in the development of gastric carci- noma. J Clin Pathol 1987;40:1287-1290.
3. Filipe MI, Munoz N, Matko I, et al. Intestinal metaplasia types and the risk of gastric cancer: a cohort study in Slovenia. Int J Cancer 1994;57:324-329.
4. Knawy BA, Morad NA, Jamal A, Hamdi J, Mirdad S. Non- neoplastic changes in gastric antrum: are they different in dis- tally located intestinal and diffuse-type gastric adenocarcinoma?
Eur J Cancer Prev 1997;6:167-170.
5. Stemmermann GN. Intestinal metaplasia of the stomach. A status report. Cancer 1994;113:478-486.
6. Uemura N, Okamoto S, Yamamoto S, et al. Helicobacter py- lori infection and the development of gastric cancer. N Engl J Med 2001;345:784-789.
7. Filipe MI. mucins in the human gastrointestinal epithelium; a review. Invest Cell Pathol 1979;2:195-216.
8. Huang CB, Xu J, Huang JF, Meng XY. Sulphomucin colonic type intestinal metaplasia and carcinoma in the stomach. Can- cer 1986;57:1370-1375.
9. Lee JH, Kim HJ, Lee HY, Cho JH, Ko GH. Relationship between the gastric carcinoma and subtypes of intestinal me- taplasia in gastric mucosa. Korean J Gastroenterol 1997;30:1-8 10. Silva S, Filipe MI, Pinho A. Variants of intestinal metaplasia in the evolution of chronic atrophic gastritis and gastric ulcer.
A follow up study. Gut 1990;31:1097-1105.
11. Sobala GM, Pignatelli B, Schorah CJ, et al. Level of nitrite, nitrate, N-nitroso compounds, ascorbic acid and total bile acids in gastric juice of patient with and without precancerous con- ditions of the stomach. Carcinogenesis 1991;12:193-198.
12. Kuipers EJ, Uyterlinde AM, Pena AS, et al. Long term seque- lae of Helicobacter pylori gastritis. Lancet 1995;345:1525- 1528.
13. Honda S, Fujioka T, Tokieda M, Satoh R, Nishizono A, Nasu M. Development of Helicobacter pylori-induced gastric carci- noma in Mongolian gerbils Cancer Res 1998;58:4255-4259.
14. Yuasa Y. Control of gut differentiation and intestinal-type gas- tric carcinogenesis. Nat Rev Cancer 2003;3:592-600.
15. Silberg DG, Sullivan J, Kang E, et al. Cdx1 ectopic expression induces gastric intestinal metaplasia in transgenic mice. Gas- troenterology 2002;122:689-696.
16. Paik CH, Han DS, Lee SH, et al. Expression of Cdx-2 homeo- box gene in intestinal metaplasia and gastric adenocarcinoma.
Korean J Gastroenterol 2004;44:186-192.
17. The Eurogast Study Group. An international association bet- ween Helicobacter pylori infection and gastric cancer. Lancet 1993;341:1359-1362.
18. Sugiyama A, Maruta F, Ikeno T, et al. Helicobacter pylori
infection enhances N-methyl-N-nitrosourea-induced stomach carcinogenesis in Mongolian gerbil. Cancer Res 1998;58:2067- 2069.
19. Smith VC, Genta RM. Role of Helicobacter pylori gastritis in gastric atrophy, intestinal metaplasia, and gastric neoplasia. Mi- crosc Res Tech 2000;48:313-320.
20. Yamazaki S, Yamakawa A, Ito Y, et al. The CagA protein of Helicobacter pylori is translocated into epithelial cells and binds to SHP-2 in human gastric mucosa. J Infect Dis 2002;
187:334-337.
21. Huang JQ, Zheng GF, Sumanac K, Irvine EJ, Hunt RH. Meta- analysis of the relationship between cagA seropositivity and gastric cancer. Gastroenterology 2003;125:1636-1644.
22. van Doorn LJ, Figueiredo C, Sanna R, et al. Clinical relevance of the cagA, vacA, and iceA status of Helicobacter pylori. Gas- troenterology 1998;115:58-66.
23. Lee JW, Jeong S, Kim IH, et al. Relationship between gene subtype of Helicobacter pylori virulence factor and upper gas- trointestinal diseases. Korean J Gastroenterol 2001;38:89-97.
24. He J, Whitacre CM, Xue LY, Berger NA, Oleinick NL. Pro- tease activation and cleavage of poly (ADP-ribose) polymerase:
an integral part of apoptosis in response to photodynamic treatment. Cancer Res 1998;58:940-946.
25. Jin SH, Kim TI, Yeo M, Choi JW, Kim C, Kim WH. The activation of caspase-3 and mitogen activated protein kinases during H. pylori-induced apoptosis in gastric cancer cells.
Korean J Gastroenterol 2001;37:418-427.
26. Moss SF. Epithelial cell turnover and apoptosis. In: Hunt RH, Tytgat GNJ, ed. Helicobacter pylori, basic mechanism to clini- cal cure. London: Kluwer Academic Publishers, 1998:110-122.
27. Hoshino T, Tsutsumi S, Tomisato W, Hwang HJ, Tsuchiya T, Mizushima T. Prostaglandin E2 protects gastric mucosal cells from apoptosis via EP2 and EP4 receptor activation. J Biol Chem 2003;278:12752-12758.
28. Mueller A, Merrell DS, Grimm J, Falkow S. Profiling of microdissected gastric epithelial cells reveals a cell type-spe- cific response to Helicobacter pylori infection. Gastroentero- logy 2004;127:1446-1462.
29. Tahara H, Kuniyasu H, Yokosaki H, et al. Telomerase activity in preneoplastic and neoplastic gastric and colorectal lesions.
Clin Cancer Res 1995;1:1245-1251.
30. Choudhuri S. Microarrays in biology and medicine. J Biochem Mol Toxicol 2004;18:171-179.
31. Ramaswamy S, Golub TR. DNA microarrays in clinical onco- logy. J Clin Oncol 2002;20:1932-1941.
32. Kumar A, Goel G, Fehrenbach E, Puniya AK, Singh K.
Microarrays: the technology, analysis and application. Eng Life
224 The Korean Journal of Gastroenterology: Vol. 49, No. 4, 2007
Sci 2005;3:215-222.
33. Inoue H, Matsuyama A, Mimori K, Ueo H, Mori M. Pro- gnostic score of gastric cancer determined by cDNA micro- array. Clin Cancer Res 2002;8:3475-3479.
34. Haraguchi N, Inoue H, Mimori K, et al. Analysis of gastric cancer with cDNA microarray. Cancer Chemother Pharmacol 2004;54(suppl):21S-24S.
35. Hasegawa S, Furukawa Y, Li M, et al. Genome-wide analysis of gene expression in intestinal-type gastric cancers using a complementary DNA microarray representing 23,040 genes.
Cancer Res 2002;62:7012-7017.
36. Lee JH, Choi SR, Hwang TH, et al. A gene expression in study human gastric adenocarcinoma using a cDNA micro- array. Korean J Gastroenterol 2003;42:484-495.