334
©The Korean Society of Food Science and Technology
16S rRNA 유전자 염기서열 분석에 기반한 국내 재배 오이의 상재균총 분석
서동우
1,†· 김승민
2,†· 이현열
1· 염수진
1· 정희곤
1,*
1충남대학교 식품공학과, 2한국방송통신대학교 생활과학부
16S rRNA gene-based sequencing of cucumber (
Cucumis sativus L.)
microbiota cultivated in South Korea
Dong Woo Seo1,†, Seung Min Kim2,†, Heoun Reoul Lee1,Su-jin Yum1, and Hee Gon Jeong1,*
1Department of Food Science and Technology, Chungnam National University 2Division of Human Ecology, Korea National Open University
Abstract Various vegetables, including cucumbers, have a high probability of foodborne illness because they are usually eaten raw. In this study, we analyzed the 16S rRNA gene sequences of the cucumber (Cucumis sativus L.) microbiota. The diversity indices of cucumber cultivated in May were higher than in cucumber cultivated in November. At the phylum level, Proteobacteria, Firmicutes, and Actinobacteria were predominant. The classes generally comprised Gammaproteobacteria, Bacilli, Alphaproteobacteria, and Actinobacteria. At the genus level, the proportions of Aureimonas, Escherichia, and Microbacterium in samples from May were relatively high, whereas Enterococcus, Pseudomonas, and Rhizobium accounted for a higher proportion in samples from November. Moreover, it is noteworthy that potential pathogenic genera such as Acinetobacter, Aerococcus, Aureimonas, Enterobacter, Enterococcus, Escherichia, Pantoea, Pseudomonas, and Staphylococcus were detected. Although further studies on the characteristics of potential pathogens are required, our results can be used to improve the food safety of vegetables.
Keywords: 16S rRNA gene-based sequencing, Food safety, Cucumber, Microbiota
서
론
식중독은 매년 전세계적으로 사망자를 포함하여 많은 환자를 야기하는 커다란 문제 중 하나이다. 미국 질병통제예방센터(Centers for Disease Control and Prevention, CDC)에 따르면 2014년부터 2018년까지 미국에서 연평균 15,672건의 식중독이 발생하였고 (CDC, 2020), 국내의 경우 2015년에서 2019년까지 5년간 연평균 6,874명의 식중독 환자가 보고되었다(MFDS, 2020). 지속적인 식 중독의 발생은 보다 높은 수준의 식품 안전성에 대한 필요성을 보여주고 있으며, 이를 위해서는 정확한 식중독 발생 원인 규명 이 필수적이다. 그러나 현재까지는 식중독 원인을 파악하기 어려 운 사례가 빈번하게 보고되고 있다. 통계에 따르면 2016년 미국 에서 발생한 식중독 사고 중 원인을 알 수 없는 식중독의 발생 건수는 약 21%를 차지하고 있고(CDC, 2019), 2015년에서 2019 년사이 국내에서 발생한 식중독 중 원인불명의 사고는 약 41% 에 달한다(MFDS, 2020). 이러한 원인불명 식중독은 기존의 배양 기반(culture dependent) 검출법으로 검출이 불가능한 미생물에 의 한 것으로 추측되고 있다(McCabe-Sellers과 Beattie, 2004). 기존의 배양 기반 검출법이 가지는 한계를 극복하기 위해, 최 근에는 metagenomics 관점에서 식품 내 미생물을 분석하고 식품 안전성의 수준을 향상시키기 위한 연구들이 활발히 이루어지고 있다(Bergholz 등, 2014; Handelsman, 2004). Metagenomics는 특 정 환경 내에 존재하는 모든 미생물의 DNA를 추출하여 미생물 군집(community)의 다양성 및 특성을 연구하는 방법이다. Metagenomics를 활용하여 미생물 군집을 분석하는 방법으로는 계 통분석학적 마커 유전자(phylogenetic marker gene)를 이용하는 방 법과 whole genome shotgun sequencing을 이용하는 방법이 주로 이용된다. Phylogenetic marker 유전자를 이용한 접근법의 경우 일 반적으로 16S rRNA 유전자 분석을 이용하여 특정 환경 내의 미 생물 군집의 다양성 및 분포를 밝힐 수 있다. 또한 특정 환경 내 존재하는 모든 미생물의 유전물질을 대상으로 whole genome shotgun sequencing을 활용해 존재하는 유전물질의 기능과 역할을 규명하는 것이 가능하다(Langille 등, 2013; Shah 등, 2011). 이러 한 방법들을 활용할 경우 배양 가능한 미생물 뿐 아니라 난배양 성 미생물을 포함하여 신변종 미생물까지 동정하는 것이 가능하 기 때문에 기존 식중독 원인 규명 방법의 한계를 극복할 수 있 을 것으로 기대된다(Bergholz 등, 2014; Handelsman, 2004; Miller 등, 2013).
최근 건강에 관한 관심의 증대에 따라 샐러드 형태를 포함한 채소류의 소비가 증가하는 추세이다(Abadias 등, 2008; Jo 등, 2011). 채소류의 경우 가열 과정을 거치지 않고 그대로 섭취하는 경우가 많아 유통과정 상에서 미생물을 비롯한 각종 오염원에 의
†These authors contributed equally to this work as co-first authors.
*Corresponding author: Hee Gon Jeong, Ph. D., Department of Food Science and Technology, College of Agriculture and Life Sciences, Chungnam National University, 99 Daehak-ro, Yuseong-gu, Daejeon 305-764, Korea
Tel: +82-42-821-6726 E-mail: jeonghg@cnu.ac.kr
Received January 26, 2021; revised April 12, 2021; accepted May 10, 2021
해 문제가 발생할 가능성이 높기 때문에 안전성에 각별한 주의 가 요구된다(Jo 등, 2011; Shim 등, 2014). Metagenomics 기반의 방법을 이용하여 다양한 채소류에 존재하는 배양이 불가능한 미 생물들을 포함한 모든 상재균(microbiota) 군집의 특성에 대해 이 해한다면 식중독 사고 발생 예측과 관련한 정보를 얻을 수 있을 것이며, 나아가 식품 안전성의 향상에 기여할 수 있을 것으로 예 상된다.
오이(Cucumis sativus L.)는 박과(Cucurbitaceae)에 속하는 한해 살이 덩굴식물로 세계적으로 샐러드 등 여러 음식의 식재료로 많 이 소비되고 있으며, 국내에서도 연간 20만 톤 이상이 생산 및 소비되는 대표적인 채소류이다(Chang 등, 2014; Park 등, 2004). 국내에서는 다다기, 취정, 노각 등의 품종이 주로 재배되고 있으 며, 4월에서 6월 사이에 많이 생산되고 있다(KAMIS, 2018). 오 이는 노지 재배와 시설 재배가 동시에 이루어지고 있으며, 재배 환경의 온도, 습도 등 다양한 환경 요인에 의해서 차이가 발생할 수 있다(Ingraham, 1958; Kim 등, 2018; Turner 등, 2013). 2018 년을 기준으로 국내에서 생산된 약 39만 톤의 오이 중 시설재배 로 생산된 오이는 약 34만 톤으로 더 큰 비율을 차지하기에 시 설 재배로 생산된 오이의 상재균총 분석이 선행될 필요가 있다 (MAFRA, 2019). 오이는 다른 채소류와 마찬가지로 가열처리를 하지 않고 생식이나 샐러드의 형태로 섭취되고 있어 식중독과 같 은 문제가 발생할 가능성이 높아 각별한 주의가 요구된다. 또한, Salmonella enterica spp. (serotype Saint Paul, Oslo, Newport 등), Escherichia coli O157:H7 등의 식중독 발병 사례가 존재하며, 2014년부터 2018년까지 5년간 미국에서 보고된 오이 관련 식중 독 사례는 1,582건에 달하고 있어 안전성 측면에서 매우 주의가 필요한 식품이다(CDC, 2020). 본 연구에서는 한국에서 5월과 11월 두 시기에 걸쳐 국내 생 산량이 높은 지역인 천안, 진천, 공주에서 시설 재배를 통해 재 배된 오이를 수거하여 지역과 시기와 같은 환경적 요인을 다각 적으로 고려해 상재균의 군집 특성 및 잠재적 유해균의 분포를 분석하였다(KAMIS, 2018). 본 연구의 결과는 식중독 다발성 식 품인 채소류에 속하는 오이의 식품 재배 환경에 따른 식중독 모 델 식품으로서 오이의 식중독 원인 규명 및 발병 예측을 위한 새 로운 biomarker로서 활용 가능할 것이며, 다양한 채소류의 식품 안전성 향상에 중요한 기초자료로 이용될 수 있을 것으로 사료된다.
재료 및 실험방법
시료 준비 및 metagenomic DNA 추출 분석에 사용된 오이(Cucumis sativus L.)는 2016년 5월과 11월 두 시기에 천안, 진천, 공주에서 시설 재배되었다. 재배 당시 5월 의 평균 기온은 25.9±2.9oC (천안 25.3±2.9oC, 진천 26.6±3.0oC, 공주 25.7±2.7oC)이었고, 11월의 평균 기온은 11.7±4.8oC (천안 11.1±4.8oC, 진천 11.3±4.8oC, 공주 12.6±4.7oC) 이었다. 시설 내부 의 온도는 약 15oC로 유지하여 재배가 이루어 졌지만, 비교적 기 온이 높은 5월 주간에는 시설의 출입문을 개방한 채 오이의 재 배가 이루어졌다. 수확한 오이는 살균된 장갑과 용기를 이용하여 미생물의 오염을 차단한 상태로 4oC를 유지하여 실험실로 옮겨 졌다. 총 28개의 시료를 수집하였으며, 5월 공주 시료와 11월 진 천 시료는 각각 4개씩, 나머지 그룹은 각 5개 시료가 수집되어 분석되었다(총 시료수 28; 각 월별 시료수 14). 껍질과 과육이 골 고루 포함된 오이 25 g과 buffered peptone water (OXOID, Hampshire, UK) 225 mL를 filter bag (Labplas, Quebec, Canada) 에 담았으며, filter bag에 담긴 혼합물을 BagMixer 400 W(Intersceince, Saint Nom, France)를 이용하여 미생물을 탈리하였 고, 탈리액은 10분간 4oC에서 원심분리(10,000×g)하여 상등액은
버리고 남겨진 미생물 펠렛을 얻었다. 이후 얻어진 미생물 펠렛 에 5 mL의 TES buffer (0.1 M NaCl, 10 mM Tris-HCl, pH 8.0, 1 mM EDTA)를 첨가 후 혼합 과정을 거치고 원심분리 후 상등 액을 버리는 과정을 2번 거쳐 불순물을 제거하였다.
Metagenomic DNA는 기존의 DNA 추출법을 변형한 방법으로 미생물 펠렛에서 추출되었다(Jeon 등, 2019; Naravaneni과 Jamil, 2005; Yu 등, 2018). 미생물 펠렛에 500 μL의 PVP/CTAB buffer (1% polyvinylpyrrolidone, cetyltrimethylammonium bromide)와 50 μL의 lysozyme (100 mg/mL)를 첨가 후 37oC에서 1시간 동안 반
응시켜 미생물의 세포벽을 용해시키고 식물체에서 유래되는 폴 리페놀 및 다당류를 제거하였다. 반응이 완료된 미생물 혼합액은 −80oC에서 10분간 냉동 후 37oC에서 10분간 해동되었으며, 미생
물 혼합액에 200 μL의 proteinase K mixture [0.5 M EDTA 140 μL, proteinase K (20 mg/mL) 20 μL, 10% sodium dodecyl sulfate 40μL]를 첨가 후 56oC에서 1시간 동안 반응시켰다. 반응 이 완료된 미생물 혼합액을 상온에서 1분간 원심분리(21,206×g) 하여 상등액을 얻어낸 후, 100 μL의 5 M NaCl와 80 μL의 CTAB/ NaCl solution (NaCl 41 mg/mL, CTAB 100 mg/mL)를 첨가한 후 65oC에서 10분간 반응시켰다. 반응 후 상등액과 동일한 양의 phenol/chloroform/isoamyl alcohol [25:24:1 (v/v/v)]을 첨가 후 상 온에서 5분간 원심분리(21,206×g)하여 취한 상등액을 새로운 튜 브로 옮기고, 다시 상등액과 동일한 양의 chloroform을 첨가 후 상온에서 5분간 원심분리(21,206×g)하여 상등액을 취하였다. 취한 상등액에 3 μL의 RNase A (100 mg/mL)을 첨가 후 1시간 동안 37oC에서 반응시키고, 상등액 부피 10%의 3 M sodium acetate (pH 5.0)와 2배 부피의 차가운 ethanol을 첨가한 후 4oC에서 20 분간 원심분리(21,206×g)하였다. 이후 추가적인 불순물 제거를 위 해 상등액을 버리고 1 mL의 차가운 70% ethanol을 넣은 후 4oC 에서 5분간 원심분리(21,206×g)하였다. 상등액은 버리고 남아있을 수 있는 ethanol을 상온에서 건조시킨 후 DNA를 50 μL의 TE buffer에 용해시켰다. 이후 Optizen Nano Q (Mecasys, Deajeon, South Korea)을 이용하여 metagenomic DNA의 순도 및 농도를 측정하였고 −20oC에서 보관되었다.
16S rRNA 염기서열 분석 및 상재균총 군집 다양성 분석 16S rRNA 유전자 부분의 V5-V6 region을 증폭하는 프라이머 인 799F-mod6 CMGGATTAGATACCCKGGT-3')과 1114R (5'-GGGTTGCGCTCCTTGC-3')를 이용하여 분석을 진행하였으며, 엽 록체와 미토콘드리아의 polymerase chain reaction (PCR) 증폭이 최소화되도록 고안되었다 (Hanshew 등, 2013; Kumar 등, 2011). PCR을 통한 DNA 증폭은 PrimeSTAR HS DNA Polymerase (TAKARA, Shiga, Japan)을 이용하였으며, initial denaturation (98oC, 3분), denaturation (98oC, 10초), annealing (57oC, 15초), elongation (72oC, 30초), 30 cycle, final elongation (72oC, 3분)의 조건으로 진행되었다. PCR product는 MEGAquick-spin Plus kit (iNtRON, Seongnam, South Korea)를 이용하여 정제되었다. Index PCR은 Illumina Nextera XT Index kit (Illumina, San Diego, CA, USA)를 이용하였고 라이브러리는 제조사의 설명서에 따라 AMPure XP beads (Beckman Coulter, Indianapolis, IN, USA)에 의해서 정제되었다. 라이브러리의 크기 및 퀄리티는 Agilent Bioanalyzer 1000 chip (Agilent, Santa Clara, USA)과 KAPA qPCR kit (KAPA biosystems, Woburn, MA, USA)를 통해 확인 하였으며, 이후 Illumina Miseq system (Illumina, San Diego,
CA, USA)을 기반으로 pair-end sequencing (2×300 bp) 분석을 실 시하였으며, raw sequencing data는 National Center for Biotechnology Information (NCBI; https://www.ncbi.nlm.nih.gov/) 에 등록되었다(등록번호: PRJNA718715). 얻어진 염기서열은 MOTHUR software (ver. 1.38.1)를 통해 barcode 서열, 프라이머 서열, 낮은 품질(average quality score <25), 8번 이상 같은 염기 서열로 반복되는 부분(homopolymer)이 제거 되었으며, UCHIME 을 이용하여 chimeric 서열을 제거하였다(Edgar 등, 2011). 본 연 구에서는 세균 이외의 염기서열을 제거한 187,773 reads가 상재 균총 분석에 이용되었다. CLC Genomics Workbench (ver. 9.5.3, CLC bio, Aarhus, Denmark)를 이용해 97%의 유사도 기준으로 분 석하였으며, SILVA database (ver. 123, 80% confidence threshold) 를 통해 OTUs (operational taxonomic units)를 선정하였다. α-diversity 분석(observed OTUs, Chao 1 index, Shannon diversity index)의 경우 2,106 reads로 표준화(normalization)를 거친 후 시 료 간의 상재균총 군집 특성을 비교하는 방식으로 이루어졌다. β-diversity 분석을 위해 UniFrac distance의 기반의 principal coordinate analysis (PCoA)가 실시되었다.
Quantitative Real-Time Polymerase Chain reaction (qRT-PCR)을 이용한 시료 내 총균수 정량
총균수를 정량하기 위하여 qRT-PCR을 이용하였다. 16S rRNA 유전자를 증폭하는 프라이머를 사용하였고(Miseq platform 분석 과 동일) CFX Connect Optics Module (Bio-Rad, Hercules, CA, USA)기기와 KOD SYBR qPCR Mix (TOYOBO, Osaka, Japan) 를 통해 정량분석을 실시하였다. PCR 혼합액은 KOD SYBR qPCR Mix 10μL, 10 μM front & reverse primer 각 1 μL, DNA template 1μL와 증류수로 20 μL를 채워 PCR을 진행하였다. PCR 조건은 initial denaturation (98oC, 3분), denaturation (98oC, 10초),
annealing (57oC, 15초), elongation (68oC, 30초), 40 cycle, final elongation (68oC, 1분)으로 진행하였다. 시료 내 총균수 정량을 위 해 측정된 Ct value와 미리 얻어진 표준곡선(standard curve)을 이 용하였으며(Jeon 등, 2019; Yu 등, 2018), 자연환경에 주로 존재 하는 미생물의 평균 16S rRNA copy number (4.2)를 고려하여 계 산하였다(Větrovský과 Baldrian, 2013). E. coli의 genomic DNA를 log 수준의 연속희석법(serial dilution)으로 희석 후 qRT-PCR을 통 해 표준곡선을 작성하였다(regression coefficient, r2=0.9975).
통계적 분석
시료 간 통계적적인 수치 차이는 student t-test 또는
Mann-Whitney test를 통하여 확인하였다. 유의 확률이 0.05 이하(p-value <0.05)일 때 통계적으로 유의한 차이가 존재한다고 판단하였으며, Prism software(ver. 5.02)를 이용하였다.
결과 및 고찰
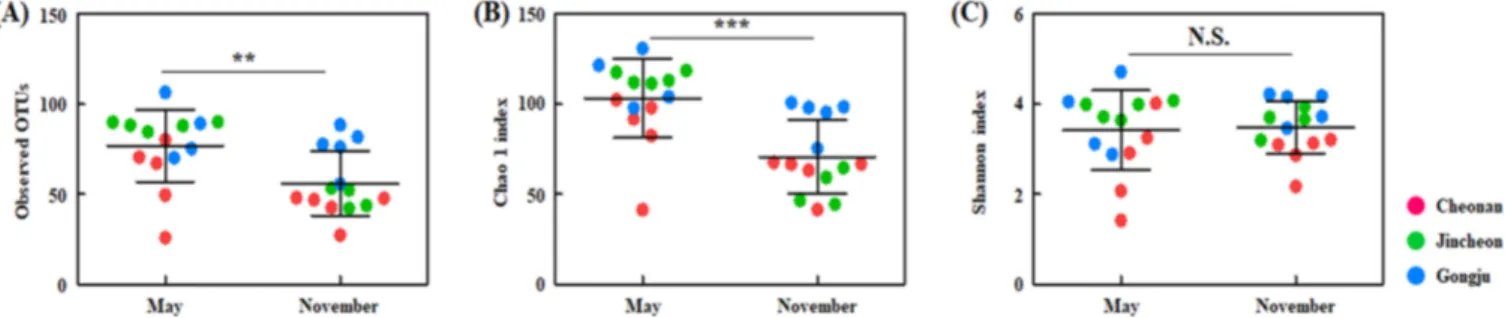
오이의 수확 시기 및 지역에 따른 상재균총 군집의 다양성 및 총균수 분석 국내에서 시설재배를 통해 재배된 오이 시료 내 상재균총 군 집의 다양성을 수확시기와 지역에 따라 비교하였으며, 단일 시료 내 상재균총 군집의 다양성을 나타내는 척도인 α-diversity를 Observed OTUs, Chao1 index와 Shannon index를 통하여 나타내 었다(Fig. 1). 수확 시기에 따른 Shannon index는 각각 5월 시료 에서 3.42±0.88과 11월 시료에서는 3.48±0.58로 통계적으로 유의 한 차이가 관찰되지 않았으나, Chao1 index는 5월과 11월 시료에 서 각각 103.06±21.91, 70.64±20.44로 5월 시료가 약 1.5배 더 높 게 나타났다(p<0.001). 또한 5월 시료에서 관찰된 OTUs는 76.82±20.02로 11월 시료의 55.99±17.89에 비해 약 1.4배 높게 나 타났다(p<0.01). Chao1 index는 미생물 abundance기반의 richness 평가 지표이며, Shannon index는 여기에 evenness의 요소가 포함 되어 있는 지표이다(Kim 등, 2017; Thukral, 2017). 따라서 5월 시 료의 Observed OTUs와 Chao1 index가 11월 시료에 비해 높았음 은 5월 오이의 상재균총이 더욱 다양했음을 의미한다. 수확 지역 간 비교를 하였을 때, Observed OTUs는 각각 천안, 진천, 공주에 서 50.64±17.61, 70.27±21.57, 80.07±14.12로 통계적으로 천안-진 천(p<0.05)과 천안-공주(p<0.001)에서 통계적으로 유의한 차이가 관찰되었으나, 진천-공주(p=0.27)에서는 차이가 관찰되지 않았다. Chao1 index는 각각 천안, 진천, 공주에서 72.18±21.39, 87.52± 32.67 102.48±15.87로 공주에서 천안보다 통계적으로 유의한 수 준으로 높게 나타났다(p<0.01). Shannon index도 2.79±0.73, 3.76± 0.27, 3.83±0.59로 천안-공주 그룹 비교 시 공주에서 통계적으로 유의한 수준에서 높게 나타났다(p<0.01). 진천과 공주 시료에 비 해 천안 시료의 상재균총의 다양성이 비교적 낮은 것을 확인할 수 있었으며, 상재균총의 다양성이 수확 지역에 따라 다를 수 있 음을 확인하였다. 상재균총의 다양성에는 일반적으로 온도를 비 롯해 습도, 토양 조건, 미생물간의 상호작용 등 다양한 환경적 요 인들이 작용할 수 있다(Ingraham, 1958; Kim 등, 2018; Turner 등, 2013). 본 연구에서 분석된 오이는 시설에서 재배되었지만, 5월의 시설 재배 환경과 11월의 시설 재배 환경은 온도를 포함하여 다 양한 환경적 차이가 존재하였다. 따라서 이러한 다양성 지수 차Fig. 1. Comparison of microbial diversity in the cucumber collected at each sampling time and site. The microbial diversity from each cucumber sample is estimated by Chao1 diversity and Shannon diversity indices. Shapes indicate each sampling region [Cheonan (red), Jincheon (green), Gongju (blue))]. The midline signifies mean and error bar indicates the standard deviation (SD). The x-axis indicates different sampling time and site. The y-axis indicates the number of OTUs (A), Chao1 index (B) and Shannon index (C). The asterisk signifies statistical difference between groups (*p<0.05, **p<0.01, ***p<0.001). N.S. means that there is not significant difference between groups.
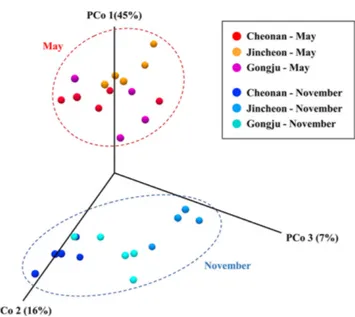
이는 재배 시기의 차이에 기인하였을 것으로 사료된다. 시료 내 총균수는 qRT-PCR을 통해 16S rRNA 유전자 염기서 열을 증폭하여 정량분석 하였다(Fig. 2). qRT-PCR을 비롯한 PCR 기반의 분석법은 생균 뿐 아니라 세포 사멸 후 잔존하는 DNA 도 함께 검출 될 수 있다는 한계가 존재한다(Young 등, 2007). 하 지만, 배양 기반의 분석법의 경우 난배양성 미생물의 검출이 어 렵고(Bergholz 등, 2014; Handelsman, 2004; Miller 등, 2013), 16S rRNA 유전자 염기서열 분석을 통한 상재균총 분석에도 사 균에 대한 분석이 포함될 수 있다. 게다가 원료식품에서 사균의 DNA가 전체 결과에 영향을 줄 정도의 양이 오랜 시간 존재하기 어렵기에 qRT-PCR을 총균수 정량에 활용하였다. qRT-PCR은 짧 은 시간에 균의 정량이 가능하다는 이점이 있어 많은 연구에서 세균수 정량에 이용되고 있다(Kralik과 Ricchi, 2017). 총균수를 수 확 시기에 따라 비교하였을 때, 5월 시료의 평균 총균수는 약 7.37×104 cell/g였고, 11월은 약 4.21×105 cell/g으로 수확 시기에 따른 통계적 차이가 존재하지 않았으며, 진천 지역을 제외하고는 같은 지역 내 시기적 차이가 관찰되지 않았다. 수확 지역에 따라 비교하였을 때에도 천안(5.77×105 cell/g), 진천(8.42×104 cell/g), 공 주(8.28×104 cell/g)간 지역적 차이는 관찰되지 않았기에 오이 시 료내 총균수는 시기 및 지역에 따른 환경적 요인에 대한 영향이 적은 것으로 판단된다. 오이 시료내 총균수는 104~105 cell/g 수준 으로, 선행연구에서 상추와 깻잎의 상재균총 수가 106~108 cell/g 수준이었던 것과 비교하였을 때 비교적 낮은 수준으로 관찰되었 다(Jeon 등, 2019; Yu 등, 2018). 채소류 표면에 존재하는 미생물 의 생장에 영향을 미치는 요인에는 식물 표면의 친수성, 기공의 수, 기공의 크기, wax 농도 등 이화학적 요인들이 있음이 밝혀진 바 있으며(Lu 등, 2015), 오이에 존재하는 미생물의 총균수에는 오이 표면의 식물로서의 특징이 반영 되었을 것으로 보여진다. PCoA 분석을 통한 수확 시기에 따른 상재균총 분석 16S rRNA 유전자 염기서열간 계통분류학적 유사도를 고려한 unweighted UniFrac distance을 기반으로 하는 PCoA 분석을 통해
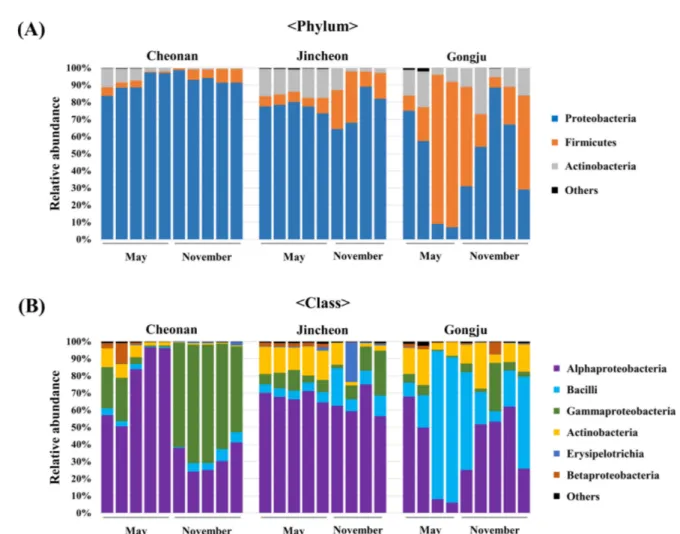
수확 시기에 따른 오이 내 상재균총 군집 특성의 차이를 나타내 었다(Fig. 3). Unweighted UniFrac 분석은 OTUs 간 계통분류학적 유사도를 고려한 방법이며, Weighted UniFrac 분석은 OTUs 간 계통학적 거리와 풍부도(abundance)을 함께 고려하여 군집구조의 차이를 시각화한 방법이다(Lozupone 등, 2007). Unweighted UniFrac distance 기반의 PCoA 분석을 통해 5월 시료 내 상재균 총 군집과 11월 시료 내 상재균총 군집의 클러스터가 뚜렷하게 나뉘는 것을 확인할 수 있으며, 이를 통해 수확 시기에 따른 상 재균총의 군집 구성에 있어 특성의 차이가 존재함을 확인할 수 있었다. 오이 상재균총의 Phylum (문)과 Class (강) 분포 분석 PCoA 분석 결과 나타난 수확 시기에 따른 오이 내 상재균총 의 군집 특성 차이를 분류학적으로 규명하기 위해 수집된 오이 의 OTUs로부터 수확 시기 혹은 지역에 따른 상재균총의 군집 분포를 Phylum과 Class 수준에서 분석 및 비교하였다(Fig. 4).
수확 시기에 따른 차이를 분석한 결과, Phylum 수준에서는 모 든 5월과 11월 시료에서 Proteobacteria, Firmicutes, Actinobacteria 가 우점하는 Phylum으로서 발견되었다(Fig. 4A). 이들은 모두 선 행 연구에서 오이를 포함한 여러 식물체에서 흔히 발견되었다고 보고된 바 있다(Kim 등, 2018; Turner 등, 2013; Williams과 Marco, 2014). Proteobacteria가 차지하는 비율의 경우 5월 시료는 70.86±28.48%, 11월 시료는 74.50±23.12% 였으며(p=0.7129), Firmicutes는 5월 시료에서 17.60±29.36%, 11월 시료에서 18.93± 17.93%였다(p=0.8862). Actinobacteria의 경우 5월에 시료에서는 11.14±6.06%, 11월 시료에서는 6.66±7.94%가 존재하였다(p= 0.1051). 이러한 결과는 PCoA분석에서 나타난 것과는 다르게 수 확 시기에 따른 상재균총 조성의 차이가 Phylum 수준에서는 드 러나고 있지 않음을 의미한다.
Class 수준에서는 Proteobacteria에 속하는 Alphaproteobacteria, Gammaproteobacteria와 Firmicutes에 속하는 Bacilli 그리고 Fig. 2. Comparison of total bacterial loads in the cucumber
according to sampling time and site. The quantity of total bacteria (log CFU/g) was measured using quantitative real-time PCR. The asterisk signifies statistical difference between groups (*p<0.05, **p<0.01, ***p<0.001). Error bar indicates the standard deviation (SD).
Fig. 3. UniFrac-based principal coordinate analysis (PCoA) according to sampling times and sites. Sampling sites are represented as color in the box. Dot circles mean cluster according to sampling time. The percentage contributions to the variance of the data from principal components 1,2 and 3 (PCo 1, PCo 2, PCo 3) are listed along axes representing them.
Actinobacteria가 주로 발견되었다(Fig. 4B). 선행 연구에서 Alphaproteobacteria는 식물 뿐만 아니라 다양한 자연환경에서 빈 번하게 발견되었다는 보고가 있으며(Pérez-García 등, 2011), Gammaproteobacteria도 식물체에서 빈번하게 보고되고 있다 (Compant 등, 2011; Jeon 등, 2019; Lopez-Velasco 등, 2011; Yu 등, 2018). Bacilli는 토양에 유래하는 미생물들이 많이 속해있을 뿐만 아니라, 농업분야에서 많은 이점을 가지고 있는 것들이 많 기 때문에 발견되었을 것으로 사료된다(Pérez-García 등, 2011). Class 수준에서의 수확 시기에 따른 차이는 Gammaproteobacteria 에서 관찰되었다. 5월 시료에서 Gammaproteobacteria가 차지하는 비율은 7.47±7.96%였던 반면, 11월 시료에서는 28.79±27.25%로 유의하게 높은 수준을 보였다(p<0.001). 질소 첨가는 토양의 상재 균총에 영향을 줄 수 있고 Gammaproteobacteria를 비롯한 여러 미생물의 증식을 촉진한다는 연구결과가 존재하며(Zhang 등, 2014), 토양의 질소 함량은 토양의 온도와 반비례하는 것으로 알려져 있 다(Kirschbaum, 1995). 이를 통해 질소 함량과 같은 토양의 특성 이 수확시기에 따른 기온에 의해 영향을 받았고, 토양의 상재균 총과 식물체의 상재균총은 밀접한 관련이 있기 때문에 토양의 상 재균총이 식물체의 상재균총에 영향을 준 것으로 보이며(Turner 등, 2013), 오이의 상재균총에서 Gammaproteobacteira는 수확 시 기에 따른 차이가 관찰된 것으로 예상된다. 재배 지역에 따른 군집 구성의 차이는 phylum과 class 수준에 서 명확히 드러났다. Phylum 수준에서 Proteobacteria의 경우 천 안 시료에서 92.40±4.62%, 진천 시료에서 77.00±7.43%, 공주 시 료에서 46.44±28.84%가 검출 되었으며 모든 지역간 유의적 차이 가 관찰되었다(p<0.01). Firmicutes의 비율 역시 천안 시료에서 4.14±2.79%, 진천 시료에서 12.11±8.87%, 공주 시료에서 40.11± 31.75%로 지역간 차이가 존재하였다(p<0.05). Class 수준에서 Alphaproteobacteria의 경우 천안 시료에서는 54.20±28.44%, 진천 시료에서는 65.78±6.00%, 공주 시료에서는 38.78±22.95%로 존재 하였으며 진천과 공주 시료 사이에서 유의한 차이를 보였다 (p<0.01). Gammaproteobacteria의 경우 천안 시료에서는 36.41± 28.72%, 진천 시료에서는 9.78±7.12%, 공주에서는 6.16±8.42%로 검출되었으며, 천안과 진천 시료(p<0.05) 그리고 천안과 공주 시 료(p<0.01) 간에 유의적인 차이가 존재하였다. Bacilli의 경우 천 안 시료에서는 3.38±2.29%, 진천 시료에서는 8.33±5.61%, 공주 시료에서는 39.44±31.71%로 검출되었으며, 세 지역간 모두 유의 적인 차이가 존재하였다(p<0.05). Actinobacteria의 경우 천안 시료 에서는 3.32±3.86%, 진천 시료에서는 10.89±6.58%, 공주 시료에 서는 13.11±7.51%로 검출 되었으며, 천안과 진천 시료(p<0.01), 그리고 천안과 공주 시료(p<0.01) 사이에 통계적으로 유의적 차 이가 존재하였다. 이러한 결과는 일반적으로 기온 등 미생물 상 재균총 군집에 영향을 주는 요인들보다 농업용수, 토양, 재배 방 법 등 재배 환경의 차이에 기인 하는 것으로 보여진다(Cruz-Martínez 등, 2009; Frenk 등, 2014; Kirschbaum, 1995).
Fig. 4. Comparison of taxonomic composition at phylum and class level. The legend only showed the phyla (A) and classes (B) that found more than 1% in at least one sample.
Genus (속) 수준에서의 상재균총 군집 특성 및 잠재적 유해 균 분석
Spearman’s rank correlation 기반의 heat map을 통해 상재균총 군집 특성을 genus 수준에서 분석하였으며, 하나의 시료 이상에 서 0.5% 이상인 genus만을 포함하였다(Fig. 5A). Methylobacterium (5월 시료 26.71±12.65%, 11월 시료 12.94±16.65%)와 Sphingomonas (5월 시료 8.35%±4.75%, 11월 시료 10.07±5.48%)의 경우는 두 수 확시기에서 모두 우점하고 있었으며, 다양한 자연 환경 및 식물 체에서 자주 발견되는 genus로 보고되고 있다(Innerebner 등, 2011). Methylobacterium은 발아 촉진 및 질소 고정화 등의 작용 을 통하여 식물체의 성장을 촉진한다고 알려져 있고(Madhaiyan 등, 2006), Sphingomonas의 경우 식물 질병에 대한 증상을 완화 시키고 식물의 성장에 악영향을 미칠 수 있는 균의 성장을 억제 하여 식물 생육에 도움을 주는 것으로 알려져 있다(Innerebner 등, 2011). 선행연구에 따르면 미국에서 유통되는 다양한 품종(Long English cucumber, Mini cucumber 등)의 오이 상재균총에서 Rhizobium, Pseudomonas, Pantoea, Stenotrophomonas, Acinetobacter 등의 genus가 우점하는 것으로 보고되었다(Pérez-Díaz 등, 2019). 본 연구 결과에서는 Rhizobium 및 Pseudomonas를 포함하여 다양 한 genus들이 공통적으로 발견되었는데, 이는 metagenomic sequencing data의 분석과정에서의 차이를 감안하더라도 매우 유 사한 결과로 오이는 품종이나 국가간 재배 방식 등 다양한 영향 에 비해 세균 군집의 구성에 차이가 크게 드러나지 않음을 의미 한다. 분석 결과를 통해 오이의 전체적인 상재균총 군집 특성은 수 확 시기와 더 밀접한 관계를 가지는 것을 확인할 수 있었다(Fig. 5A). 수확 시기에 따른 차이가 존재하는 genus group을 heat map 에서 두 그룹(I, II)으로 표시하였다. 그룹 I은 11월 시료에서 더 높은 비율로 존재하는 genus를, 그룹 II는 5월 시료에서 더 높은 비율로 존재하는 genus를 나타낸다. 그룹 I의 경우 Enterococcus (5월 시료 0.95±1.37%, 11월 시료 8.46±6.97%, p<0.001), Pseudomonas (5월 시료 4.24±6.22%, 11월 시료 14.49±16.03%, p<0.05), Rhizobium (5월 시료 0.57±0.73%, 11월 시료 9.84±11.69%, p<0.01)이 수확 시기에 따른 차이를 보 였다. Rhizobium은 질소 고정화 등의 작용으로 식물이 성장하는 데 좋은 영향을 주는 것으로 알려져 있으며, 공생을 통해 식물체 가 낮은 온도에서 적응할 수 있도록 작용한다고 보고된 바 있는 genus이다(Liu 등, 2019). 일부 Rhizobium은 0~15oC의 낮은 온도 에서도 증식이 가능하다고 알려져 있어 11월 시료에서 높은 비 율을 차지할 수 있었던 것으로 보여진다(Drouin 등, 2000). Enterococcus는 주로 인간의 장이나 다양한 자연환경에서 주로 관 찰되며 E. faecium와 E. faecalis는 심내막염(endocardit)을 비롯한 심각한 질병의 감염을 일으키는 것으로 알려져 있다(Murray, 1990). Enterococcus는 추위에 내성을 가져 낮은 온도에서도 생존 및 증 식이 가능하다고 보고된 바 있어 11월 시료에서 더 높은 비율을 차지할 수 있었던 것으로 예상된다(Panoff 등, 1997). Pseudomonas 도 토양을 비롯한 자연계에 널리 존재한다고 알려져 있으나(Filip 과 Hermann, 2001), 낮은 온도로 인한 생리적 변화가 적은 것으 로 알려져 있어 기온이 낮은 11월에 더 높은 비율로 존재하였던 것으로 생각된다(Fonseca 등, 2011; Yabuuchi 등, 1993). 폐렴, 패 혈증, 요로감염 등의 원인이 되는 P. aeruginosa와 같이 병원성을 가지는 균주가 속할 수 있으며(Stover 등, 2000), P. aeruginosa는 우유(Chen 등, 2011), 육류 및 어류(Geornaras 등, 1999), 각종 채 소 및 샐러드(de Quadros Rodrigues 등, 2014; Xu 등, 2019)에서 도 빈번하게 관찰되기 때문에 주의가 필요하다. 그룹 II에서는 Aureimonas (5월 시료 22.26±20.96%, 11월 시료 6.09±8.68, p<0.01), Escherichia (5월 시료 0.32±0.31%, 11월 시료 0.05±0.08%, p<0.01), Microbacterium (5월 시료 8.69±5.48%, 11 월 시료 1.39±1.81%, p<0.001)이 수확시기에 따른 차이를 보였다. Aureimonas는 오이 외에 다른 식물체에서도 발견된 사례가 있으 며(Aydogan 등, 2016), 10~40oC의 비교적 다양한 범위의 온도에 서 증식이 가능하고 고온에 강하다고 알려져 있어 5월 샘플의 상 재균총에서 높은 비율을 차지하는 것으로 보여진다(Schröttner 등, 2014). Aureimonas에 속하는 A. altamirensis는 낭포성 섬유증 (cystic fibrosis), 췌장암, 각막염(keratitis), 흉수(pleural diffusion), 복막염(peritonitis) 등 다양한 환자에게서 발견된 사례가 있다 (Eshaghi 등, 2015; Schröttner 등, 2014). Escherichia는 다른 채소 류의 상재균총 분석들과는 달리 오이에서는 그룹 II에 속하는 것 으로 나타났다(Jeon 등, 2019; Yu 등, 2018). Escherichia는 온혈 동물의 장내에서 주로 발견되기에 분변오염의 지표생물로 이용 되며(Jang 등, 2017), Enterohemorrhagic E. coli (EHEC), Enterotoxigenic E. coli (ETEC) 등과 같은 병원성 Escherichia도 존재하기에 주의 가 필요한 genus다(Gaastra과 Svennerholm, 1996; Sperandio과 Nguyen, 2012). Escherichia의 최적의 성장 조건은 약 35~37oC로 알려져 있고 일정 범위 내에서 온도가 높을수록 증식속도가 증 가한다고 보고된 바 있어(Bronikowski 등, 2001), 비교적 높은 기 온에서 재배된 5월 시료에서 더 높은 비율을 차지할 수 있었던 것으로 보인다. Escherichia는 다른 식물체의 상재균총을 분석한 선행연구에서도 관찰되었지만 오이의 상재균총과는 달리 수확 시 기 및 지역에 따른 Escherichia의 차이가 관찰되지 않았다(Jeon 등, 2019; Yu 등, 2018). 각 식물체가 가지는 친수성, 기공, wax 농도 등의 이화학적 요인이 미생물의 생식에 영향을 줄 수 있으 며(Lu 등, 2015), 오이 표면의 식물체로서의 특성이 영향을 준 것 으로 예상된다. Microbacterium도 오이를 포함한 다양한 식물체 에서 발견된 사례가 존재하고 식물의 생장을 촉진시키고 유해미 생물의 억제하는 능력을 있음이 보고된 바 있다(Cordovez 등, 2018; Kim 등, 2011; Williams과 Marco, 2014). 이에 속하는 일부 균주는 강한 열 저항성을 가지고 있다고 알려져 있어 비교적 온 도가 온화한 5월에 높은 비율을 차지한 것으로 예상된다(Nakata, 2000). 이러한 결과는 오이가 가지는 생물학적 위험 요인은 수확 시기에 따라 상이할 수 있음을 의미한다. 본 연구의 결과를 오이의 상재균총을 분석한 다른 선행연구와 비교해 보았을 때, 오이의 상재균총을 구성하는 genus가 유사함 을 알 수 있었다. 미국에서 유통되는 다양한 품종(Long English cucumber, Mini cucumber 등)의 오이 상재균총에서 Rhizobium, Pseudomonas, Pantoea, Stenotrophomonas, Acinetobacter 등의 genus가 우점하는 것으로 보고되었고(Pérez-Díaz 등, 2019), 일본 에서 재배된 오이 줄기의 상재균총에서도 Methylobacterium, Microbacterium, Pseudomonas, Rhizobium, Sphingomonas 등이 존 재함이 보고되었다(Mahmood 등, 2019). 본 연구의 결과에서도 Methylobacterium, Pseudomonas, Rhizobium 등 다양한 Genus가 공통적으로 상재균총을 구성하고 있었으며, 오이는 품종이나 국 가간 재배 방식 등 다양한 영향에 비해 세균 군집의 구성에는 차 이가 크게 드러나지 않는 것으로 보여진다.
앞서 언급된 Aureimonas, Enterococcus, Escherichia, Pseudomonas 이외에도 여러 잠재적 유해 genus가 관찰되었다(Fig 5B). Staphylococcus (5월 시료 13.17±26.39%, 11월 시료 6.65±10.89%) 는 대표적인 식중독균인 Staphylococcus aureus를 포함할 수 있으 며, 사람에서 식품으로 이동 가능한 것으로 알려져 있기에 주의 를 필요로 한다(Le Loir 등, 2003). 수확시기에 따른
Staphylococcus의 비율의 차이는 통계적으로 유의하지 않았지만, 다른 지역과 달리 공주지역에서(천안 0.63±0.58%, 진천 1.13± 1.03%, 공주 29.00±29.29%) 높은 비율로 존재하는 것이 관찰되었 다. 이는 토양, 농업용수, 농업기구 또는 작업자를 포함한 재배환 경으로 인한 영향으로 예측되며, 오이의 생산 및 유통 과정에서 주의가 필요함을 의미한다. Acinetobacter (5월 시료 1.27±2.29%, 11월 시료 0.32±0.35%), Aerococcus (5월 시료 0.38±0.70%, 11월 시료 0.15±0.26%), Enterobacter (5월 시료 0.66±1.28%, 11월 시 료 0.39±0.57%), Pantoea (5월 시료 0.09±0.09%, 11월 시료 1.33±3.20%) 등도 오이의 상재균총에서 관찰되었다(Fig. 5B). Acinetobacter baumannii와 A. lwoffi (Antunes 등, 2014; Rathinavelu 등, 2003), Aerococcus urinae, Aero. sanguinicola (Rasmussen, 2016), Enterobacter sakazakii, En. aerogenes (Davin-Regli, 2015; Iversen과 Forsythe, 2003), Pantoea agglomerans (Cruz 등, 2007; Dutkiewicz 등, 2016) 등의 병원성 세균이 속하는 것으로 알려져 있으나, 이 중 식품에서 발견될 가 능성이 있는 세균에 대한 추가적인 연구는 계속 진행되어야 할 것이다. 이러한 정보들은 식물성 식품의 유통 또는 조리 단계에 서의 식중독균 오염 뿐만 아니라, 생산 단계에서 식품에 상재하 고 있는 잠재적 유해균이 직접적인 식중독의 원인이 될 수 있다 는 가능성을 검증하는데 중요한 자료로 활용될 수 있을 것이다 (Berger 등, 2009; Kisluk과 Yaron, 2012; Shaw 등, 2011; Xicohtencatl-Cortes 등, 2009).
오이에서 주로 존재하는 난배양성 미생물로는 Sphingomonadales AKIW852 uncultured bacterium (1.17±20.45%), Enterobacteriaceae uncultured bacterium (0.20±0.51%), Staphylococcaceae uncultured bacterium (0.12±0.20%), Gammaproteobacteria B38 uncultured bacterium (0.11±0.17%), Micrococcaceae uncultured bacterium
(0.04±0.09%) 등이 확인되었다. 특히 Enterobacteriaceae uncultured bacterium은 선행 연구결과 다양한 식물성 식품에서 발견된 바 있 다(Jeon 등, 2019; Yu 등, 2018). Enterobacteriaceae uncultured bacterium은 Enterobacteriaceae family에 속하며 장내에서 주로 발 견된다(Nordmann 등, 2011). Escherichia, Enterobacter, Enterococcus, Klebsiella 등 잘 알려진 유해 genus가 포함되어 있기 때문에, 오 이를 포함한 여러 식물성 식품에서 이러한 난배양성 미생물의 생 리적 특징 규명, 배양기술개발 및 병원성 확인과 같은 추가적인 연구가 필요할 것이다. 채소류의 소비가 증가하면서 비가열 섭취로 인한 식중독 발생 을 예방하기 위해 효율적인 채소류 관리방법 및 식중독균의 제 어방안이 필요하다. 이를 위해서 상재균총 군집 특성에 대한 이 해가 선행되어야 한다. 본 연구에서는 상재균총의 다양성, 총균 수, PCoA 분석, 잠재적 유해균 분석 등 상재균총 군집 특성을 다각적 측면에서 분석하였다. 또한 오이에 잠재적 유해 genus가 상재함을 확인하였으며, 그 중 식중독과 밀접한 연관이 있을 수 있는 Escherichia와 Staphylococcus의 분포 특성을 확인하였다. 향 후 종(species) 이상의 분류학적 기준에 대한 연구가 진행되어야 할 것이다. 이러한 결과는 상재균총 군집 특성에 대한 정보와 함 께 환경적 요인에 따른 유해 미생물의 분포 양상 및 난배양성 미 생물에 대한 정보를 제시하여 다양한 식중독의 원인 규명을 위 한 기초자료로 사용될 수 있을 것으로 사료되며, 채소류 안전성 및 위생 수준을 향상시키는데 기여할 수 있을 것으로 기대된다.
요
약
본 연구에서는 16S rRNA 염기서열 분석을 통하여 시설재배 오이 내 상재균총 군집 특성을 분석하였으며, 수확 시기 및 지역 Fig. 5. Analysis of microbiota composition in cucumber at the genus level. (A) The tendency of relative abundance at the genus level was represented through Spearman’s rank correlation coefficient-based Heat map (Ch-Cheonan, J-JIncheon, G-Gongju). Colors mean proportion of each genus (>0.5% at least in one sample). (B) The relative abundances of potential pathogenic genera were compared according to sampling time. The asterisk signifies statistical difference between each groups (*p<0.05, **p<0.01, ***p<0.001).에 따른 상재균총에 대한 정보를 제공하였다. 상재균총 다양성 분석(α-diversity)의 경우 5월 시료에서 더 높은 수치의 Observed OTUs와 Chao1 index가 나타났다. PCoA (β-diversity)분석을 통해 서 수확 시기에 따른 상재균총의 차이가 존재함을 확인하였다. Phylum 수준에서는 Proteobacteria, Firmicutes, Actinobacteria가 우 점하였고, class 수준에서는 Gammaproteobacteria, Bacilli, Alphaproteobacteria, Actinobacteria가 주로 존재하였다. Genus 수 준에서는 시기적인 요인이 주로 상재균총에 영향을 끼치는 것을 확인할 수 있었으며, 일부 지역적 요인의 영향도 관찰 되었다. 5 월 시료에서는 Aureimonas, Escherichia, Microbacterium이 11월 시료에서는 Enterococcus, Pseudomonas, Rhizobium이 더 높은 비 율을 차지하였다. 이외에도, Acinetobacter, Aerococcus, Aureimonas, Enterobacter, Enterococcus, Escherichia, Pantoea, Pseudomonas, Staphylococcus와 같이 잠재적인 위험성을 가지는 genus가 존재함 을 확인하였다.
감사의 글
이 연구는 충남대학교 학술연구비의 지원을 받아 수행됨.
References
Abadias M, Usall J, Anguera M, Solsona C, Viñas I. Microbiological quality of fresh, minimally-processed fruit and vegetables, and sprouts from retail establishments. Int. J. Food Microbiol. 123: 121-129 (2008)
Antunes L, Visca P, Towner KJ. Acinetobacter baumannii: evolution of a global pathogen. Pathog. Dis. 71: 292-301 (2014)
Aydogan EL, Busse HJ, Moser G, Müller C, Kämpfer P, Glaeser SP. Aureimonas galii sp. nov. and Aureimonas pseudogalii sp. nov. isolated from the phyllosphere of Galium album. Int. J. Syst. Evol. Microbiol. 66: 3345-3354 (2016)
Berger CN, Shaw RK, Brown DJ, Mather H, Clare S, Dougan G, Pallen MJ, Frankel G. Interaction of Salmonella enterica with basil and other salad leaves. ISME J. 3: 261-265 (2009)
Bergholz TM, Switt AIM, Wiedmann M. Omics approaches in food safety: fulfilling the promise? Trends Microbiol. 22: 275-281 (2014)
Bronikowski AM, Bennett AF, Lenski RE. Evolutionary adaptation to temperature. VIII. Effects of temperature on growth rate in natu-ral isolates of Escherichia coli and Salmonella enterica from dif-ferent thermal environments. Evolution 55: 33-40 (2001)
CDC. Annual Summaries of Foodborne Outbreaks. Available from: https://www.cdc.gov/fdoss/annual-reports/index.html. Accessed Nov. 11, 2019.
CDC. National Outbreak Reporting System (NORS). Available from: https://wwwn.cdc.gov/norsdashboard. Accessed Dec. 7, 2020. Chang YH, Choo JH, Lee SY, Kim TY, Jin MH, Chang MY, Lee
SH, Lee CK, Park SG. Inhibition of melanogenesis by cucurbita-cin B from Cucumis sativus L. J. Soc. Cosmet. Scientists Korea 40: 403-412 (2014)
Chen TR, Wei QK, Chen YJ. Pseudomonas spp. and Hafnia alvei growth in UHT milk at cold storage. Food Control 22: 697-701 (2011)
Compant S, Mitter B, Colli-Mull JG, Gangl H, Sessitsch A. Endo-phytes of grapevine flowers, berries, and seeds: identification of cultivable bacteria, comparison with other plant parts, and visual-ization of niches of colonvisual-ization. Microb. Ecol. 62: 188-197 (2011)
Cordovez V, Schop S, Hordijk K, de Boulois HD, Coppens F, Hans-sen I, Raaijmakers JM, Carrion VJ. Priming of plant growth pro-motion by volatiles of root-associated Microbacterium spp. Appl. Environ. Microbiol. 84: e01865-18 (2018)
Cruz-Martínez K, Suttle KB, Brodie EL, Power ME, Andersen GL, Banfield JF. Despite strong seasonal responses, soil microbial
consortia are more resilient to long-term changes in rainfall than overlying grassland. ISME J. 3: 738-744 (2009)
Cruz AT, Cazacu AC, Allen CH. Pantoea agglomerans, a plant pathogen causing human disease. J. Clin. Microbiol. 45: 1989-1992 (2007)
Davin-Regli A. Enterobacter aerogenes and Enterobacter cloacae; versatile bacterial pathogens confronting antibiotic treatment. Front. Microbiol. 6: 392-401 (2015)
de Quadros Rodrigues R, Loiko MR, de Paula CMD, Hessel CT, Jacxsens L, Uyttendaele M, Bender RJ, Tondo EC. Microbiologi-cal contamination linked to implementation of good agricultural practices in the production of organic lettuce in Southern Brazil. Food Control 42: 152-164 (2014)
Drouin P, Prëvost D, Antoun H. Physiological adaptation to low tem-peratures of strains of Rhizobium leguminosarum bv. viciae asso-ciated with Lathyrus spp. FEMS Microbiol. Ecol. 32: 111-120 (2000)
Dutkiewicz J, Mackiewicz B, Lemieszek MK, Golec M, Milanowski J. Pantoea agglomerans: a mysterious bacterium of evil and good. Part III. Deleterious effects: infections of humans, animals and plants. Ann. Agric. Environ. Med. 23: 197-205 (2016)
Edgar RC, Haas BJ, Clemente JC, Quince C, Knight R. UCHIME improves sensitivity and speed of chimera detection. Bioinform. 27: 2194-2200 (2011)
Eshaghi A, Shahinas D, Patel SN, Kus JV. First draft genome sequence of Aureimonas altamirensis, isolated from patient blood culture. FEMS Microbiol. Lett. 362: (2015)
Filip Z, Hermann S. An attempt to differentiate Pseudomonas spp. and other soil bacteria by FT-IR spectroscopy. Eur. J. Soil Biol. 37: 137-143 (2001)
Fonseca P, Moreno R, Rojo F. Growth of Pseudomonas putida at low temperature: global transcriptomic and proteomic analyses. Environ. Microbiol. Rep. 3: 329-339 (2011)
Freiberg C, Fellay R, Bairoch A, Broughton WJ, Rosenthal A, Perret X. Molecular basis of symbiosis between Rhizobium and legumes. Nature 387: 394-401 (1997)
Frenk S, Hadar Y, Minz D. Resilience of soil bacterial community to irrigation with water of different qualities under M editerranean climate. Environ. Microbiol. 16: 559-569 (2014)
Gaastra W, Svennerholm AM. Colonization factors of human entero-toxigenic Escherichia coli (ETEC). Trends Microbiol. 4: 444-452 (1996)
Geornaras I, Kunene NF, von Holy A, Hastings JW. Amplified frag-ment length polymorphism fingerprinting of Pseudomonas strains from a poultry processing plant. Appl. Environ. Microbiol. 65: 3828-3833 (1999)
Handelsman J. Metagenomics: application of genomics to uncultured microorganisms. Microbiol. Mol. Biol. Rev. 68: 669-685 (2004) Hanshew AS, Mason CJ, Raffa KF, Currie CR. Minimization of
chloroplast contamination in 16S rRNA gene pyrosequencing of insect herbivore bacterial communities. J. Microbiol. Methods 95: 149-155 (2013)
Ingraham JL. Growth of psychrophilic bacteria. J. Bacteriol. 76: 75-80 (1958)
Innerebner G, Knief C, Vorholt JA. Protection of Arabidopsis thaliana against leaf-pathogenic Pseudomonas syringae by Sphingomonas strains in a controlled model system. Appl. Environ. Microbiol. 77: 3202-3210 (2011)
Iversen C, Forsythe S. Risk profile of Enterobacter sakazakii, an emergent pathogen associated with infant milk formula. Trends Food Sci. Technol. 14: 443-454 (2003)
Jang JH, Hur HG, Sadowsky MJ, Byappanahalli MN, Yan T, Ishii S. Environmental Escherichia coli: ecology and public health impli-cations-a review. J. Appl. Microbiol. 123: 570-581 (2017)
Jeon DY, Yum SJ, Seo DW, Kim SM, Jeong HG. Leaf-associated microbiota on perilla (Perilla frutescens var. frutescens) cultivated in South Korea to detect the potential risk of food poisoning. Food Res. Int. 126: 108664 (2019)
Jo MJ, Jeong AR, Kim HJ, Lee NR, Oh SW, Kim YJ, Chun HS, Koo MS. Microbiological quality of fresh-cut produce and organic vegetables. Korean J. Food Sci. Technol. 43: 91-97 (2011)
KAMIS. Cool and delicious summer vegetables (Cucumber). Avail-able from: https://www.kamis.or.kr/customer/trend/product/prod-uct.do?action=detail&brdctsno=426245&pagenum=1&search_optio n=SUBJECT&search_keyword=%EC%98%A4%EC%9D%B4&. Accessed Jun. 21, 2018.
Kim BR, Shin JW, Guevarra RB, Lee JH, Kim DW, Seol KH, Lee JH, Kim HB, Isaacson RE. Deciphering diversity indices for a better understanding of microbial communities. J. Microbiol. Bio-technol. 27: 2089-2093 (2017)
Kim DH, Hong SH, Kim YT, Ryu SY, Kim HB, Lee JH. Metage-nomic approach to identifying foodborne pathogens on Chinese cabbage. J. Microbiol. Biotechnol. 28: 227-235 (2018)
Kim BY, Weon HY, Park IC, Lee SY, Kim WG, Song JK. Microbial diversity and community analysis in lettuce or cucumber culti-vated greenhouse soil in Korea. K.J.S.S.F. 44: 1169-1175 (2011) Kirschbaum MU. The temperature dependence of soil organic matter
decomposition, and the effect of global warming on soil organic C storage. Soil Biol. Biochem. 27: 753-760 (1995)
Kisluk G, Yaron S. Presence and persistence of Salmonella enterica serotype Typhimurium in the phyllosphere and rhizosphere of spray-irrigated parsley. Appl. Environ. Microbiol. 78: 4030-4036 (2012)
Kralik P, Ricchi M. A basic guide to real time PCR in microbial diagnostics: definitions, parameters, and everything. Front. Micro-biol. 8: 108-117 (2017)
Kumar PS, Brooker MR, Dowd SE, Camerlengo T. Target region selection is a critical determinant of community fingerprints gen-erated by 16S pyrosequencing. PLoS One 6: e20956 (2011) Langille MG, Zaneveld J, Caporaso JG, McDonald D, Knights D,
Reyes JA, Clemente JC, Burkepile DE, Thurber RLV, Knight R, Beiko RG, Huttenhower C. Predictive functional profiling of microbial communities using 16S rRNA marker gene sequences. Nat. Biotechnol. 31: 814-821 (2013)
Le Loir Y, Baron F, Gautier M. Staphylococcus aureus and food poi-soning. Genet. Mol. Res. 2: 63-76 (2003)
Liu YU, Geng JC, Sha XY, Zhao YX, Hu TM, Yang PZ. Effect of Rhizobium Symbiosis on Low-Temperature Tolerance and Antiox-idant Response in Alfalfa (Medicago sativa L.) Front. Plant Sci. 10: 538-551 (2019)
Lopez-Velasco G, Welbaum GE, Boyer RR, Mane SP, Ponder MA. Changes in spinach phylloepiphytic bacteria communities follow-ing minimal processfollow-ing and refrigerated storage described usfollow-ing pyrosequencing of 16S rRNA amplicons. J. Appl. Microbiol. 110: 1203-1214 (2011)
Lozupone CA, Hamady M, Kelley ST, Knight R. Quantitative and Qualitative β Diversity Measures Lead to Different Insights into Factors That Structure Microbial Communities. J. Appl. Environ. Microbiol. 73: 1576-1585 (2007)
Lu L, Ku KM, Palma-Salgado SP, Storm AP, Feng H, Juvik JA, Nguyen TH. Influence of epicuticular physicochemical properties on porcine rotavirus adsorption to 24 leafy green vegetables and tomatoes. PLoS One 10: e0132841 (2015)
Madhaiyan M, Reddy BV, Anandham R, Senthilkumar M, Poonguzh-ali S, Sundaram SP, Sa T. Plant growth-promoting Methylobacte-rium induces defense responses in groundnu (Arachis hypogaea L.) compared with rot pathogens. Curr. Microbiol. 75: 988-996 (2007)
MAFRA. 2018 Facility vegetables greenhouse status and vetegable production performance. Available from: https://www.mafra.go.kr/ mafra/366/subview.do?enc=Zm5jdDF8QEB8JTJGYmJzJTJGbW F m cm El M kY3M S UyR jM yM T Y2O CU yRm FydG NsV m ll -dy5kbyUzRg%3D%3D. Accessed Oct. 15, 2019.
Mahmood A, Takagi K, Ito K, Kataoka R. Changes in endophytic bacterial communities during different growth stages of cucumber (Cucumis sativus L.). World J. Microbiol. Biotechnol. 35: 1-13 (2019)
McCabe-Sellers BJ, Beattie SE. Food safety: emerging trends in foodborne illness surveillance and prevention. J. Am. Diet. Assoc. 104: 1708-1717 (2004)
MFDS. Food safety information portal. Available from: https:// www.foodsafetykorea.go.kr/portal/healthyfoodlife/foodPoisoning-Stat.do?menu_no=3724&menu_grp=MENU_NEW02. Accessed
Dec. 7, 2020.
Miller RR, Montoya V, Gardy JL, Patrick DM, Tang P. Metagenom-ics for pathogen detection in public health. Genome Med. 5: 1-14 (2013)
Murray BE. The life and times of the Enterococcus. Clin. Microbiol. Rev. 3: 46-65 (1990)
Nakata K. High resistance to oxygen radicals and heat is caused by a galactoglycerolipid in Microbacterium sp. M874. J. Biochem. 127: 731-737 (2000)
Naravaneni R, Jamil K. Rapid detection of food-borne pathogens by using molecular techniques. J. Med. Microbiol. 54: 51-54 (2005) Nordmann P, Naas T, Poirel L. Global spread of
carbapenemase-pro-ducing Enterobacteriaceae. Emerg. Infect. Dis. 17: 1791-1798 (2011)
Panoff JM, Corroler D, Thammavongs B, Boutibonnes P. Differentia-tion between cold shock proteins and cold acclimaDifferentia-tion proteins in a mesophilic gram-positive bacterium, Enterococcus faecalis JH2-2. J. Bacteriol. 179: 4451-4454 (1997)
Park SH, Park WS, Kim MR. Quality characteristics of commercial Oiji, Korean cucumber pickle. Korean J. Food Sci. Technol. 36: 385-392 (2004)
Pérez-Díaz IM, Hayes JS, Medina E, Webber AM, Butz N, Dickey AN, Lu Z, Azcarate-Peril MA. Assessment of the non-lactic acid bacteria microbiota in fresh cucumbers and commercially fer-mented cucumber pickles brined with 6% NaCl. Food Microbiol. 77: 10-20 (2019)
Pérez-García A, Romero E, De Vicente A. Plant protection and growth stimulation by microorganisms: biotechnological applica-tions of Bacilli in agriculture. Curr. Opin. Biotech. 22: 187-193 (2011)
Rasmussen M. Aerococcus: an increasingly acknowledged human pathogen. Clin. Microbiol. Infect. 22: 22-27 (2016)
Rathinavelu S, Zavros Y, Merchant JL. Acinetobacter lwoffii infection and gastritis. Microbes Infect. 5: 651-657 (2003)
Schröttner P, Rudolph WW, Taube F, Gunzer F. First report on the isolation of Aureimonas altamirensis from a patient with peritoni-tis. Int. J. Infect. Dis. 29: 71-73 (2014)
Shah N, Tang H, Doak TG, Ye Y. Comparing bacterial communities inferred from 16S rRNA gene sequencing and shotgun metage-nomics. Biocomput. 16: 165-176 (2011)
Shaw RK, Lasa I, García BM, Pallen MJ, Hinton JC, Berger CN, Frankel G. Cellulose mediates attachment of Salmonella enterica Serovar Typhimurium to tomatoes. Environ. Microbiol. Rep. 3: 569-573 (2011)
Shim WB, Lee CW, Jeong MJ, Kim JS, Ryu JG, Chung DH. An investigation of the hazards associated with cucumber and hot pepper cultivation areas to establish a good agricultural practices (GAP) model. Korean J. Food Sci. Technol. 46: 108-114 (2014) Sperandio V, Nguyen Y. Enterohemorrhagic E. coli (EHEC)
patho-genesis. Front. Cell. Infect. Microbiol. 2: 91-97 (2012)
Stover CK, Pham XQ, Erwin AL, Mizoguchi SD, Warrener P, Hickey MJ, Brinkman FSL, Hufnagle WO, Kowalik DJ, Lagrou M. Complete genome sequence of Pseudomonas aeruginosa PAO1, an opportunistic pathogen. Nature 406: 959-964 (2000) Thukral AK. A review on measurement of Alpha diversity in
Biol-ogy. Agric. Res. 54: 1-10 (2017)
Turner TR, James EK, Poole PS. The plant microbiome. Genome Biol. 14: 1-10 (2013)
Větrovský T, Baldrian P. The variability of the 16S rRNA gene in bacterial genomes and its consequences for bacterial community analyses. PLoS One 8: e57923 (2013)
Williams TR, Marco ML. Phyllosphere microbiota composition and microbial community transplantation on lettuce plants grown indoors. mBio. 5: e01564-14 (2014)
Xicohtencatl-Cortes J, Chacón ES, Saldana Z, Freer E, Girón JA. Interaction of Escherichia coli O157: H7 with leafy green pro-duce. J. Food. Prot. 72: 1531-1537 (2009)
Xu Z, Xie J, Soteyome T, Peters BM, Shirtliff ME, Liu J, Harro JM. Polymicrobial interaction and biofilms between Staphylococcus aureus and Pseudomonas aeruginosa: an underestimated concern in food safety. Curr. Opin. Food. Sci. 26: 57-64 (2019)
pseudomallei strains at 5 degrees. Kansenshogaku zasshi 67: 331-335 (1993)
Young G, Turner S, Davies JK, Sundqvist G, Figdor D. Bacterial DNA persists for extended periods after cell death. J. Endod. 33: 1417-1420 (2007)
Yanagida F, Chen Y, Onda T, Shinohara T. Durancin L28-1A, a new bacteriocin from Enterococcus durans L28-1, isolated from soil. Lett. Appl. Microbiol. 40: 430-435 (2005)
Yu YC, Yum SJ, Jeon DY, Jeong HG. Analysis of the microbiota on lettuce (Lactuca sativa L.) cultivated in South Korea to identify foodborne pathogens. J. Microbiol. Biotechnol. 28: 1318-1331 (2018)
Zhang X, Wei H, Chen Q, Han X. The counteractive effects of nitro-gen addition and watering on soil bacterial communities in a steppe ecosystem. Soil Biol. Biochem. 72: 26-34 (2014)