식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 1 / 9 BRIC View 2014-T03
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용
강양제
서울대학교
E-mail: kangyangjae@gmail.com
요약문
차세대 Sequencing 기술이 개발되고 생물 DNA 정보의 빅데이터 시대가 도래하였다. 식량 작물은 대량의 분자표지 개발, 존재하는 유전자의 목록화 및 기능 분석, 유전자 서열의 비교 분석을 통한 기능 분화 및 진화 분석 등을 위하여 차세대 Sequencing 기술을 기반으로 전장 유전체를 작성한다. 현재까지 식용 작물에 해당하는 유전체는 최근 완성된 고추, 커피, 가지 등을 포함한 35종에 이른다. 향후 국내 요구에 따른 식물 유전체가 다양하게 구축될 것으로 예상되며 유전체 개선, 거대 유전체 (big genome), 범 유전체 (pan genome) 관련된 연구가 활발히 진행 될 것이다. 이렇게 개발된 유전체 정보들은 핵심 유전 자원의 구축, 대량화 자동화된 유전형 분석에 의해서 육종이 가속화 될 것이며 이는 현재까지 연구되지 않던 식량 작물에도 빠르게 활용될 수 있다. 향후 대량 표현형 분석이 가능해 진다면 예측 모델에 의한 육종이 실현될 가능성이 높다.
Key Words: 전장유전체, 식량작물, NGS, de novo assembly.
목 차
서론 본론
1. NGS 를 이용한 전장 유전체 구축 소개 2. 현재까지의 식량작물 전장유전체 구축 동향 3. 향후 전장유전체 구축 전망
3.1 유전체 개선 (sequence improvement) 3.2 대형 유전체 (big genome)
3.3 범 유전체 (pan genome) 4. 육종에의 활용
BRIC View 동향리포트
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 2 / 9
4.2. 유전자원관리 4.3. 향후 전망 결론
참고문헌
서론
차세대 Sequencing 기술이 개발되고 생물 DNA 정보의 빅데이터 시대가 도래하였다. 2000년 초 인간 유전체가 완성될 때 투자된 비용과는 비교될 수 없을 만큼 저렴한 비용으로 대량의 DNA 를 생산할 수 있게 되었고 이에 따라 세계적으로 생산되는 생물 DNA 정보는 기하급수적으로 늘어나고 있다. 나아가 차차세대 Sequencing 기술은 더 길고 정확한 서열을 제공하기 위해 쉴새 없이 개발되고 발표되고 있다. 이에 발맞추어 DNA을 분석하는 유전체학 역시 급속히 발달하고 있으며 이러한 데이터 기반 생물학은 컴퓨터학과 생물학의 융합 학문으로서 새롭게 대두하고 있다.
이러한 학문의 흐름은 과연 식량 작물의 연구에는 어떻게 적용되고 있고, 어떤 영향을 미치게 되었으며, 나아가 육종에는 어떻게 활용될 수 있는지 알아 보도록 한다.
본론
1. NGS를 이용한 전장 유전체 구축 소개
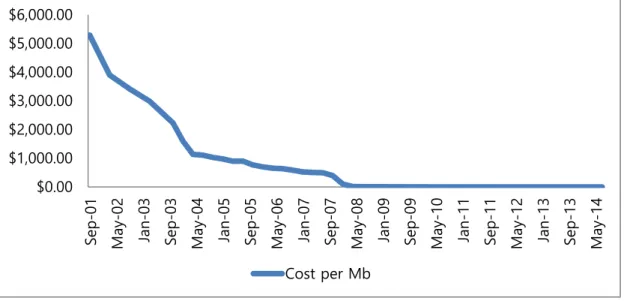
식량 작물은 대량의 분자표지 개발, 존재하는 유전자의 목록화 및 기능 분석, 유전자 서열의 비교 분석을 통한 기능 분화 및 진화 분석 등을 위하여 전장 유전체를 작성한다. 애기장대 (Ara- bidopsis thaliana) 및 대두 (Glycine max) 등 초창기의 모델 유전체들은 한번에 약 500-700 base pair (BP) 를 읽는 Sanger sequencing 기술을 통해 최적, 최소화 된 Bacterial Artificial Chromosome (BAC) Library를 Sequencing하고 예측된 Physical map 에 따라 이어 붙여 구축되었다. 하지만 천문학적인 액수가 하나의 유전체를 완성하는데 투자되어야 했기 때문에 개별적인 비인기 식물들은 Sequencing 될 수 없었다. 그런데 Illumina, Roche GS-FLX 와 같은 차세대 시퀀서 (Next generation sequencer, NGS)가 개발되고, 한번에 긴 서열을 읽는 Sanger sequencing 기술에 비해서 총 생산량이 기하급수적으로 증가하여 단위 시간당 그리고 단위 비용당 생산되는 총 DNA 서열량은 급속하게 증가하게 되었다. 따라서 유전체 구축을 위한 비용적 측면이 단일 연구소에서 감당할 수준으로 줄어들게 되었다(Figure 1, http://www.genome.gov/sequencingcosts/).
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 3 / 9
Figure 1. 단위 DNA (Mb) 당 비용의 감소 추이
현재 유전체 서열 구축을 위해서는 원하는 Sequencing platform을 통한 DNA의 대량 생산, 그리고 de novo assembly를 통한 draft scaffold sequence구축, 그리고 genetic map 및 physical map을 이용하여 알려진 연관군 혹은 염색체로 Anchoring 이 수행되어 최종적으로 Pseudo- molecule 을 만드는 과정을 거치게 된다. 유전체 구축을 위한Sequencing platform은 Illumina Hiseq, Pacbio, Illumina Moleculo 등이 현재 여러 업체에서 서비스 되고 있으며 특히 Pacbio와 Illumina Moleculo는 긴 서열을 제공하기 때문에 복잡하고 거대한 유전체를 Assemble하는데 좋은 대안으로 등장하였다. 이러한 raw data들을 이용하여 연속성 있는 DNA로 assembly 하기 위해서는 여전히 고사양의 서버급 컴퓨터가 필요하며 적절한 소프트웨어의 도움이 필요하다. Broad institute 에서 제공하는 de novo assembly 기반 DNA 변이 탐색 소프트웨어 Discovar 의 benchmarking server정보를 참조하면 단일 연구소의 공동기기로 구매하기에는 무리가 없는 사양을 찾아볼 수 있다 (http://www.broadinstitute.org/software/discovar/blog/?page_id=187). 그리고 연관군 혹은 염색체 수준의 assembly를 원할시에 필요한 genetic map은 mapping population만 준비 되어 있다면 Genotyping by sequencing (GBS)기술을 이용하여 샘플당 약 2~5만원 수준의 비용으로 수천 개의 단일염기변이를 탐색 할 수 있다1. 그리고 여전히 높은 비용이지만 Optical mapping은 광학 기술을 이용하여 빠르고 정확한 Physical mapping을 가능하게 하였기 때문에 이를 통한다면 더욱 정교한 유전체 정보를 구축할 수 있다2.
2. 현재까지의 식량작물 전장유전체 구축 동향
현재까지 약 90 종 정도의 이상의 식물이 Sequencing 되었고 현재 진행 중인 프로젝트를 고려한다면 100종 이상의 식물의 서열을 알고 있다고 볼 수 있다. 이 중 식용 작물에 해당하는 유전체는 최근 완성된 고추, 커피, 가지 등을 포함한 35종에 이른다. 따라서 세계 사람들이 일반적으로 식용으로 이용하는 식물의 유전체 정보 및 유전자 서열 정보는 대부분 알게 되었다고 말할 수 있다. 앞으로는 각 국에서 국내에서 많이 요구되는 식량 작물에 대해서 Sequencing을 할
$0.00
$1,000.00
$2,000.00
$3,000.00
$4,000.00
$5,000.00
$6,000.00
Sep-01 May-02 Jan-03 Sep-03 May-04 Jan-05 Sep-05 May-06 Jan-07 Sep-07 May-08 Jan-09 Sep-09 May-10 Jan-11 Sep-11 May-12 Jan-13 Sep-13 May-14
Cost per Mb
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 4 / 9
유전체 사업의 목적 중 하나는 고유 농생명 자원의 표준 유전체 정보 생산 및 체계를 갖추는 것이다. 해당 사업에서는 유전체 해독이 완료되었거나 진행이 추진되는 품목은 제외 하고 농림축산업에서 중요한 품목이며 우리 고유 자원이면서 유전체 해독에 적합한 품목이며 유전체 크기가 작고 유전적 순도가 높은 이배체인 식용 작물을 선정하여 표준 유전체를 만들 예정이다.
이에 식량 작물로서 들께, 메밀, 고구마가 선정되었고, 원예 작물로서 양파, 국화, 배, 특용 작물로서 만가닥 잎새버섯, 도라지, 결명자가 선정되었다. 그외 곤충, 축산, 산림 관련 대상 생물이 10 종 선정되었다.
3. 향후 전장유전체 구축 전망
3.1 유전체 개선 (sequence improvement)
현재까지 많은 식물 유전체가 생산되고 유명한 저널에 소개되었으나 실제로 그 데이터를 확 인 해보면 그 완성도는 천차 만별이다. 초기의 대량의 자본을 소모하여 작성한 표준 유전체인 애기 장대, 쌀, 대두 등은 기본적으로 Bacterial Artificial Chromosome (BAC)을 선발하여 Minimum Tiling Path (MTP) 를 작성하여 Physical Map을 작성하는 과정을 거친 뒤 모든 Clone들을 Sanger Sequenc- ing 기술을 통해서 정교하게 만들어진 반면, 최근 생성된 표준 유전체 들은 Illumina Hiseq 과 같은 대량의 Short reads들로 Assemble이 되었기 때문에 Assemble의 완성도 면에서 떨어지는 면을 보인 다 (Figure 2). 특히 Assemble한 Sequence의 전체 합에서 반이 되는 위치의 Contig 혹은 Scaffolds 길이를 뜻 하는 N50 값과 염색체 및 연관군에 Anchoring 되지 못한 scaffolds 의 갯수를 살펴보면 대두의 경우는 N50값이 48Mb에 1170개의 Scaffold 가 연관군에 속하지 않는다 3. 반면 비둘기콩 (Pigeon pea)의 경우에는 N50값이 약 540Kb 밖에 되지 않으며 137,531개의 Scaffolds 가 연관군에 속하지 못하였다 4. 차세대 시퀀서를 이용한 Assemble이 모두 비둘기 콩처럼 완성도가 떨어지지는 않지만 연구 그룹에 따라서 완성도에 투자를 많이 한 경우와 그렇지 않은 경우가 존재하기 때문에 Assemble 결과를 신뢰하기 힘든 경우도 있다. 따라서 향후 해당 식물의 이용 목적에 따라서 정교한 유전체 정보가 필요한 경우에는 Long read Sequencing을 통한 유전체 Assemble의 개선을 수행하는 것이 좋을 것이다.
Figure 2. Assemble리 전략 A. Minimal tiling path를 이용한 Sanger 기반 Assemble, B. 다양한 NGS short read library 를 이용한 Assemble5
A B
Assembly paired end
mate pair
NNN NNN NNN
...
...
Physical mapMTP BAC Clones Final assembly
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 5 / 9
3.2 대형 유전체 (big genome)
식용으로서 그리고 산업으로서 가치가 높은 식량 작물들은 높은 배수성을 가지는 경우가 많 다. 예를 들면 사탕수수 (Sugar cane) 는 Saccharum officinarum (2n=8x=80) 와 S. spontaneum (2n=5x=40 ~ 2n=16x=128)의 종간 교잡으로 만들어졌으며 10Gb 이상의 대형 유전체를 가지고 있다.
그래서 Donor식물의 Sequencing을 통한 사탕수수 유전체의 재구성 및 직접 Sequencing 전략을 이 용해서 전장유전체 Sequencing으로 접근하고 있다 6. 그리고 높은 단백질 함량 (종자 건물중의 24 – 35 %)를 가지고 있어 아프리카 및 일부 아시아 지역 그리고 라틴 아메리카 지역의 저소득 층의 주 요 단백질 공급원으로 활용되고 있는 Faba bean (Vicia faba) 은 염색체 수는 2n = 2x = 12 로 작지 만 유전체 크기는 16 Gb로 매우 크다 7. 이러한 작물들의 유전체 Sequencing은 현재의 NGS - short read 기술로는 배수성에 의한 유사 서열의 구분 및 retrotransposon에 의한 반복 서열의 구분이 어 렵기 때문에 Pacbio 나 Illumina Moleculo 같은 긴 서열 기술이 필수적이며, de novo assembly에는 Long read의 경우 최소 50x 이상의 Sequencing이 필요한 것을 감안한다면 대형유전체 Sequencing 에 들어갈 투자 비용은 상당할 것으로 예상 되므로 해당 대형유전체 작물의 산업적 가치를 충분히 계산한 뒤 접근하는 것이 옳을 것으로 생각한다.
3.3 범 유전체 (pan genome)
표준유전체 작성 이후에 연구자들이 알게된 사실은 단일 개체의 표준 유전체 하나를 기준으 로 하는 Resequencing만으로는 많은 생물 현상을 설명하기 부족하며 특히 개체 특이적으로 존재하 는 Sequence에 대한 분석이 빠진다는 것이다. 그에 대한 대안으로 이미 표준 유전체가 있다 하더라 도 특이한 혹은 중요한 개체를 선발하여 다시 de novo assembly를 수행하고 각 개체의 독립적인 Assemble을 비교하여 해당 종에 존재하는 모든 유전체 정보를 뜻하는 pan-genome 과 해당 종에서 없으면 안 되는 core genome을 확인하고 이를 해당 개체들의 특이성을 판별하는데 이용하고 그에 대한 생물학적 의미를 도출한다.
이러한 연구는 최근 중국 연구 그룹에서 지역적으로 특이한 7개의 야생콩 (Glycine soja) 을 이용하여 수행되었다. 야생콩은 현재 재배콩에는 존재하지 않는 다양한 대립 유전자의 Pool로 생각 되고 이를 향후 육종에 활용한다면 다양한 병충해 및 환경 변화에 대응할 수 있을 것으로 간주되고 있다. 따라서 야생콩의 pan-genome을 확인하고 core-genome과 개체별 특이 서열을 추려내는 것은 육종적인 의미가 매우 크다 하겠다. 해당 연구로 인해 7개의 야생콩간 Copy number variation, large mutation 을 많이 발견하였으며 특히 병저항성 그리고 개화에 관련된 유전자의 구조적 변이를 발견 할 수 있었다. 이는 다양한 대립 유전자의 발견 뿐 아니라, 종내에서의 빠른 진화를 보이는 유전자 들의 목록을 확인 할 수 있다는 점에서도 의미가 있다 8.
4. 육종에의 활용
4.1. 대량의 분자표지 분석
일단 표준 유전체가 완성되면 다음과 같은 분자 육종 재료가 확보된다. 첫째는 대량의 마커이
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 6 / 9
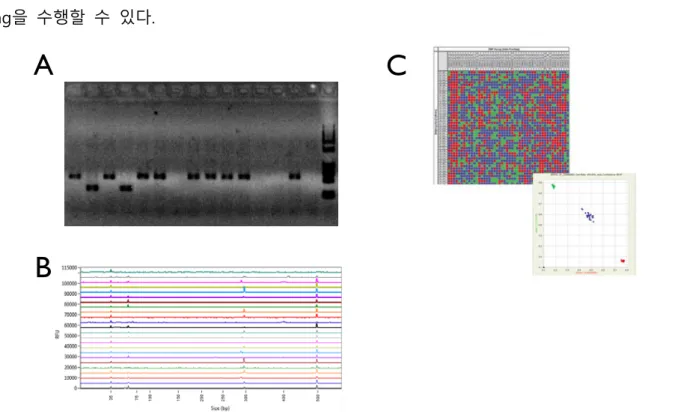
조사하여 쉽게 Primer 서열을 제작할 수 있으며 반복 서열의 갯수 및 Primer 서열의 유전체 내의 중 복 여부 등을 확인하여 다형성을 보이는 실용적 마커로 개발 가능하다. 일단 잘 증폭되는 Primer가 개발이 되면 전기 영동을 통해 다형성을 판별하여 분자 육종에 이용할 수 있으며 최근 연구소 및 종자 회사에는 Fragment analyzer를 통한 자동화된 대량 genotyping platform을 사용하고 있다 (Figure 3). 그리고 표준 유전체에 육종에 이용되는 대표적 유전 자원을 Resequencing하여 유용 SNP 를 개발하고 안정적으로 증폭되는 Intron spanning Primer를 개발한다면 Fluidigm SNPtype를 이용하 여 92개의 샘플을 92 개의 마커로 동시에 분석할 수 있다. 따라서 연구자가 육종 집단 혹은 재래 집단을 가지고 있을 경우 예전보다 노동력 및 비용 면에서 매우 적은 수준에서 분자 육종 및 QTL mapping을 수행할 수 있다.
Figure 3. Genotyping methods A. 전기영동을 통한 genotyping, B. Fragment analyzer(http://aati- us.com/)를 이용한 SSR genotyping, C. Fluidigm SNPtype example (http://www.fluidigm.com)
4.2. 유전자원관리
효과적인 육종을 위해서는 유전적 거리가 적당히 존재하는 유전 자원을 활용하는 것이 중요 하다. 현재 유전 자원은 수집지에 따라 정리되어 있는데 유전체 상으로는 중복된 개체가 매우 많다.
그래서 이를 표준 유전체에 따른 Genotyping을 통해 핵심 집단으로 재 정리할 필요성이 있다. 이 때 쓰일 수 있는 저렴한 Genotyping 방식이 Genotyping by sequencing (GBS) 이다 (Figure 4). 이는 Illumina Hiseq 의 1 lane 에 최대 192개까지 pooling하여 sequencing 하는 방식으로 제한 효소의 근방의 유전체 영역만을 Sequencing 하는 sub-genome genotyping 이다. 1 lane 의 Sequencing 가 격 및 라이브러리 제작 비용을 고려하였을 때 개체당 3~4만원의 비용이 들기 때문에 매우 경제적인 방법으로 많이 이용되고 있다. 이렇게 정리된 핵심 집단은 GBS에서 개발된 SNP정보를 그대로 이용 하여 육종에 이용될 수 있다.
A
B
C
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 7 / 9
Figure 4. GBS library 준비과정
4.3. 향후 전망
표준 유전체를 가지고 있을 경우 core genome에 대한 유전형 분석은 앞으로 더 낮은 가격과 노동력을 통해서 분석이 가능할 것으로 보이며 그 데이터량은 엑셀에서 분석할 수 있는 수준을 넘 어설 것이다. 그리고 더 다양화 되고 세밀한 육종을 위해 필요한 다음 단계는 표현형 분석으로 생각 한다. 현재 표현형 데이터의 자동화를 추진하고 있는 회사는 lemnatech 으로 control되는 시설 기반 표현형 관찰 시스템 및 Field에서 직접 표현형을 관찰하는 시스템을 개발하고 있다. 현재까지는 환경 에 따른 초형의 변화 및 도복이 Field 표현형 관찰의 커다란 장애이지만 이를 해결하기 위해 많은 투자를 하고 있다 (Figure 5). Control된 자동화 온실 시스템에서는 초형이 안정된 작물의 표현형 데 이터를 효과적으로 획득하고 있다. 이렇게 확보할 수 있는 표현형은 엽면적, Chlorophyll 함량, 줄기 직경, 초장, 성장률, 엽 색 (Leaf color) 변화 분석, 병징 분석, 생체중, 마디 길이, 초형 등등 이며 앞 으로 더 많은 표현형을 자동으로 조사할 수 있을 것이다. 표현형 관찰의 해상력 증가 및 자동화가 된다면 진정한 육종의 빅데이터 분석 시대로 넘어가게 될 것이며 “감”에 의한 육종이 아닌 예측 기 반의 육종이 더 이상 꿈이 아니게 될 것이다.
Plate DNA & adapter pair Digest DNA with selected RE
Ligate adapters Pool DNAs & Clean up
Perform PCR Clean up PCR
Evaluation
Illumina Hiseq
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 8 / 9
Figure 5. lemnatech의 ScanalyzerField의 구상도 (http://www.lemnatec.com/)
결론
차세대 Sequencing 기술은 식량 작물의 연구에 크게 영향을 미치고 있다. 분자 육종이 이루 어 지지 않던 비인기 작물들도 분자 육종의 기반을 쉽게 구축할 수 있게 되었기 때문에 식량 작물 의 개발 및 육종이 다양화 될 수 있는 기회가 되었다. 이는 먹거리에 대한 국민적 관심이 다양해져 건강 식품에 대한 요구가 높아지고 식물 추출물을 이용한 제약, 화장품 개발 역시 여러 방향으로 시 도되고 있는 상황에 적극적으로 부응할 수 있는 기술적인 진보이다. 대량 분자 표지의 개발, 핵심 유전 자원의 구축, 집단의 대량 유전형 조사가 과거에 비해서 저렴하고 빠르고 효과적으로 진보하였 으며 대량의 표현형 조사가 곁들여 진다면 향 후 예측 모델을 통한 빠른 육종도 충분히 실현 가능 성이 있다고 생각된다.
참고문헌
[1]. Elshire, R.J. et al. A robust, simple genotyping-by-sequencing (GBS) approach for high diversity species.
PLoS One 6, e19379 (2011).
[2]. Zhang, Q. et al. The genome of Prunus mume. Nat Commun 3, 1318 (2012).
[3]. Schmutz, J. et al. Genome sequence of the palaeopolyploid soybean. Nature 463, 178-183 (2010).
식량 작물들의 전장유전체 구축 동향 및 육종에의 활용 강양제 Page 9 / 9 [4]. Varshney, R.K. et al. Draft genome sequence of pigeonpea (Cajanus cajan), an orphan legume crop of re- source-poor farmers. Nature Biotechnology 30, 83-U128 (2012).
[5]. Shultz, J.L. et al. Three minimum tile paths from bacterial artificial chromosome libraries of the soybean (Glycine max cv. 'Forrest'): tools for structural and functional genomics. Plant Methods 2, 9 (2006).
[6]. de Setta, N. et al. Building the sugarcane genome for biotechnology and identifying evolutionary trends.
BMC Genomics 15, 540 (2014).
[7]. Alghamdi, S.S., Migdadi, H.M., Ammar, M.H., Paull, J.G. & Siddique, K.H.M. Faba bean genomics: current sta- tus and future prospects. Euphytica 186, 609-624 (2012).
[8]. Li, Y.H. et al. De novo assembly of soybean wild relatives for pan-genome analysis of diversity and agro- nomic traits. Nat Biotechnol (2014).
The views and opinions expressed by its writers do not necessarily reflect those of the Biological Research Information Center.
강양제 (2014). 식량 작물들의 전장유전체 구축 동향 및 육종에의 활용. BRIC View 2014-T03.
Available from http://bric.postech.ac.kr/myboard/read.php?Board=review0&id=2271 (Nov 18, 2014) Email: member@bric.postech.ac.kr