- 국문요약 -
시신 머리의 연속절단면영상으로 만든
구역화영상과 표면 3 차원영상
목적: 이 연구의 목적은 시신 머리를 대상으로 간격과 화소크기가 0.1 mm 인 연속절단면영상을 만든 다음에, 이것을 바탕으로 수평, 이마, 마루영상을 만들고 퍼뜨려서 뇌의 자기공명영상을 판독하는 데 도움 주며, 또한 구역화영상, 표면 3 차원영상을 만들고 퍼뜨려서 머리의 해부학을 익히는 데 도움 주는 것이다. 또 다른 목적은 이마, 마루영상을 만들 때 수평줄을 지우는 방법, 자동화 정도가 높은 기술로 구역화영상과 표면 3 차원영상을 만드는 방법을 개발하고 알려서 다른 연구자가 의료영상을 마련하는 데 도움 주는 것이다. 대상 및 방법: 고정액 또는 색소를 주입하지 않은 시신 머리를 연속절단해서 연속절단면영상(수평 방향, 간격 0.1 mm, 화소크기 0.1 mm, 빛깔개수 48 bit color)을 만들고 다듬었다. 연속절단면영상을 쌓아서 이마, 마루영상을 만들었고 이마, 마루영상에 있는 수평줄을 자동으로 지웠다. 연속절단면영상, 이마영상, 마루영상에서 보이는 구조물에 낱낱이 이름 붙였다. 연속절단면영상과 비슷한 생체의 7 Tesla 자기공명영상을 찍은 다음에, 자기공명영상의 머리 구조물에 낱낱이 이름 붙였다. 상용 소프트웨어에서 빠른선택도구와 보간을 써서 자동화 정도를 높인 구역화 방법을 개발하였다. 이 방법으로 머리 구조물 64 개의 테두리를 그려서 임시구역화영상(PSD 파일, 1 mm 간격)과 구역화영상(TIFF 파일, 1 mm 간격)을
만들었다. 부피재구성해서 구역화한 구조물의 부피 3 차원영상을 만들었다. 상용 소프트웨어에서 자동화 정도를 높인 표면재구성 방법을 개발하였다. 이 방법으로 머리 구조물의 표면 3 차원영상을 만들었다. 구역화한 구조물의 테두리를 모두 합쳐서 합친 구역화영상을 만들었고, 이것을 둘러보기 소프트웨어에 넣었다. 결과: 2,343 개의 연속절단면영상(수평 방향, 간격 0.1 mm, 화소크기 0.1 mm, 빛깔개수 48 bit color)을 마련하였다. 연속절단면영상과 수평줄을 지운 이마, 마루영상에 구조물 이름을 붙인 것을 마련하였다. 연속절단면영상과 비슷한 생체의 7 Tesla 자기공명영상에 이름 붙인 것을 마련하였다. 234 쌍의 임시구역화영상(PSD 파일, 1 mm 간격)과 구역화영상(TIFF 파일, 1 mm 간격)을 마련하였다. 구역화한 머리 구조물의 부피 3 차원영상을 마련하였다. 향상된 빠른 방법으로 표면재구성해서 머리 구조물 64 개의 표면 3 차원영상을 마련하였다. 연속절단면영상과 이에 들어맞는 구역화영상을 함께 둘러보는 소프트웨어를 마련하였다. 결론: 이 연구에서 만든 시신 머리의 연속절단면영상과 이름 붙인 수평, 이마, 마루영상을 퍼뜨리면 뇌의 자기공명영상을 판독하는 데 도움 될 것이다. 또한 머리 구조물의 구역화영상, 부피 3 차원영상, 표면 3 차원영상을 퍼뜨리면 머리의 해부학을 익히는 데 도움 될 것이다. 즉 다른 연구자는 이 영상으로 머리의 3 차원영상, 가상해부 소프트웨어, 가상수술 소프트웨어 등을 만듦으로써 의학 교육에 이바지할 것으로 기대된다. 머리의 연속절단면영상과 합친 구역화영상을 둘러보는 소프트웨어를 퍼뜨리면 머리의 절단해부학을 익히는 데 도움 될 것이다. 이 연구에서는 수직영상의 수평줄을 지우는 방법, 자동화 정도가 높은 기술로
구역화영상과 표면 3 차원영상을 만드는 방법을 개발하였는데, 이 방법을 알리면 다른 연구자가 의료영상을 마련하는 데 도움 될 것이다.
차 례
국문요약 ··· i 차례 ··· iv 그림 차례 ··· vi 표 차례 ··· xv . Ⅰ 서론 ··· 1 . Ⅱ 연구대상 및 방법 ··· 5 A. 시신의 머리를 연속절단해서 연속절단면영상을 만듦. ··· 5 B. 수평, 이마, 마루영상을 다듬음. ··· 13 1. 이마, 마루영상을 보고 수평영상을 다듬음. ··· 13 2. 이마, 마루영상에서 수평줄을 지움. ··· 18 C. 수평, 이마, 마루영상에서 보이는 구조물에 이름을 붙임. ··· 23 D. 구조물의 테두리를 그려서 구역화영상을 만듦. ··· 29 1. 테두리를 그려서 임시구역화영상(PSD 파일)을 만듦. ··· 29 2. 임시구역화영상을 구역화영상(TIFF 파일)으로 바꿈. ··· 37 3. 구역화영상을 합치고 테두리 속에 빛깔을 채움. ··· 40 E. 부피재구성해서 부피 3 차원영상을 만듦. ··· 45 1. 구역화영상을 임시구역화영상(PSD 파일)으로 바꿈. ··· 45 2. 각 구조물의 부피 3 차원영상을 만듦. ··· 45 F. 표면재구성해서 표면 3 차원영상을 만듦. ··· 49 1. 임시구역화영상의 테두리를 벡터화함. ··· 492. 테두리를 쌓아서 등고선영상을 만듦. ··· 50 3. 등고선 사이에 면을 채움. ··· 51 4. 표면 3 차원영상을 다듬고 합침. ··· 56 . Ⅲ 결과 ··· 62 A. 연속절단면영상 ··· 62 B. 다듬은 수평, 이마, 마루영상 ··· 65 C. 이름 붙인 수평, 이마, 마루영상 ··· 65 D. 구역화영상 ··· 66 E. 부피 3 차원영상 ··· 67 F. 표면 3 차원영상 ··· 68 . Ⅳ 고찰 ··· 73 . Ⅴ 결론 ··· 84 참고문헌 ··· 85 ABSTRACT ··· 90
그림 차례
Fig. 1. Comparison of serially-sectioned images. Visible Human Project data set (left), Visible Korean Human data set (center), and Chinese Visible
Human data set (right) ··· 1
Fig. 2. A Korean male cadaver, which is donated ··· 5
Fig. 3. Comparison of the colors in serially-sectioned images. General colors in the VKH data are similar to a living body (left). General colors in the VHP data are relatively white due to the formalin perfusion (right) ··· 6
Fig. 4. Cadaver head, which is MR scanned ··· 7
Fig. 5. MRIs of cadaver head. The third image shows both anterior commissure and posterior commissure ··· 7
Fig. 6. A horizontal line is drawn on the cadaver face along the laser indicator of
MR machine ··· 9
Fig. 7. The cadaver, which is put into a freezer and frozen to -70℃ ··· 10
experiments (left); embedding agent which is being poured into the
box (right) ··· 10
Fig. 9. Embedding box and the cryomacrotome on their process of a 0.1 mm sectioning by cutting disc (left); sectioned surface, which is being photographed using digital camera (right) ··· 12
Fig. 10. A serially-sectioned image which shows scattered light reflections (left) that
are no longer apparent after using polarizing filters (right) ··· 12
Fig. 11. Schematic image of making coronal and sagittal images after stacking
serially-sectioned images ··· 14
Fig. 12. Software to make coronal and sagittal images of serially-sectioned images with graphic user interface ··· 15
Fig. 13. Serially-sectioned images, which are stacked (top left). After stacking, coronal (top right), sagittal (bottom left), and oblique (bottom right) images are
made ··· 15
Fig. 14. Continuous serially-sectioned images with incorrect alignment (top row); those with the automatically reformed alignment on Photoshop
Fig. 15. Coronal image with incorrect brightness (left); that with the reformed
brightness (right) ··· 17
Fig. 16. Horizontal serially-sectioned image with no horizontal line (left). Coronal (center) and sagittal (right) images with horizontal lines ··· 18
Fig. 17. Sagittal image with horizontal lines. Visible Korean Human (left) and
Visible Human Project (right) ··· 19
Fig. 18. Schematic images (left column) and corresponding fourier spectrums
(right column) ··· 20
Fig. 19. A sagittal image (left) and corresponding fourier spectrum (right) ··· 21
Fig. 20. Comparison of length of notch filter. Length of notch filter is too short (2nd row). Length of notch filter is suitable (1st row). Length of notch
filter is too long (3rdh row) ··· 22
Fig. 21. Corresponding coronal image with horizontal lines (top left), fourier spectrum with notch filter (top center), and resultant image (top right). Corresponding sagittal image with horizontal lines (bottom left), fourier spectrum with
Fig. 22. Serially-sectioned images (top), coronal images (bottom left), and sagittal images (bottom right) of brain, which are selected at 2 mm intervals to be
labeled ··· 25
Fig. 23. Axes x, y, z passing the center of anterior commissure and posterior commissure. Number of each image is numbered according to the axes
x, y, z ··· 26
Fig. 24. Structures are labeled on a serially-sectioned image (left). Red circle is
drawn to identify lobes of cerebrum ··· 27
Fig. 25. Important region of serially-sectioned images is expanded to be shown in detail (top row). Scale is accompanied to identify real size of head
structures ··· 28
Fig. 26. Comparison of MR image and serially-sectioned image. Labeled structures are almost same ··· 29
Fig. 27. A serially-sectioned image showing a bone, which is clarified by 'median' filtering (top row); another one showing a vessel, which is clarified by
Fig. 28. Outline, which fits the structure, is automatically drawn with appropriate tolerance of magic wand tool (left); outline, which doesn't fit the structure, is drawn either with small tolerance (center) or with great tolerance (right) on Photoshop ··· 34
Fig. 29. Outline in small area of globus pallidus (left) to be expanded to fit for all area of globus pallidus (right) using mouse drag by 'quick selection' tool on
Photoshop ··· 36
Fig. 30. Outlines manually drawn (left, right); outline automatically generated
by interpolation (center) ··· 37
Fig. 31. Temporary segmented images of cerebellum with PSD format (left);
segmented image with TIFF format (right) ··· 39
Fig. 32. Comparison of outline thickness in the segmented image with TIFF format. Thickness is 1 pixel (left); thickness is 2 pixel (right) ··· 39
Fig. 33. Comparison of thickness of bitmapped outline. Thickness of bitmapped outline is 2 pixel (left); Thickness of bitmapped outline is 10 pixel (right) ···· 40
Fig. 34. Segmented images of cerebrum, cerebellum, and brainstem, which are
Fig. 35. Serially-sectioned images (left column) and corresponding segmented images (right column) of head. The second row image shows both anterior commissure (1) and posterior commissure (2) ··· 42
Fig. 36. Software to make coronal and sagittal images of segmented images with graphic user interface ··· 43
Fig. 37. A couple of sagittal serially-sectioned images and segmented images
(top row); a coronal couple (bottom row) ··· 44
Fig. 38. A serially-sectioned image (left), which is changed grayly for volume
reconstruction (right)··· 46
Fig. 39. Volume model of head, which were sectioned to display the sectional
planes··· 47
Fig. 40. Volume model of brain, eyeballs, and optic nerves (top row); those of
lentiform nuclei, caudate nuclei, thalami, and brainstem (bottom row) ··· 48
Fig. 41. Comparison of tolerance during vectorization of the outline in temporary segmented image. Tolerance is 2 pixel (left), 1 pixel (center), and 10 pixel
Fig. 42. Surface reconstruction of a schematic structure with three outlines (top row) and that of the cerebellum (bottom row), where neighboring outlines are overlapped. From the left to the right represents outlines model, packed outlines model, columns volume model, columns surface model, and
smoothed surface model ··· 53
Fig. 43. Surface reconstruction of a schematic structure with four outlines (top row) and that of the vessel (bottom row), where neighboring outlines were not-overlapped. Outlines model, disassembled surface models, assembled surface model, and smoothed surface model (From the left column to the right column) ··· 55
Fig. 44. The surface model of cerebellum with (a) 1,214 triangular surfaces,
(b) 4,340 ones, and (c) 284 ones ··· 56
Fig. 45. The surface model of cerebellum with incorrect region (left) and corrected one (right) ··· 57
Fig. 46. Vertices of the lattice (left), linked dynamically to the triangular surfaces of the surface model (right) ··· 58
outlines (left) and by serially-sectioned image (right) ··· 58
Fig. 48. Surface models of sinuses, which are differently colored (left). Transverse sinuses (red), confluence of sinuses (green), occipital sinus (violet), sigmoid sinuses (dark gray), superior sagittal sinus (yellow), inferior sagittal sinus (black), straight sinus (light blue), inferior petrosal sinuses (orange), superior petrosal sinuses (dark blue), cavernous sinuses (light gray). surface models of arteries, which are differently colored (right). common carotid arteries (yellow), external carotid arteries (red), superficial temporal arteries (green), maxillary arteries (violet), internal carotid arteries (dark gray), anterior cerebral arteries, middle cerebral arteries, posterior cerebral arteries, posterior communicating arteries (light blue), basilar artery (light gray), vertebral
arteries (dark blue) ··· 59
Fig. 49. Stacked outlines of cerebrum and cerebellum (left). Surface models of cerebrum and cerebellum are kept proper location and horizontal-vertical
proportion of the structures (right) ··· 60 Fig. 50. Combined surface model, accompanied by layer window ··· 61
Fig. 51. Coronal images showing the head including brain (left), the yellow cerebral arterial wall by atherosclerosis (center), and the anastomosis (arrow) between the occipital sinus and the left internal jugular vein (right) ··· 63
Fig. 52. Zoomed-in serially-sectioned images of the cerebellum (left), midbrain (center), and left ear (right) showing detailed structures ··· 64
Fig. 53. New serially-sectioned image with 0.2 mm pixel size, 24 bits color, and 0.2 mm intervals (left) and old serially-sectioned image with 0.1 mm pixel size,
48 bits color, and 0.2 mm intervals (right) ··· 64
Fig. 54. 1.5 Tesla MRI (left) of a cadaver and 7 Tesla MRI (right) of a living person ··· 66
Fig. 55. The surface model of cerebellum, which is opaque with triangular surfaces (left), opaque without triangular surfaces (center), and semi-transparent
without triangular surfaces (right) ··· 69
Fig. 56. Combined surface model of the head structures, which is differently selected to display ··· 70
Fig. 57. Semitransparently colored combined surface model of the head structures, which is rotated ··· 71
Fig. 58. Browsing software of Visible Korean Human, in which segmented structures'
표 차례
Table 1. Features of the serially-sectioned images and segmented images ··· 8
Table 2. Sixty-four outlined head structures, which are categorized according to
the systems ··· 31
Table 3. Segmented structures; their starting and ending segmented images; and their red, green, blue values for segmented images ··· 38
Table 4. Maya script used for opening and moving outlines of the cerebellum
to construct a outline model ··· 51
Table 5. Different methods to fill the gaps between stacked outlines in order to
I. 서 론
시신 머리의 연속절단면영상은 높은 해상도와 생체의 실제 빛깔을 가지고 있기 때문에 연속절단면영상에 들어맞는 생체의 자기공명영상을 판독하는 데 도움이 된다. 미국, 한국, 중국에서 이미 만든 연속절단면영상이 있지만 다음과 같은 문제가 있다. 연속절단면영상을 만들 때 고정액 또는 색소를 시신에 주입하면 산 사람의 빛깔과 달라지기 때문에 산 사람의 자기공명영상과 비교하는 것이 어렵다는 문제가 있다. 미국에서 만든 온몸의 연속절단면영상(Visible Human Project)은 시신에 고정액을 주입하였기 때문에 연속절단면영상의 빛깔이 생체의 빛깔과 다르다는 문제가 있었고(Fig. 1)(Spitzer 등, 1996), 중국에서 만든 온몸의 연속절단면영상(Visible Chinese Human)은 시신에 색소를 주입하였기 때문에 연속절단면영상의 빛깔이 생체의 빛깔과 다르다는 문제가 있었다(Fig. 1)(Zhang 등, 2006). 이러한 문제를 해결하기 위해서는 연속절단면영상을 만들 때 고정액 또는 색소를 시신에 주입하지 않아야 한다.Fig. 1. Comparison of serially-sectioned images. Visible Human Project data set (left),
Visible Korean Human data set (center), and Chinese Visible Human data set (right).
연속절단면영상의 절단간격이 크면 연속절단면영상을 쌓아서 만든 이마영상, 마루영상의 질이 떨어지기 때문에 이마, 마루자기공명영상을 판독하기 어렵다는
문제가 있다. 미국에서 만든 온몸의 연속절단면영상(Visible Human Project)은 절단간격이 남성 시신의 경우 1 mm, 여성 시신의 경우 0.33 mm 였기 때문에 이마, 마루영상에서 이보다 작은 해부구조물은 볼 수 없다는 문제가 있다(Fig. 1)(Spitzer 등, 1996). 중국에서 만든 온몸의 연속절단면영상(Chinese Visible Human)과 한국에서 만든 온몸의 연속절단면영상(Visible Korean Human)은 절단간격이 0.2 mm 였기 때문에 이마, 마루영상에서 이보다 작은 해부구조물은 볼 수 없다는 문제가 있다(Fig. 1)(김진용 등, 2002; 박진서 등, 2002; Park 등, 2005a; Park 등, 2006; Zhang 등, 2006). 이러한 문제를 해결하기 위해서는 연속절단면영상의 간격이 작아야 한다. 또 연속절단면영상을 쌓아서 만든 이마영상, 마루영상에 수평줄이 생기는데 이 수평줄을 없애면 이마, 마루영상을 개선할 수 있다.
연속절단면영상의 화소크기가 크면, 작은 구조물을 확인할 수 없다는 문제가 있다. 미국에서 만든 온몸의 연속절단면영상(Visible Human Project)은 화소크기가 0.33 mm 였기 때문에 이보다 작은 해부구조물은 볼 수 없다는 문제가 있다(Fig. 1)(Spitzer 등, 1996). 중국에서 만든 온몸의 연속절단면영상(Chinese Visible Human)은 화소 크기가 0.2 mm 였기 때문에 이보다 작은 해부구조물은 볼 수 없다는 문제가 있다(Fig. 1)(Zhang 등, 2006). 이러한 문제를 해결하기 위해서는 연속절단면영상의 화소크기가 작아야 한다. 연속절단면영상과 들어맞는 자기공명영상을 마련해야 하는데 시신의 자기공명영상은 화질이 좋지 않다는 문제가 있다. 미국, 한국, 중국에서 만든 자기공명영상은 시신을 대상으로 찍었기 때문에 뇌의 화질이 좋지 않다는 문제가 있다. 이러한 문제를 해결하기 위해서는 생체의 자기공명영상을 찍어야 하며, 이것이 시신의 연속절단면영상과 비슷한 것이 좋다. 또한 시신의 연속절단면영상과 생체의
자기공명영상에 낱낱이 이름을 붙이면 뇌의 자기공명영상을 판독하는 데 도움될 것이다. 구역화하고 표면재구성할 때 시간이 오래 걸린다는 문제가 있다. 표면 3 차원영상을 만들기 위해서는 구역화영상을 만든 다음에, 구역화영상을 써서 표면재구성해야 하는데, 구역화하고 표면재구성할 때 자동화 정도가 떨어지면 시간이 오래 걸린다. 이러한 문제를 해결하기 위해서는 구역화하고 표면재구성할 때 자동화 정도를 높여서 구역화하고 표면재구성해야 한다. 틀린 구역화영상 때문에 틀린 표면 3 차원영상이 만들어질 수 있는데, 이런 경우에는 다시 구역화영상을 만들지 않고 표면 3 차원영상을 직접 고치는 것이 바람직하다. 이제까지는 주로 부피재구성하는 프로그램을 만들어서 3 차원영상을 만들었기 때문에 프로그램을 만드는 데 시간이 오래 걸리고 다른 연구자가 사용하기 힘들다는 문제가 있었다. 따라서 무료 소프트웨어로 부피재구성해서 부피 3 차원영상을 만드는 것이 편리하다. 연속절단면영상을 둘러보는 소프트웨어에 합친 구역화영상을 넣지 않으면 연속절단면영상에 있는 구조물의 이름이 나타나지 않으며, 따라서 구조물이 무엇인지 알기 어렵다. 이러한 문제를 해결하기 위해서는 구역화영상을 합쳐서 만든 합친 구역화영상을 연속절단면영상을 둘러보는 소프트웨어에 넣어야 한다. 이 연구의 목적은 시신 머리를 대상으로 간격과 화소크기가 0.1 mm 인 연속절단면영상을 만든 다음에, 이것을 바탕으로 수평, 이마, 마루영상을 만들고 퍼뜨려서 뇌의 자기공명영상을 판독하는 데 도움 주며, 또한 구역화영상, 표면 3 차원영상을 만들고 퍼뜨려서 머리의 해부학을 익히는 데 도움 주는 것이다. 또 다른 목적은 이마, 마루영상을 만들 때 수평줄을 지우는 방법, 자동화 정도가 높은
기술로 구역화영상과 표면 3 차원영상을 만드는 방법을 개발하고 알려서 다른 연구자가 의료영상을 마련하는 데 도움 주는 것이다. 이를 위해 이 연구에서는 고정액 또는 색소를 주입하지 않은 시신 머리를 연속절단해서 연속절단면영상(수평 방향, 간격 0.1 mm, 화소크기 0.1 mm, 빛깔개수 48 bit color)을 만들고 다듬었다. 연속절단면영상을 쌓아서 이마, 마루영상을 만들었고 이마, 마루영상에 있는 수평줄을 자동으로 지웠다. 연속절단면영상, 이마영상, 마루영상에서 보이는 구조물에 낱낱이 이름 붙였다. 연속절단면영상과 비슷한 생체의 7 Tesla 자기공명영상을 찍은 다음에, 자기공명영상의 머리 구조물에 낱낱이 이름 붙였다. 상용 소프트웨어에서 빠른선택도구와 보간을 써서 자동화 정도를 높인 구역화 방법을 개발하였다. 이 방법으로 머리 구조물 64 개의 테두리를 그려서 임시구역화영상(PSD 파일, 1 mm 간격)과 구역화영상(TIFF 파일, 1 mm 간격)을 만들었다. 부피재구성해서 구역화한 구조물의 부피 3 차원영상을 만들었다. 상용 소프트웨어에서 자동화 정도를 높인 표면재구성 방법을 개발하였다. 이 방법으로 머리 구조물의 표면 3 차원영상을 만들었다. 구역화한 구조물의 테두리를 모두 합쳐서 합친 구역화영상을 만들었고, 이것을 둘러보기 소프트웨어에 넣었다.
II. 연구대상 및 방법
A. 시신의 머리를 연속절단해서 연속절단면영상을 만듦. 이 연구를 위해 한국 남성 시신을 기증받았다. 시신의 나이는 67 세이고 키는 1,620 mm, 몸무게는 45 kg 이었다(Fig. 2). 시신은 생전에 중증근육무력증 환자로 알려졌는데, 뇌를 포함한 머리는 정상이었다. 시신을 고정하면 살아있는 사람의 뇌, 근육, 그 외 다른 조직의 실제 빛깔을 얻을 수 없기 때문에 시신을 고정하지 않았다(Fig. 3)(Spitzer 등, 1996; Zhang 등, 2006).
Fig. 3. Comparison of the colors in serially-sectioned images. General colors in the VKH
data are similar to a living body (left). General colors in the VHP data are relatively white
due to the formalin perfusion (right).
돌아가신 지 4 시간 후에 시신의 3 Tesla 자기공명영상을 찍었다(Fig. 4). 자기공명영상을 찍기 전에 시신의 머리를 조절해서 좌우구조물이 대칭이고 앞맞교차, 뒤맞교차가 함께 보이는 수평선을 찾았고, 이 방향으로 3 차원 자기공명영상을 찍었다(Fig. 5). 자기공명영상을 찍을 때, repetition time 과 echo time 은 각각 39 msec, 19 msec 였다. 뇌의 구조물을 뚜렷하게 보기 위해 T2 를 강조해서 찍었다. FOV(field of view)와 해상도는 각각 256 mm X 256 mm, 640 X 640 이었다. 자기공명영상의 화소크기는 0.1 mm 였다. slice thickness 와 interslice gap 은 각각 0.4 mm, 0 mm 였다. 따라서 자기공명영상의 간격은 0.4 mm 였다. excitations 횟수는 한번이었다. MRIcro 소프트웨어(MRIcro™, version 1.39)에서 3 차원 자기공명영상을 0.4 mm 간격으로 잘라서 312 개의 수평자기공명영상으로 만들었다. 자기공명영상은 tag image file
가장자리를 잘라내서 해상도를 640 X 640 에서 448 X 576 으로 줄였다(Fig. 5, Table 1). 촬영이 끝난 후, 자기공명영상을 찍을 때 자기공명영상 촬영기에서 표시했던 레이저표시선을 따라 수평선을 피부에 그렸다(Fig. 6). 대뇌낫과 대뇌세로틈새를 따라서 마루선도 그렸다(Fig. 6).
Fig. 4. Cadaver head, which is MR scanned.
Fig. 5. MRIs of cadaver head. The third image shows both anterior commissure and
Table 1. Features of the serially-sectioned images and segmented images
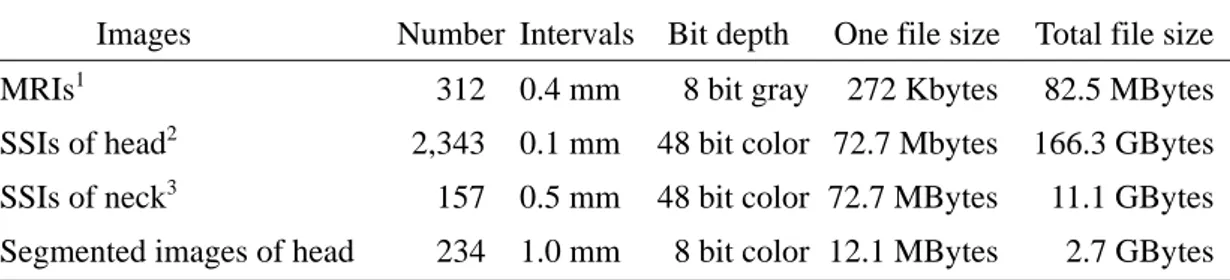
Images Number Intervals Bit depth One file size Total file size MRIs1 312 0.4 mm 8 bit gray 272 Kbytes 82.5 MBytes SSIs of head2 2,343 0.1 mm 48 bit color 72.7 Mbytes 166.3 GBytes SSIs of neck3 157 0.5 mm 48 bit color 72.7 MBytes 11.1 GBytes Segmented images of head 234 1.0 mm 8 bit color 12.1 MBytes 2.7 GBytes 1
Images from vertex to foramen magnum 1
Resolution, pixel size, and file format was 448 X 576, 0.1 mm, and TIFF, respectively. 2
Images from vertex to chin 3
Images from chin to upper border of sternum 2,3
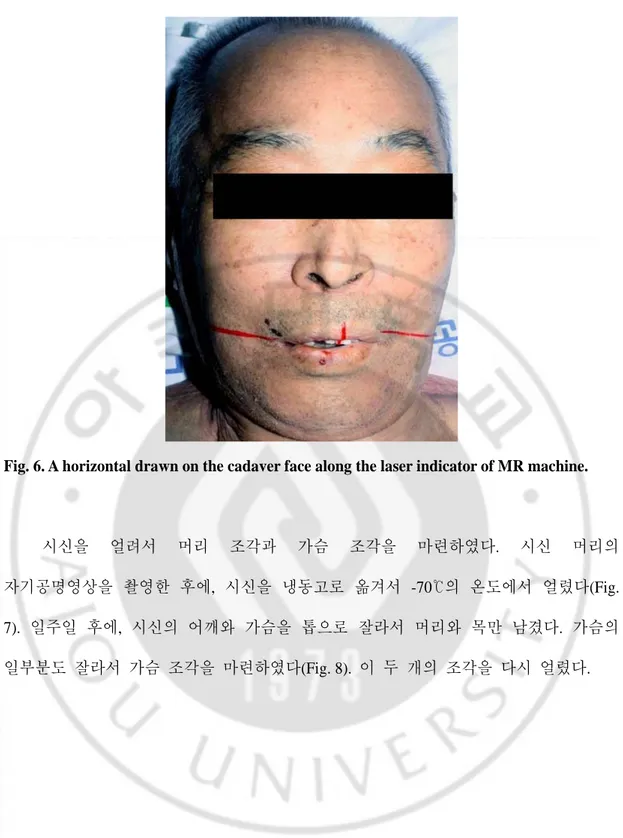
Fig. 6. A horizontal drawn on the cadaver face along the laser indicator of MR machine.
시신을 얼려서 머리 조각과 가슴 조각을 마련하였다. 시신 머리의 자기공명영상을 촬영한 후에, 시신을 냉동고로 옮겨서 -70℃의 온도에서 얼렸다(Fig. 7). 일주일 후에, 시신의 어깨와 가슴을 톱으로 잘라서 머리와 목만 남겼다. 가슴의 일부분도 잘라서 가슴 조각을 마련하였다(Fig. 8). 이 두 개의 조각을 다시 얼렸다.
Fig. 7. The cadaver, which is put into a freezer and frozen to -70℃.
Fig. 8. Head and thorax blocks in the embedding box for the main and preliminary
experiments (left); embedding agent which is being poured into the box (right).
포매상자를 만든 다음에 포매제를 붓고 얼려서 머리 조각과 가슴 조각을 포매하였다. 포매상자의 옆판과 중간판을 나무로 만들었고, 앞판, 뒷판, 바닥판을
강철로 만들었다. 머리 조각을 넣을 때, 시신에 그린 수평선, 마루선과 포매상자의 짧은 축, 긴 축을 평행하게 맞추어서 넣었다. 가슴 조각은 예비실험을 위해 머리 조각의 앞쪽에 놓았다(Fig. 9). 포매상자에 단단하게 고정되도록 포매제(증류수 1,000 L, 젤라틴 30 kg, 메틸렌 블루 0.5 kg)를 만들어서 포매상자의 바닥에 붓고 얼렸다. 바닥에 포매제가 3 cm 정도 쌓였을 때, 머리 조각과 가슴 조각을 포매상자에 넣었다. 포매제를 조금 붓고 얼리는 것을 반복하였다(Fig. 8). 이것은 포매제가 얼면서 시신을 누르는 것을 막기 위해서였다. 절단면을 잘 만들고, 사진을 잘 찍기 위한 알맞은 조건을 찾기 위해 예비실험을 하였다. 가슴 조각이 들어있는 포매상자를 연속절단기로 연속절단하였다. 가장 좋은 품질의 절단면을 얻을 수 있도록 절단원반의 회전속도와 포매상자의 이동속도를 조절하였다. 절단원반에 붙어있는 칼날의 교체주기와 드라이 아이스를 절단면에 대는 주기도 찾았다(Fig. 9). 이 결과, 0.1 mm 간격으로 좋은 연속절단면을 얻을 수 있었다. 이 간격은 이전 연구에서 만든 연속절단면영상의 간격보다 작았다(Park 등, 2005). 절단면의 사진을 찍기 위한 최적의 조건을 찾았다. 가슴 조각을 사용한 예비실험에서 Canon EOS 5D™ 디지털사진기와 Canon 50 mm 마이크로 렌즈를 사용하였다. 해상도는 4,368 X 2,912 였고 빛깔개수는 48 비트였다. 절단면의 크기(436.8 mm X 291.2 mm)는 이전 연구에서 만든 온몸의 연속절단면영상의 크기(600 mm X 400 mm)보다 작았다. 절단면의 크기가 436.8 mm X 291.2 mm 가 되도록 디지털사진기와 절단면과의 거리를 조절하였다(Fig. 9). 이 결과, 화소크기가 0.1 mm 인 연속절단면영상을 얻을 수 있었다. 절단면의 밝기를 일정하게 유지하기 위해 두 개의 스트로브(ElinchromTM Digital S strobes)를 설치하고 최적의 위치와 방향을 찾았다. 절단면에서 반사된 빛이 퍼지는 것을 막기 위해 디지털사진기의 렌즈와 스트로보에 편광필터를 씌웠다(Fig. 10). 최적의 필터 구경과 셔터 속도를 찾았다.
Fig. 9. Embedding box and the cryomacrotome on their process of a 0.1 mm sectioning by
cutting disc (left); sectioned surface, which is being photographed using digital camera
(right).
Fig. 10. A serially-sectioned image which shows scattered light reflections (left) that are no
longer apparent after using polarizing filters (right).
본 실험에서, 시신 머리를 연속절단하고, 절단면을 사진 찍어서
연속절단면영상을 만들었다. 연속절단기를 써서 머리 조각을 머리끝에서 턱까지 0.1 mm 간격으로 연속절단하였다. 계속해서, 157 개의 시신 목의 연속절단면영상을 얻기 위해 같은 머리 조각을 턱에서 복장뼈의 위모서리까지 0.5 mm 간격으로
연속절단하였다(Fig. 9). 이 결과로 시신 머리의 연속절단면영상 2,343 개를 마련하였다(Table 1). 절단면에 있는 성에와 가루를 제거한 후에 절단면을 사진 찍어서 연속절단면영상으로 만들었다. 각 연속절단면영상은 포매제, 그레이 스케일(gray scale), 컬러 패치(color patch)를 포함하였다(Fig. 9). 연속절단면영상을 디지털사진기와 연결된 컴퓨터로 옮겨서 빛깔개수가 24 bit color 인 JPEG(joint photographic coding experts group) 파일과 빛깔개수가 48 bit color 인 CR2(Canon raw 2) 파일로 저장하였다. 컴퓨터 모니터에서 JPEG 파일로 된 연속절단면영상을 Photoshop(10 판, Adobe PhotoshopTM, 이하 Photoshop)에서 살펴서 잘 찍혔나 확인하였다. 확인이 끝나면 연속절단의 다음 단계를 진행하였다. 하루의 실험이 끝나면, Canon EOS utility 소프트웨어를 써서 CR2 파일로 된 연속절단면영상들을 빛깔개수가 48 bit
color 인 TIFF 파일로 바꾸었다. 연속절단면영상을 의료영상의 형식에 맞도록 Photoshop 에서 좌우를 뒤집었다. 시신 머리와 목의 연속절단면영상 2,500 개를 만드는 데 50 일이 걸렸다 (Table 1). B. 수평, 이마, 마루영상을 다듬음. 1. 이마, 마루영상을 보고 수평영상을 다듬음. 이마영상, 마루영상을 만들어서 틀린 연속절단면영상을 찾았다. 이마영상과 마루영상을 만들기 위해 연속절단면영상을 쌓아서 자를 수 있는 소프트웨어(Figs. 11, 12)를 만들었다. 이 소프트웨어에서 2,343 개의 연속절단면영상을 연 다음에, 이마영상과 마루영상을 한꺼번에 만들었다(Fig. 13). 이 때 이마영상과 마루영상의 간격을 연속절단면영상과 같은 0.1 mm 로 정했다. 이 소프트웨어에서는 이마,
마루영상뿐 아니라, 비스듬한 각도로 자른 영상도 만들 수 있었다(Fig. 13). 절단면을 사진 찍을 때 정렬을 정확하게 맞추었지만 가끔 정확하게 맞지 않는 경우가 있었다. 이마영상, 마루영상, 자기공명영상을 살펴서 정렬이 틀린 연속절단면영상을 찾았다(Fig. 14). 이마영상, 마루영상에서 밝기가 한결같지 않은 연속절단면영상을 찾았다(Fig. 15).
Fig. 11. Schematic image of making coronal and sagittal images after stacking
Fig. 12. Software to make coronal and sagittal images of serially-sectioned images with
graphic user interface.
Fig. 13. Serially-sectioned images, which are stacked (top left). After stacking, coronal (top
Fig. 14. Continuous serially-sectioned images with incorrect alignment (top row); those with
Fig. 15. Coronal image with incorrect brightness (left); that with the reformed brightness (right). 정렬이 틀리거나 밝기가 한결같지 않은 연속절단면영상을 Photoshop 에서 고쳤다. 연속절단면영상 간의 정렬이 한결같지 않은 경우가 있었다. 이것은 연속절단과 절단면 촬영이 반복되는 사이에 포매상자나 카메라가 조금씩 움직이기 때문에 생긴다(Fig. 14). 연속절단면영상 간의 밝기가 한결같지 않은 경우가 있었다(Fig. 15). 이것은 연속절단과 절단면 촬영이 반복되는 사이에 조명의 노출이나 사진기의 노출 설정이 조금씩 변하기 때문에 생긴다(Fig. 15). 정렬이 한결같지 않은 연속절단면영상을 Photoshop 에서 ‘load files into stack in script’ 도구를 써서 앞뒤 연속절단면영상과 견주어 보면서 ‘move’ 도구와 ‘rotate canvas’ 도구를 써서 정렬을 맞추었다(Fig. 14). 밝기가 한결같지 않은 연속절단면영상을 앞뒤 연속절단면영상과 견주어 보면서 ‘curves’ 도구를 써서 밝기를 맞추었다(Fig. 15). 연속절단면영상의
정렬과 밝기를 한결같게 고친 다음에, 이마, 마루영상을 새로 만들어서 정렬과 밝기를 마지막으로 살폈다. 2. 이마, 마루영상에서 수평줄을 지움. 이마영상, 마루영상에 수평줄이 생기는 문제가 있었다. 연속절단면영상의 정렬과 밝기를 고쳤지만 완벽하게 고칠 수 없었기 때문에 이마, 마루영상에 수평줄이 생겼다(Fig. 16). 이전에 만든 이마, 마루영상은 이러한 문제에 대해서 깊이 있게 연구, 개선하지 않고 사용되었지만, 이 연구에서는 이 문제를 영상처리 방법을 써서 개선하였다(Fig. 17)(조두희 등, 2007).
Fig. 16. Horizontal serially-sectioned image with no horizontal line (left). Coronal (center)
Fig. 17. Sagittal image with horizontal lines. Visible Korean Human (left) and Visible
Human Project (right).
이마, 마루영상의 수평줄을 없애기 위해 이마, 마루영상을 푸리에 스펙트럼으로 변환하였다. 이마, 마루영상을 주파수 스펙트럼으로 변환하기 위한 방법으로, 푸리에 변환(Fourier transform)을 사용하였다(Fig. 18)(황선규, 2007). 푸리에 변환을 할 때 DFT(Discrete fourier transform)라는 방법을 써서 영상을 푸리에 스펙트럼으로 변환시키는 것이 일반적이지만 이 방법은 많은 양의 중복된 계산을 수행하기 때문에 시간이 오래 걸린다는 문제가 있다. 따라서 FFT(Fast fourier transform)라는 방법을 써서 이마, 마루영상을 푸리에 스펙트럼으로 빠르게 변환하였다(Fig. 18)(황선규, 2007). 푸리에 스펙트럼에서 밝은 선으로 나타나는 부분은 선의 방향으로 인접한 화소와의 밝기 차이가 크다는 의미이고, 어두운 부분은 인접한 화소와의 밝기 차이가 작다는 것을 의미한다(Fig. 18). 또한 푸리에 스펙트럼의 가운데에는 저주파 성분이 모여있고, 푸리에 스펙트럼의 가장자리로 갈수록 고주파 성분이 모여있다. 이마, 마루영상은
수평줄 때문에 수직방향으로 인접한 화소와의 밝기 차이가 크기 때문에 푸리에 스펙트럼에서 수직방향으로 밝은 선이 나타났다(Fig. 19).
Fig. 18. Schematic images (left column) and corresponding Fourier spectrums (right
Fig. 19. A sagittal image (left) and corresponding Fourier spectrum (right).
FFT 수식을 컬러 영상에 적용하기 위해 red, green, blue 채널을 나누어서 적용하였다. 연속절단면영상으로부터 얻은 이마, 마루영상에서 보이는 수평줄을 없애기 위한 첫 번째 단계로 FFT 를 이용하여 푸리에 스펙트럼으로 변환시켜야 한다. 그러나 FFT 는 단색 채널 영상에만 적용이 가능하기 때문에 컬러 영상을 얻기 위하여 FFT 와 함께 컬러를 고려한 과정이 추가되어야 한다. 이것을 위해 red, green, blue 채널을 나누고 각각의 채널을 FFT 푸리에 스펙트럼으로 바꾼 다음에 필터를 적용시키고, 그 결과를 다시 합침으로써 컬러 구현을 가능하게 하였다. 푸리에 스펙트럼으로 변환된 이마, 마루영상에 놋치 필터(notch filter)를 적용해서 수직 방향의 고주파 성분을 제거하였다. 푸리에 스펙트럼에서 밝게 보이는 수직 방향의 고주파 성분을 검정색으로 지워서 수평줄의 빛깔을 주변 빛깔과 비슷하게 만들었다. 이렇게 특정 방향에 있는 주파수 성분을 제거하는 과정을 통칭하는 것이 놋치 필터이다. 놋치 필터를 적용할 때, 필터의 길이를 알맞게 조절하였다. 필터의 길이가 짧으면 영상에 변화가 적었고 너무 길면 영상이 검게 변하였다(Fig. 20). 여러 차례의 예비실험을 통해 이마, 마루영상에 알맞은 필터의 길이를 찾아서 모든 이마, 마루영상에 적용하였다. 이 결과로, 이마, 마루영상에 있는 수평줄이 없어졌다(Fig. 21).
Fig. 20. Comparison of length of notch filter. Length of notch filter is too short (2nd row).
Fig. 21. Corresponding coronal image with horizontal lines (top left), fourier spectrum with
notch filter (top center), and resultant image (top right). Corresponding sagittal image with
horizontal lines (bottom left), Fourier spectrum with notch filter (bottom center), and
resultant image (bottom right).
C. 수평, 이마, 마루영상에서 보이는 구조물에 이름을 붙임. 이 연구에서 만든 연속절단면영상을 써서 신경해부학 그림책을 만들기로 하였다. 이미 출판된 신경해부학 그림책은 절단간격이 넓기 때문에 작은 구조물을 볼 수 없고, 고정한 시신으로 만들었기 때문에 생체의 실제 빛깔을 볼 수 없었다(Spitzer 등, 1998). 이러한 문제들을 해결하기 위해 이 연구에서 만든 연속절단면영상에서 보이는 구조물의 이름을 되도록 많이 붙여서 그림책을 만들기로 하였다.
뇌를 포함한 연속절단면영상을 가지고 2 mm 간격의 수평영상, 이마영상, 마루영상을 추렸다(Fig. 22). 각 영상의 위치를 나타내기 위해 다음과 같은 좌표를 만들었다. 앞맞교차와 뒤맞교차의 중점을 원점으로 정하였다(Fig. 23). 원점을 중심으로 x 축의 +방향은 머리 오른쪽, -방향은 축은 머리 왼쪽으로 정하였다(Fig. 23). 원점을 중심으로 y 축의 +방향은 머리 앞쪽, -방향은 머리 뒤쪽으로 정하였다(Fig. 23). 원점을 중심으로 z 축의 +방향은 머리 위쪽, -방향은 머리 아래쪽으로 정하였다(Fig. 23). 원점으로 삼고 x 축, y 축, z 축을 그린 다음에, 2 mm 간격으로 추렸다. 이 결과로 수평영상은 z 축을 기준으로 +62 mm 부터 -58 mm 까지 61 개의 영상을 추렸다. 이마영상은 y 축을 기준으로 +64 mm 부터 -72 mm 까지 69 개의 영상을 추렸다. 마루영상은 z 축을 기준으로 +60 mm 부터 -60 mm 까지 61 개의 영상을 추렸다.
Fig. 22. Serially-sectioned images (top), coronal images (bottom left), and sagittal images
Fig. 23. Axes x, y, z passing the center of anterior commissure and posterior commissure.
Number of each image is numbered according to direction of the axes.
수평, 이마, 마루영상에서 보이는 구조물의 이름을 붙였다. 해부학자, 병리학자, 신경과 전문의, 신경외과 전문의 등이 함께 수평영상, 이마영상, 마루영상에서 보이는 구조물의 이름을 낱낱이 붙였다(Fig. 24). 수평영상에서 대뇌의 어느 엽인지 쉽게 알 수 있도록 테두리를 그려서 대뇌의 엽을 표시하였다(Fig. 24). 수평, 이마, 마루영상의 중요한 부위는 확대해서 구조물의 이름을 낱낱이 붙였다(Fig. 25). 수평, 이마, 마루영상에 이름 붙인 것이 한결같도록 하였다. 수평, 이마, 마루영상에서 머리의 실제 크기를 쉽게 알 수 있도록 자를 표시하였다(Fig. 25). 또, 머리의 어디를 자른 영상인지 쉽게 알 수 있도록 여러 방향에서 자른 그림을 표시하였다(Fig. 25).
Fig. 24. Structures are labeled on a serially-sectioned image (left). Red circle is drawn to
Fig. 25. Important region of serially-sectioned images is expanded to be shown in detail (top
row). Scale is accompanied to identify real size of head structures.
생체의 자기공명영상을 찍고, 자기공명영상에서 보이는 구조물의 이름을 붙였다. 시신과 비슷한 사람을 골라서 7 Tesla 자기공명영상을 찍은 다음에, 자기공명영상에서
보이는 구조물의 이름을 붙이기로 하였다. 하지만 시신의 연속절단면영상과 생체의 자기공명영상이 완전히 들어맞지 않았기 때문에 생체의 자기공명영상을 시신의 상대적인 크기에 맞추었다. 보기를 들어서 수평자기공명영상의 +62 는 실제로는 +66 mm 였다. 생체의 자기공명영상을 연속절단면영상과 견주어보면서 자기공명영상에서 보이는 구조물에도 낱낱이 이름을 붙였다(Fig. 26). 연속절단면영상과 자기공명영상에 이름 붙인 것이 한결같도록 하였다.
Fig. 26. Comparison of MRI and serially-sectioned image. Labeled structures are almost
same.
D. 구조물의 테두리를 그려서 구역화영상을 만듦.
1. 테두리를 그려서 임시구역화영상(PSD 파일)을 만듦.
연속절단면영상에 이름 붙인 구조물 중 64 개를 구역화해서 구역화영상을 만들기로 하였다. 구조물 64 개를 계통에 따라서 피부계통 1 개, 뼈대계통 10 개, 관절계통 4 개, 혈관계통 26 개, 내분비계통 1 개, 신경계통 21 개, 감각기관 1 개로
나누었다. 구조물의 영어 이름은 해부학용어(다섯째 판)를 따랐다(Table
Table 2. Sixty-four outlined head structures, which are categorized according to the systems
Systems Structures
Skeletal (10) Cranium without mandible, Mandible, Hyoid bone, Cervical vertebrae (first to seventh)
Articular (4) Intervertebral discs (second to fifth)
Vascular (26) Common carotid arteries, External carotid arteries, Superficial temporal arteries, Maxillary arteries, Internal carotid arteries, Anterior cerebral arteries, Middle cerebral arteries, Posterior cerebral arteries, Posterior communicating arteries, Basilar artery, Vertebral arteries, Internal jugular veins, Transverse sinuses, Confluence of sinuses, Occipital sinus, Sigmoid sinuses, Superior sagittal sinus, Inferior sagittal sinus, Straight sinus, Inferior petrosal sinuses, Superior petrosal sinuses, Cavernous sinuses, Emissary veins, Superior cerebral veins, Great cerebral vein, Internal cerebral veins
Endocrine (1) Pituitary gland
Nervous (21) Spinal cord, Brainstem, Fourth ventricle, Substantiae nigra, Red nuclei, Cerebellum, Dentate nuclei, Emboliform nuclei, Cerebrum, Thalami, Mammillary bodies, Corpus callosum, Fornices, Septa pellucida, Lateral ventricles, Caudate nuclei, Putamina, Globi pallidi, Optic tracts, Optic chiasm, Optic nerves
Sensory organ (1) Eyeballs Integumentary (1) Skin (Number of outlined structures)
시신 머리의 연속절단면영상을 1 mm 간격으로 추렸다. 시신 머리의 연속절단면영상 2,343 개(0001.tif, 0002.tif, …, 2343.tif; 파일 형식 tag image file format (TIFF), 간격 0.1 mm, 해상도 4,368 X 2,912, 빛깔개수 48 bits color) 중에서 파일 이름이 10 의 배수인 연속절단면영상 234 개(001.tif, 002.tif, …, 234.tif)를 추렸으며, 이 결과로 간격이 0.1 mm 에서 1 mm 로 늘었다.
피부, 뼈처럼 테두리가 간단한 구조물은 종이 구역화를 하지 않고 직접 컴퓨터에서 구역화하였으나, 뇌줄기, 신경처럼 테두리가 복잡한 구조물은 종이 구역화를 한 다음에 컴퓨터에서 구역화하였다. 종이 구역화를 하기 위해 연속절단면영상을 3 mm 간격으로 종이에 인쇄하였다. 종이를 넘겨 보면서 구조물이 어디에 있는지 확인한 다음에 종이에 구조물의 테두리를 그렸다. 나중에 컴퓨터에서 구역화할 때 종이 구역화를 참고하면 구조물이 포함된 영상과 구조물의 테두리를 파악하는 데 도움 되었다(박진서 등, 2006b).
연속절단면영상을 Photoshop 에서 열고, PSD(Photoshop document) 파일로 저장해서 임시구역화영상 234 개(001.psd, 002.psd, …, 234.psd; 간격 1 mm, 해상도 4,368 X 2,912)를 만들었다(Table 3).
연속절단면영상을 여과해서 구조물의 테두리를 뚜렷하게 만들었다.
연속절단면영상의 뼈를 뚜렷하게 만들 때에는 중간값 여과(median filtering)가 좋았고, 혈관을 뚜렷하게 만들 때에는 샤픈 여과(sharpen filtering)가 좋았다(Fig. 27)(정성환 등, 2006). 이처럼 테두리를 뚜렷하게 만들면 다음 과정에서 구역화할 때 자동화 정도를 높일 수 있었다. 구조물의 테두리를 그린 다음에는 여과한 연속절단면영상을 본래대로 바꾸었다. 이 일을 액션에 기록해서 일괄처리하였다.
Fig. 27. A serially-sectioned image showing a bone, which is clarified by 'median' filtering
(top row); another one showing a vessel, which is clarified by 'sharpen' filtering (bottom
row).
마술막대기도구(magic wand tool)를 써서 구조물의 테두리를 자동으로 그렸다. 마술막대기도구를 고른 다음에 마우스 포인터를 구조물 속에 놓고 딸깍해서 구조물의 테두리를 자동으로 그렸다. 테두리를 그리기에 앞서 마술막대기도구의
받아들임값(tolerance)을 30 으로 정했는데, 받아들임값을 너무 크게 정하면(최대 255) 이웃한 화소의 빛깔이 많이 달라도 받아들여서 구조물보다 큰 테두리를 그리기 때문이었고, 받아들임값을 너무 작게 정하면(최소 0) 이웃한 화소의 빛깔이 조금만 달라도 받아들이지 않아서 구조물보다 작은 테두리를 그리기 때문이었다(Fig. 28). 그러나 구조물 빛깔이 주변 빛깔과 아주 많이 다르면 받아들임값을 30 보다 크게 정해서 자동화 정도를 높였고, 구조물 빛깔이 주변 빛깔과 비슷하면 받아들임값을 30 보다 작게 정해서 자동화 정도를 낮추었다. 테두리를 그리기에 앞서 이웃함(contiguous)을 켰는데, 이웃함을 끄면 이웃하지 않지만 빛깔이 비슷한 다른 구조물에도 테두리를 그리기 때문이었다. 테두리를 그리기에 앞서 앤티에일리어스(anti-aliased)를 껐는데, 앤티에일리어스를 켜면 테두리 속에 빛깔을 채울 때 이 빛깔과 주변 빛깔의 중간 빛깔이 테두리에 나타나기 때문이었다.
Fig. 28. Outline, which fits the structure, is automatically drawn with appropriate
tolerance of magic wand tool (left); outline, which doesn't fit the structure, is drawn either
구조물의 테두리를 자동으로 그릴 수 없으면 자석올가미도구(magnetic lasso tool)를 써서 구조물의 테두리를 반자동으로 그렸다. 구조물의 테두리를 반자동으로도 그릴 수 없으면 올가미도구(lasso tool)를 써서 수동으로 그렸다(박진서 등, 2006b). 이 때 보통 컴퓨터 마우스를 쓰는 것보다는 연필처럼 생긴 컴퓨터 마우스(Wacom™ Intuos3 PTZ-630)를 쓰는 것이 편리하였다.
또 다른 방법으로 빠른선택도구(quick selection tool)를 써서 구조물의 테두리를 반자동으로 그렸다. 구조물 일부에 테두리를 그린 다음에 테두리를 잡아 당기면 구조물 전체에 맞는 테두리가 자동으로 그려졌다(Fig. 29). 빠른선택도구는 자석올가미도구보다 자동화 정도가 높았기 때문에 마술막대기도구 다음으로 많이 쓰려고 하였다.
Fig. 29. Outline in small area of globus pallidus (left) to be expanded to fit for all area of
globus pallidus (right) using mouse drag by 'quick selection' tool on Photoshop.
그린 구조물의 테두리가 틀리면 자석올가미도구(magnetic lasso tool)를 써서 구조물의 테두리를 반자동으로 고치거나, 올가미도구(lasso tool) 또는 작업패스(work path)를 써서 구조물의 테두리를 수동으로 고쳤다(황성배 등, 2003).
구역화 시간을 줄이기 위해, 넓은 간격으로 구조물의 테두리를 그린 다음에 보간해서 좁은 간격의 구조물의 테두리를 자동으로 만들기도 하였다. 넓은 간격으로 구조물의 테두리 두 개를 그린 다음에 보간하면, 두 개 테두리 사이에 좁은 간격의 테두리가 자동으로 만들어진다. 이 일을 Combustion(Autodesk™ Combustion version
Combustion 의 움직이기도구(animate tool)로 보간해서 두 개 테두리 사이의 테두리를 자동으로 만들었다(Fig. 30)(서영환, 2005). 즉 4 mm 간격으로 구역화한 테두리를 써서 1 mm 간격의 테두리를 자동으로 만들었다. 이 때 구조물에 갈라지는 부분이 없어야 보간이 가능하였다. 즉, 구조물의 이웃한 테두리의 개수가 같아야 보간할 수 있었다. 불행하게도 보간해서 만든 테두리는 임시구역화영상(PSD 파일)에 직접 담을 수 없었다. 따라서 보간해서 만든 테두리의 속에 검은 빛깔을 채우고 TIFF 파일로 저장한 다음에 Photoshop 의 마술막대기도구를 써서 임시구역화영상에 담을 수 있는 테두리로 변환하였다. 보간해서 만든 테두리가 연속절단면영상에 들어맞는지 확인하였고, 그렇지 않으면 반자동 또는 수동으로 고쳤다.
Fig. 30. Outlines manually drawn (left, right); outline automatically generated by
interpolation (center).
2. 임시구역화영상을 구역화영상(TIFF 파일)으로 바꿈.
구조물의 테두리에 빛깔을 칠한 다음에 임시구역화영상을 구역화영상(TIFF 파일)으로 저장하였다. 임시구역화영상에서 ‘stroke’ 명령을 써서 구조물의 테두리에
빛깔을 칠하였다. 이 때 이미 정한 빛깔을 칠했으며, ‘inside’옵션을 선택해서 테두리 안에 빛깔을 칠하였다(Fig. 31). 빛깔을 칠할 때 선의 굵기를 2 화소로 하였다. 선의 굵기(pixel)가 1 화소이면 구역화영상을 임시구역화영상으로 되돌릴 때 불편하였다(Fig. 32). 선의 굵기가 2 화소보다 굵으면 구조물의 테두리가 속에 있는 다른 구조물의 테두리와 겹치는 경우가 있었다(Fig. 33). 구조물의 테두리에 빛깔을 칠한 다음에 ‘save as’ 명령을 써서 TIFF 파일로 저장하였다. 이 결과로 모든 임시구역화영상을 구역화영상으로 바꾸었다.
Table 3. Segmented structures; their starting and ending segmented images; and their red,
green, blue values for segmented images
Folder name \ structure’s name Starting Ending Red Green Blue segmented image segmented image value value value
Skeletal \ Cranium without mandible 006.tif 184.tif 150 120 0
Skeletal \ Mandible 124.tif 227.tif 0 210 210
Skeletal \ Cervical vertebra (first) 144.tif 163.tif 180 180 0 Skeletal \ Cervical vertebra (second) 146.tif 189.tif 0 0 240
Integumentary \ Skin 001.tif 234.tif 30 240 0
Fig. 31. Temporary segmented images of cerebellum with PSD format (left); segmented
image with TIFF format (right).
Fig. 32. Comparison of outline thickness in the segmented image with TIFF format.
Fig. 33. Comparison of outline thickness in the segmented image with TIFF format.
Thickness is 2 pixel (left); thickness is 10 pixel (right).
어느 구조물을 포함한 모든 구역화영상을 한 폴더에 담았다(Table 3). 보기를 들어서 피부를 포함한 모든 구역화영상(001.tif, 002. tif, …, 234. tif)을 Integumentary 폴더의 하위 폴더인 Skin 폴더에 담았다. 각 구조물이 어느 구역화영상에 있는지 표로 간추렸다. 소뇌의 경우에는 0811.tif, 0821.tif, …, 2411.tif 에 있었다.
3. 구역화영상을 합치고 테두리 속에 빛깔을 채움.
여러 구조물의 구역화영상을 한 개의 구역화영상으로 합쳤다. 여러 구조물의 구역화영상들을 합치는 원리는 다음과 같았다. 각 구역화영상의 배경은 검은색이고, 검은색의 RGB 값은 각각 0, 0, 0 이다. 이 논문의 그림에서는 구조물의 테두리를 잘 나타내기 위해 배경을 흰색으로 나타냈다. 구역화영상에 있는 여러 구조물의
255, 뇌줄기는 255, 255, 0 의 RGB 값을 가진다. 구조물의 테두리는 2 화소 굵기의 선으로 그렸기 때문에 서로 겹치지 않는다. 따라서, 각 구역화영상의 RGB 값을 모두 더해주면 검은색을 배경으로 구조물의 테두리들이 모두 더해지게 된다. 이 결과로 모든 구조물의 구역화영상을 합쳐서 243 개의 합친 구역화영상을 만들었다(Fig. 34).
Fig. 34. Segmented images of cerebrum, cerebellum, and brainstem, which are combined
(left to right). 합친 구역화영상에서 구조물의 테두리 속에 테두리와 같은 빛깔을 채웠다. Photoshop 에서 합친 구역화영상을 열고, ‘eyedropper’ 도구를 써서 한 구조물의 테두리의 빛깔을 선택하였다. ‘magic wand’ 도구를 써서 한 구조물의 테두리 속을 모두 선택한 다음에 ‘paint bucket’ 도구를 써서 한 구조물의 테두리 속을 테두리와 같은 빛깔로 채웠다. 이와 같은 방법으로 합친 구역화영상에 있는 모든 구조물의 테두리 속에 테두리와 같은 빛깔을 채웠다(Fig. 35). 이 결과로 테두리 속에 빛깔을 칠한 합친 구역화영상이 만들어졌으며 이것을 JPEG 파일로 저장하였다.
Fig. 35. Serially-sectioned images (left column) and corresponding segmented images (right
column) of head. The second row image shows both anterior commissure (1) and posterior
테두리 속에 빛깔을 칠한 합친 구역화영상으로 이마영상과 마루영상을 만들어서 구역화영상을 확인하였다(Park 등, 2005b). 구역화영상의 이마, 마루영상을 만들기 위해 연속절단면영상의 이마, 마루영상을 만들었던 소프트웨어를 썼다(Fig. 36). 이 결과로 구역화영상의 이마, 마루영상을 만들었다. 이마, 마루영상에서 구조물의 테두리가 매끄러운지 확인하였고, 연속절단면영상에 들어맞는지 확인하였다(Fig. 37). 이마, 마루영상에서 구조물의 테두리가 매끄럽지 않거나, 연속절단면영상에 들어맞지 않는 영상이 있으면 임시구역화영상을 고친 다음에 이마, 마루영상을 새로 만들었다.
Fig. 36. Software to make coronal and sagittal images of segmented images with graphic
Fig. 37. A couple of sagittal serially-sectioned images and segmented images (top row); a
E. 부피재구성해서 부피 3 차원영상을 만듦. 1. 구역화영상을 임시구역화영상(PSD 파일)으로 바꿈. 구역화영상(TIFF 파일)의 테두리를 본래의 임시구역화영상(PSD 파일)으로 바꾸었다. Photoshop 에서 구역화영상을 열고 마술막대기도구를 써서 구역화영상의 테두리를 본래대로 만들었다. 보기를 들어서 소뇌의 구역화영상(081.tif, 082.tif…140.tif)을 Photoshop 에서 열고 마술막대기도구를 고른 다음에 포인터를 피부의 구역화영상의 구석에 놓고 딸각해서 구역화영상에 있던 피부의 테두리를 임시구역화영상의 테두리로 바꾸었다. 이 일을 액션에 기록해서 일괄처리하였다. 2. 각 구조물의 부피 3 차원영상을 만듦. MRIcro 에서 부피재구성하기 위해 연속절단면영상의 화소크기, 빛깔개수를 조절하였다. 연속절단면영상을 1 mm 간격으로 추렸기 때문에 화소크기가 0.1 mm 이면 직사각형인 화적소가 만들어진다. 따라서, 정사각형의 화적소를 만들기 위해 화소크기를 0.1 mm 에서 1 mm 로 늘였다. 빛깔개수를 48 bit color 에서 16 bit gray 로 줄였다(Fig. 38).
Fig. 38. A serially-sectioned image (left), which is changed grayly for volume reconstruction
(right).
임시구역화영상(PSD 파일)을 써서 연속절단면영상의 구조물 바깥을 모두 검은 색으로 만들었다. 부피 3 차원영상을 만들기 위해서는 구조물 바깥을 모두 검은색으로 만들어야 한다. 따라서 임시구역화영상에서 구조물의 테두리를 선택하고 반전시킨 다음에 ‘paint bucket’ 도구를 써서 검은색으로 칠하였다(Fig. 38).
머리 전체의 부피 3 차원영상을 만들었다. 부피재구성하기 위해 공개소프트웨어인 MRIcro 버전 1.4 (MRIcro)를 사용하였다. 연속절단면영상에서 보이는 피부 바깥의 포매제를 구역화영상을 써서 자동으로 지운 다음에 쌓았다. 이 결과로 화적소 크기가 1 mm, 빛깔개수가 16 bit gray 인 머리구조물의 부피 3 차원영상을 만들었다. 부피재구성했기 때문에 어느 방향에서도 잘라서 볼 수 있었다(Fig. 39).
Fig. 39. Volume model of head, which were sectioned to display the sectional planes. 머리에 있는 여러 구조물의 부피 3 차원영상을 만들었다. 보기를 들어서 소뇌의 부피 3 차원영상을 만들기 위해 소뇌 바깥의 구조물을 자동으로 지운 다음에 쌓았다. 이 결과로 소뇌의 부피 3 차원영상을 만들었다(Fig. 40). 여러 구조물의 부피 3 차원영상을 한꺼번에 만들기도 하였다. 보기를 들어서 대뇌, 소뇌, 뇌줄기, 시신경, 안구의 부피 3 차원영상을 만들기 위해 이 구조물들 바깥의 구조물을 자동으로 지운 다음에 쌓았다. 이 결과로 대뇌, 소뇌, 뇌줄기, 시신경, 안구의 부피 3 차원영상을 한꺼번에 만들 수 있었다(Fig. 40). 부피 3 차원영상을 돌려서 볼 수 있었다. 부피 3 차원영상이 해부학 지식에 들어맞는지 확인하였다(Fig. 40)(Moore 등, 2006).
Fig. 40. Volume model of brain, eyeballs, and optic nerves (top row); those of lentiform
F. 표면재구성해서 표면 3 차원영상을 만듦.
1. 임시구역화영상의 테두리를 벡터화함.
임시구역화영상(PSD 파일)의 테두리를 벡터화해서 임시구역화영상(AI 파일)으로 바꾸었다. Photoshop 에서 make work path 도구를 써서 임시구역화영상(PSD 파일)의 테두리를 벡터 형식으로 바꾸었다. 이 때 한계치를 정해야 한다. 임시구역화영상에서 테두리에 빛깔을 칠할 때의 선의 굵기를 2 화소로 정한 것처럼, 한계치를 2 화소로 정하였다(Fig. 41)(Park 등, 2007). 테두리를 벡터 형식으로 바꿀 때 한계치는 구역화영상의 테두리에 있는 기준점의 사이 거리이다. 한계치가 2 화소보다 작으면 기준점이 많아지기 때문에 파일 크기가 커진다(Fig. 41). 한계치가 2 화소보다 크면 기준점이 적어지기 때문에 테두리의 모양이 왜곡될 수 있다(Fig. 41). 따라서 테두리의 빛깔을 칠할 때 선의 굵기를 2 화소로 하는 것이 가장 좋았다(Fig. 41). 벡터 형식으로 바꾼 테두리를 AI(Adobe Illustrator) 파일로 저장하였다. 이 결과로 소뇌의 구역화영상을 임시구역화영상(081.ai, 082.ai,…140.ai)으로 바꾸었다. 이 일을 액션에 기록해서 일괄처리하였다.
Fig. 41. Comparison of tolerance during vectorization of the outline in temporary
segmented image. Tolerance is 2 pixel (left), 1 pixel (center), and 10 pixel (right).
2. 테두리를 쌓아서 등고선영상을 만듦.
임시구역화영상(AI 파일)에 있는 한 구조물의 테두리를 모두 쌓아서 등고선영상을 만들었다. 등고선영상을 만들기 위해는 임시구역화영상에 있는 구조물의 테두리를 위아래 비율에 맞게 쌓아야 한다. 보기를 들어서, 소뇌의 임시구역화영상 081.ai 를 열어서 Z 축으로 81 mm 만큼 옮기고, 082.ai 를 열어서 Z 축으로 82 mm 만큼 옮겼다. 소뇌의 나머지 구역화영상(083.ai, 084.ai, …, 140.ai)을 같은 방법으로 모두 열어서 Z 축으로 옮겼다. 이처럼 비슷한 일을 되풀이할 때에는 Maya(2008 판, Autodesk MayaTM, 이하 Maya)에서 스크립트를 미리 만들어서 실행하였다(Table 4)(Wilkins 등, 2005). 이 결과로 소뇌의 모든 구역화영상을 1 mm 간격으로 쌓아서 소뇌의 등고선영상을 만들었다. 이 등고선영상을 DXF(drawing exchange format)파일로 저장하였다.
Table 4. Maya script used for opening and moving outlines of the cerebellum to construct a
outline model
illustratorCurves -ch 0 –ifn "c:/cerebellum/081.ai"; move -r 0 0 81; illustratorCurves -ch 0 –ifn "c:/ cerebellum /082.ai"; move -r 0 0 82; …
illustratorCurves -ch 0 –ifn "c:/ cerebellum /140.ai"; move -r 0 0 140; 3. 등고선 사이에 면을 채움. 등고선영상의 등고선 사이에 면을 채우는 방법을 몇 가지 개발하였다. 위아래 등고선이 겹치는 경우에는 부피재구성을 거쳐서 등고선 사이에 면을 자동으로 채우는 방법을 개발하였다. 이 방법은 등고선영상을 부피재구성하는 방법과 구역화영상을 쌓고 부피재구성하는 방법으로 나눌 수 있었다. 위아래 등고선이 겹치지 않는 경우에는 부피재구성을 거치지 않고 등고선 사이에 면을 반자동으로 채우는 방법을 개발하였다. 이 때 구조물이 갈라지지 않으면 한꺼번에 표면재구성하였고, 구조물이 갈라지면 따로 표면재구성해서 합쳤다(Table 5).
Table 5. Different methods to fill the gaps between stacked outlines in order to perform
surface reconstruction Volume
reconstruction Method (Software) Procedures Volume reconstruction
was done after stacking outlines (AutoCAD).
1. Each outline of a structure was packed. 2. Volume model was made by extruding each packed outline of a structure until next packed outline.
3. Surface model was made by extracting surface from volume model.
In case that neighboring outlines are overlapped, volume reconstruction
was done. Volume reconstruction was done after
stacking segmented images (3D-doctor).
1. Volume model was made by extruding each segmented image of a structure until next segmented image.
2. Surface model was made by extracting surface from volume model.
In case that structure has no dividing region, surface reconstruction was done at once (Rhino).
1. Gaps between stacked outlines of a structure were filled with surfaces.
2. Uppermost and lowermost outlines of a structure were filled with surfaces. In case that
neighboring outlines are not overlapped, volume reconstruction was not done.
In case that structure has dividing region, surface reconstruction was done separately (Rhino).
1. Gaps between stacked outlines of each branch were filled with surfaces.
2. Uppermost and lowermost outlines were filled with surfaces.
3. Branches of a structure was combined.
AutoCAD 에서 부피재구성을 거쳐 등고선 사이에 면을 채우는 과정은 다음과 같았다. 부피재구성하기 위해는 등고선의 속이 채워져 있어야 한다. 따라서 AutoCAD 에서 ‘region’ 명령을 써서 모든 등고선의 속을 동시에 채웠다(Fig. 42)(Table 5). ‘extrude’ 명령을 써서 속이 채워진 모든 등고선을 다음 등고선까지 밀었다(이진천
등, 2007). 이 결과로, 여러 기둥이 만들어졌다(Fig. 42). ‘combine’ 명령을 써서 모든 기둥을 한 개로 합쳤다. 이 결과로 한 개의 부피 3 차원영상을 만들었다(Fig. 42). 부피 3 차원영상을 DXF 파일로 저장하였다. 부피 3 차원영상에서 표면을 꺼내기 위해 3ds max 에서 DXF 파일을 열고, ‘cap closed objects’명령을 썼다. 이 결과로 그 구조물의 표면 3 차원영상을 만들었다(Fig. 42)(Table 5). 이 표면 3 차원영상을 DXF 파일로 저장하였다.
Fig. 42. Surface reconstruction of a schematic structure with three outlines (top row) and
that of the cerebellum (bottom row), where neighboring outlines are overlapped. From the
left to the right represents outlines model, packed outlines model, columns volume model,
columns surface model, and smoothed surface model.
3D-doctor 에서 부피재구성을 거쳐 등고선 사이에 면을 채우는 과정은 다음과 같았다. 3D-doctor 에서 한 구조물의 모든 구역화영상을 연 다음에, ‘surface reconstruction’ 명령을 써서 모든 구역화영상을 다음 구역화영상까지 밀어서 여러 기둥을 만들었다. 모든 기둥을 한 개로 합치고, 부피 3 차원영상에서 표면을 꺼내는