저작자표시-비영리-변경금지 2.0 대한민국 이용자는 아래의 조건을 따르는 경우에 한하여 자유롭게 l 이 저작물을 복제, 배포, 전송, 전시, 공연 및 방송할 수 있습니다. 다음과 같은 조건을 따라야 합니다: l 귀하는, 이 저작물의 재이용이나 배포의 경우, 이 저작물에 적용된 이용허락조건 을 명확하게 나타내어야 합니다. l 저작권자로부터 별도의 허가를 받으면 이러한 조건들은 적용되지 않습니다. 저작권법에 따른 이용자의 권리는 위의 내용에 의하여 영향을 받지 않습니다. 이것은 이용허락규약(Legal Code)을 이해하기 쉽게 요약한 것입니다. Disclaimer 저작자표시. 귀하는 원저작자를 표시하여야 합니다. 비영리. 귀하는 이 저작물을 영리 목적으로 이용할 수 없습니다. 변경금지. 귀하는 이 저작물을 개작, 변형 또는 가공할 수 없습니다.
이학
석사학위 논문
DNA 손상 신호 체계에서 크로마틴
리모델링
인자인 Rsf-1의 역할
아
주 대 학 교 대 학 원
의생명과학과
/종양생물학전공
조
수 진
DNA 손상 신호 체계에서 크로마틴
리모델링
인자인 Rsf-1의 역할
지도교수
조 혜 성
이
논문을 이학 석사학위 논문으로 제출함.
2014년 2월
아
주 대 학 교 대 학 원
의생명과학과
/종양생물학전공
조
수 진
조수진의
이학 석사학위 논문을 인준함.
심사위원장
조 혜 성 인
심 사 위 원
이 영 수 인
심 사 위 원
강 호 철 인
아
주 대 학 교 대 학 원
2013년 12월 02일
- 국문요약 -
DNA 손상 신호 체계에서 크로마틴 리모델링 인자인 Rsf-1 의
역할
RSF 복합체는 뉴클레오솜 이동에 관여하는 크로마틴 리모델링 인자로서 ATPase 효소 기능을 가지고 있는 SNF2h 와 Rsf-1 으로 구성되어 있다. 크로마틴 리모델링 인자들은 DNA 상에서 뉴클레오솜을 이동시키거나 뉴클레오솜을 구성하는 히스톤을 교체함으로써 DNA 복제과정이나 RNA 합성과정에 영향을 미친다는 것이 알려져 있다. 최근에 크로마틴 리모델링 인자들이 DNA 손상 시 신호 체계나 손상 복구에 관여한다는 것이 보고되기 시작하고 있다. 본 연구에서는 Rsf-1 이 DNA 손상 신호에 관여하는 지 그리고 그 작용기전에 대해 연구하고자 하였다. Rsf-1 이 DNA 손상에 관여하는지를 알아보기 위한 방법으로 우선 DNA 손상부위로 이동하는 지를 조사하였다. Laser microirradiation 방법을 사용하여 DNA 에 손상을 준 부위에 Rsf-1 이 이동하여 축적되는 것을 관찰하였다. 또한, FokI system 을 이용하여 살아있는 세포의 염색체 특정부위에 이중나선절단을 유도하였을 때에도 Rsf-1 이 DNA 손상부위에 축적되었다. Rsf-1 이 DNA 손상부위에 축적되는 것은 결합체인 SNF2h 비의존적으로 일어나며, Rsf-1 의 PHD (plant homeodomain)도메인이 중요하다는 것을 확인하였다. 또한 DNA 손상신호의 초기 마커인γ-H2AX 보다 빨리 DNA 손상부위로 이동하고, Rsf-1 의 발현이 저하된 경우에는 γ-H2AX 의 축적이 저하되는 것으로 보아 DNA 손상시 초기에 작용할 것으로 예상된다. 흥미롭게도 Rsf-1 이 빠른 시기에 축적이 되는 것은 PARP1 억제제를 통하여 PARP1 활성에 의존하여 이루어진다는 것을 관찰하였다. 따라서 본 연구는 Rsf-1 이 DNA 손상 신호 체계에 관여하며 PARP1 활성에 의해 DNA 손상 부위로 모이게 되고 DNA 손상 신호를 조절한다는 것을 밝히고 있다. 핵심어 : DNA damage, chromatin remodeling factor, Rsf-1, PARP1, DNA damage checkpoint
차
례
국문요약 ··· ⅰ 차례 ··· ⅲ 그림차례 ··· ⅳ . Ⅰ 서론 ··· 1 . Ⅱ 재료 및 방법(혹은 연구대상 및 방법) ··· 5A. Cell culture, reagent and treatment ··· 5
B. Plasmids and RNA interference ··· 5
C. Laser micro-irradiation ··· 6 D. Immunofluorescence microscopy ··· 6 E. Antibodies ··· 7 F. FokI assay ··· 7 G. Survival assay ··· 7 . Ⅲ 결과 ··· 9
A. Rsf-1 is accumulated to the regions of DNA damage and remained ··· 9
B. Rsf-1 is recruited to the DNA damage sites ··· 10
C. Rsf-1 contributes to SNF2h recruitment to DNA damage sites ··· 11
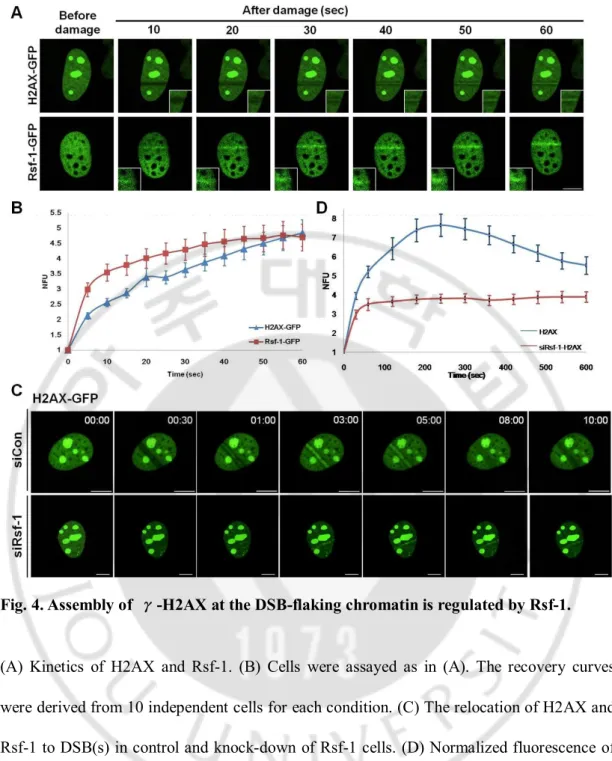
D. Assembly of γ-H2AX at the DSB-flanking chromatin is regulated by Rsf-1 ··· 12
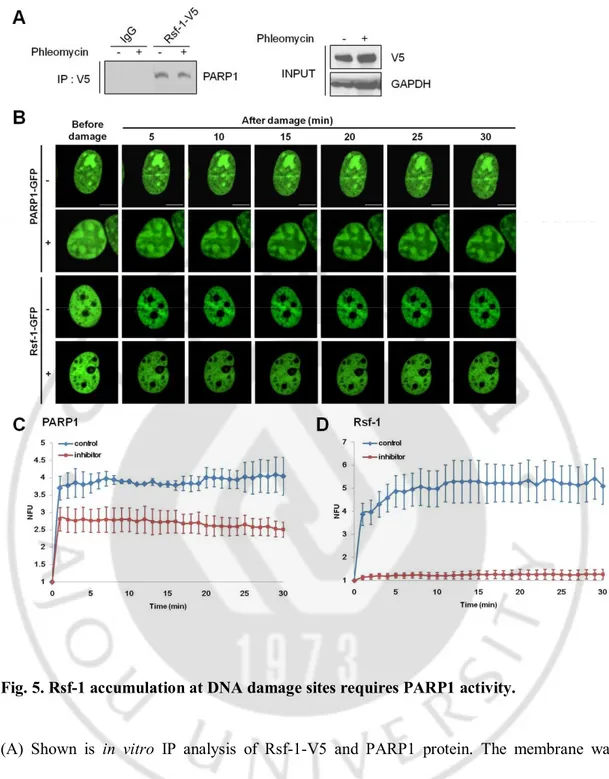
E. Rsf-1 accumulation at the DNA damage sites requires PARP1 activity ··· 12
G. Depletion of Rsf-1 diminishes cell survival and DNA damage checkpoint signals 14 . Ⅳ 고찰 ··· 26 . Ⅴ 결론 ··· 31 참고문헌 ··· 32 ABSTRACT ··· 38
그림
차례
Fig. 1. Rsf-1 is accumulated to the regions of DNA damage and remained
··· 16 Fig. 2. Rsf-1 is recruited to the DNA damage sites
··· 18 Fig. 3. Rsf-1 contributes to SNF2h recruitment to DNA damage sites
··· 19 Fig. 4. Assembly of γ-H2AX at the DSB-flanking chromatin is regulated by Rsf-1
··· 21 Fig. 5. Rsf-1 accumulation at the DNA damage site requires PARP1 activity
··· 23 Fig.6. PHD domain of Rsf-1 is required for the DNA damage response
··· 24 Fig. 7. Depletion of Rsf-1 diminishes cell survival and DNA damage checkpoint signals
··· 27
I. 서 론
세포는 DNA 복제 오류와 같은 내부적 요인과 화학물질, UV 또는 방사선과 같은 외부적 요인에 의해서 끊임없이 DNA 손상에 노출되어 있다. DNA 이중 가닥 절단(double strand break : DSBs)과 같이 DNA 가 손상되면 세포는 세포 주기 체크 포인트 시스템과 DNA 손상 복구 조직을 작동 시켜서 DNA 손상을 복구시키게 된다. DNA 손상 복구과정이 일어나는 동안 세포 내에서는
ADP 리보실화 (ADP-ribosylation), 인산화 (phosphorylation), 유비퀴틴화 (ubiquitination), 메틸화 (methylation), 아세틸화 (acetylation), 수모화 (sumoylation) 와 같은 단백질 수식화(modification)들이 발생한다(Smeenk et al. 2013). 예를 들어, DNA 이중 가닥 절단이 일어나면 poly(ADP-ribose) polymerase 1 (PARP1) 이 활성화 되어 DNA 손상 부위 에 poly(ADP-ribose) 사슬 (PAR chain)을 만들어 DSB 센서인 MRN 복합체를 DNA 손상 부위에 모이게 만든다(Haince et al. 2008). 그 다음 DNA 손상 반응 인산화 효소인 ATM 에 의해서 히스톤 변형체인 H2AX 가 인산화 되며(Rogakou et al. 1998; Burma et al. 2001) DNA 손상 복구 신호들을 전달시킨다(Stucki et al. 2005). 진핵생물 DNA 는 뉴클레오솜에 감싸져서 염색체 구조를 이루는데, 이는 DNA 손상이 발생하였을 때 DNA 복구 조직을 효과적으로 작동하게 하는데 용이하게 하기 위함이다. DNA 손상이 발생하게 되면 크로마틴은 더 조밀하게 되거나 헐거워지게 되는 리모델링 과정을 거치게 되는데, 이때 ATP 가수분해 단백질들이 관여한다(Lans et al. 2012). 최근에는 크로마틴 리모델링 인자들이 DNA 손상 신호 전달과 복구 체계에 중요한 역할을 한다는 것이 밝혀지고 있다.
크로마틴 리모델링 인자 중 SNF/SWI family 는 H2AX 와 H3 의 아세틸화(acetylation)를 통해 DNA 손상 복구가 용이하도록 만든다는 것이 알려져 있다(Lee et al. 2010). 또한 ISWI family 에 속하는 ACF1 은 DNA 손상 복구 단백질과 상호작용을 통하여 복구하는데 중요한 역할을 한다고 보고된 바 있다(Lan et al. 2010).
RSF 복합체는 SNF2h 와 Rsf-1 으로 이루어져 있다. SNF2h 는 ATP 를 가수분해하여 뉴클레오솜 구조를 변화시킬때 필요한 에너지를 공급한다고 알려져있다(Loyola et al. 2003). Rsf-1 은 잘 보존된 plant homeodomain (PHD) 을 가지고 있는데 다른 단백질들과의 상호작용을 통해 전사 활성화 인자(Shamay et
al. 2002) 또는 억제 인자(Shamay et al. 2002; Huang et al. 2004)의 역할을 한다고
알려져 있다. 다른 연구에서는 centromere protein A (CENP-A) 와 결합을 통해서 세포주기 중 mid-G1 phase 때 CENP-A 를 동원체 부분으로 재구성 하는데 중요하다고 보고되었다(Perpelescu et al. 2009). Rsf-1 은 임상에서도 연구가 되어졌는데 난소암과 유방암 등에서 발현이 높아서 종양 진행에 있어 중요한 역할을 하는 암 유전자로 알려져 있다(Shih et al.2005; Mao et al. 2006; Davidson et al. 2006; Choi et al. 2009). 하지만 아직까지 어떠한 기전으로 인해서 Rsf-1 이 cancer 에서 작용을 하는지는 아직 불분명하다. 또한 Rsf-1 이 과발현되면 DNA 손상과 유전체불안정성을 유도한다고 알려져 있는데(Sheu et al. 2010) DNA damage 상에서 정확한 기능과 작용 기전에 대해서는 알려진 바가 불분명하다.
본 연구에서는 Rsf-1 을 이입한 세포에서 microirradiation 과 FokI system 으로 DNA 손상을 발생시킨 다음 손상 부위로 모이는 것으로 크로마틴 리모델링 인자인 Rsf-1 이 DNA 손상 신호 체계에 연관 되어 있음을 확인할 수
있었다. Rsf-1 도메인 연구를 통해 DNA 손상 부위로의 모임에 있어서 PHD 부분이 중요한 것을 밝혔고, 또한 결합 파트너인 SNF2h 와 DNA 손상 복구 단백질인 γ-H2AX 의 응집을 용이하게 하는데 작용한다는 것을 확인할 수 있었다. Rsf-1 이 빠른 시간 내에 DNA 손상 부위로 모이는데 있어서 PARP1 의 활성 의해 생성된 Poly(ADP-ribosyl)ation 이 중요하다는 것을 PARP1 억제제를 처리하여서 관찰하였다. 따라서 본 연구는 Rsf-1 이 Poly(ADP-ribosyl)ation 의존적으로 DNA 손상 부위로 모이게 되고 DNA 손상 신호 체계를 조절한다는 것을 밝히고 있다.
Ⅱ
. 재료 및 방법
A. Cell culture, reagent and treatment
U2OS (human osteosarcoma) 와 293T (human embryonic kidney) 세포는 10% fetal bovine serum (FBS, Gibco) 을 넣은 DMEM (Dulbecco's Modified Eagle Medium)을 사용하여 37℃, 5% CO₂ 배양기에서 배양하였다. RPE-1 (retinal pigment epithelial cells) 세포는 10% FBS, 10 µg/ml hygromycin 이 포함된 F12 (Gibco) 배양액을 사용하여 37℃, 5% CO₂ 배양기에서 배양하였다. Rsf-1 이 knockdown 된 세포를 만들기 위해서 lentiviral system 을 이용하여 Rsf-1-shRNA 를 U2OS 세포에 감염시킨 다음 10 µg/ml puromycin 을 첨가하여 4 주동안 Rsf-1-shRNA 가 들어간 세포만을 선택하였다. PARP1 inhibitor 인 AG-4361 은 laser-microirradiation 하기 1 시간 전에 처리 하였다.
B. Plasmids and RNA interference
GFP 가 tag 된 Rsf-1 plasmid 를 만들기 위해, Dr. Ie-Ming Shih (Johns Hopkins Medical Institutions) 로부터 받은 pcDNA6-Rsf-1-V5 construct 를 사용하여 pEGFP-N1
에 Rsf-1 을 넣는 sub-cloning 을 하였다. H2AX-GFP 는 권종범
박사(이화여자대학교), SNF2h-GFP 는 Akira Yasui 박사(도호쿠대학교), PARP1-GFP 는 강호철 박사(아주대학교) 로부터 받았다.
RNA-interference 는 전기충격 방법을 사용하여 세포에 이입시켰다. Rsf-1 의 siRNA(Ambion) 의 서열 : 5′-GAAAAUGUCAAACCCAUU-3′.
C. Laser micro-irradiation
Live cell imaging 을 하기 위해 35-mm round glass bottom dishes(SPL)에 세포를 키우고 각각의 단백질에 GFP 가 tagging 된 DNA 를 이입하고 micro-irradiation 하기 30 시간 전에 10mM 의 5-bromo-2-deoxyuridine (BrdU) 를 처리하였다. Nikon A1 공초점 현미경의 60x oil 렌즈와 temperature-controlled chamber 를 사용하여 37℃, 5% CO₂ 조건을 맞추어서 imaging 을 하였다. 405 nm laser 를 사용하여 1.2 초 (32 line/sec) 동안 irradiation 을 준 후 10 분 또는 30 분 동안 1 초 간격으로 imaging 하였다.
D. Immunofluorescence microscopy
35-mm glass bottom dish 에 키운 세포를 laser micro-irradiation 한 후 PBS (phosphate buffered saline) 로 3 번 세척하고 4 % paraformaldehyde (in PBS) 로 고정시켰다. 0.1% TritonX-100 (in PBS) 로 permeablization 시킨 후 3 % BSA (in PBS) 로 상온에서 30 분 동안 blocking 하였다. 보고자 하는 단백질에 대한 1 차 항체를 상온에서 1 시간 반응 시키고 PBS 로 3 회 세척한 후 형광물질 (Alexa 488, 594) 이 결합된 2 차 항체를 30 분 상온에서 반응 시켰다. DNA 관찰을 위해 DAPI 를
이용하여 염색한 후 PBS 로 3 번 세척하고, 슬라이드 글라스 위에 고정 한 후 레이저 공초점 현미경 (Nikon A1) 을 이용해 관찰하였다.
E. Antibodies
Rsf-1, pATM (S1981) 등에 대한 항체는 Abcam 에서 구입하였고, γH2AX, SNF2h 에 대한 항체는 Millipore 에서 구입하였다. pATR(S428), pChk1(S296), pChk2(T68) 에 대한 항체는 Cell signaling 에서 구입하였다. GAPDH 항체는 Santacruz, V5 는 Invitrogen 에서 구입하였다. HRP 가 결합된 mouse 와 anti-rabbit 항체는 Invitrogen 에서 구입하였다.. 형광물질이 결합된 항체, Alexa Fluor 488, Alexa Fluor 594 는 Molecular Probes 에서, DAPI 는 Invitrogen 에서 구입하였다.
F. FokI assay
Shanbhag et al.(2010) 에서 사용한 세포주와 방법을 이용하여서 실험을 실시하였다. U2OS 2-6-3 세포에 GFP 가 tag 된 Rsf-1 mutant 와 FokI-mCherry-LacI 또는 FokI-mCherry-LacI-D450A 를 이입시켰다. 48 시간 후 4 % paraformaldehyde 로 세포를 고정 시킨 다음 Nikon A1 공초점 현미경으로 관찰하였다.
U2OS 에 siRNA 를 사용하여 Rsf-1 의 발현을 저하시킨 다음 6 well plate 에 각각의 세포수를 세어서 깔아주었다. Phleomycin, MMS, HU(sigma)를 1 시간 처리한 다음 다시 배양액으로 바꿔준다. 그 다음 37℃, 5% CO₂ 배양기에서 10 일 동안 배양하였다. 6% glutaraldehyde 와 0.5% crystal violet 으로 염색 시킨 후 콜로니의 수를 세어서 생존율을 확인하였다.
Ⅲ
. 결 과
A. Rsf-1 is accumulated to the region of DNA damage and remained
DNA 손상 신호 단백질들의 특징 중의 하나는 DNA 손상부위로 빠르게 이동한다는 것이 잘 알려져 있다. DNA 손상 신호 및 복구에 관여하는 단백질들은 일차적으로 DNA 손상 부위로 이동하는 것이 알려졌지만 이들 단백질이 어떤 역할을 수행하느냐에 따라 DNA 손상 부위로 이동하는 시점이 각각 다르다. 크로마틴 리모델링 인자로 알려진 Rsf-1 이 DNA 손상이 발생하였을 때, DNA 손상 신호 체계 영향을 미치는 지 알아보고자 하였다. 따라서, U2OS 세포에서 laser microirradiation 을 사용하여 DNA 에 손상을 준 후 Rsf-1 이 DNA 손상부위로 이동하는지 조사하였다. kinetics 를 확인하여 Rsf-1 이 어떤 시점에 DNA 손상 부위로 모이는 것인지 확인해보고자 하였다. DNA 손상 직후 H2AX, Rsf-1, SNF2h 는 모두 2 분 안에 손상 부위로 모이는 것을 관찰하였다. 또한, 흥미롭게도 H2AX 는 DNA 손상 후 6 분이 되었을 때 가장 많이 모이고 10 분이 지났을 때 손상 부위에 모인 것이 약화되어 30 분 후에는 거의 남아있지 않지만(Movie1) Rsf-1 은 꾸준히 손상 부위로 이동하여 10 분 까지도 손상 부위로 모이고 30 분 후에도 손상 부위에 남아있는 것을 확인하였다(Movie2). SNF2h 는 30 초까지 손상 부위로 이동한 것이 30 분 후에도 여전히 손상 부위에 남아있는 것을 확인할 수 있었다(Figure 1A and Movie3). 또한, RPE-1 세포에서도 Rsf-1 이 10 분까지 손상 부위에 남아있는 것을 확인 하였다(Figure 1B). 이 결과는 Rsf-1 은 DNA 가 손상되었을 때 빠른 시간에 손상 부위로 모여서 크로마틴 구조를
변화시키는데 역할을 수행할 가능성을 제시하고 있으며, 또한 DNA 손상 복구 작용에도 영향을 끼칠 수 있을 가능성을 제시하였다.
B. Rsf-1 is recruited to the DNA damage sites
다음으로 laser microirradiation 으로 DNA 에 손상을 준 후 면역형광염색법을 이용하여 조사하였다. DNA 손상되면 손상 부위로 이동한다고 알려진 γ-H2AX 단백질 또는 인산화된 ATM 을 동시에 검출하였다. DAPI 로 염색이 되는 DNA 부위에 microirradiation 에 의해 길게 DNA 손상이 발생하였고, 이 부위에 γ-H2AX 와 Rsf-1 또는 p-ATM 과 Rsf-1 이 동시에 검출되었다(Figure 2A). 다음으로, V5 로 표지된 Rsf-1 유전자를 U2OS 세포에 이입시켜 과발현 시킨 후 이들 Rsf-1 또한 DNA 손상 부위로 이동하여 축적되는 것을 확인할 수 있었다(Figure 2B). microirradiation 은 DNA 이중 나선 손상뿐만 아니라 외가닥 절단 또한 발생하기 때문에 DNA 이중 나선 절단만 만들 수 있는 시스템을 사용하여서 다시 한번 확인해 보았다. 1 번 염색체에 LacO 반복 서열이 있어 mCherry 가 tag 된 핵산내부가수분해효소(endonuclease) LacI-FokI 를 넣어주면 하나의 이중나선 절단(single double-strand break)을 발생 시킬 수 있는 세포주를 사용하여 확인해 보았다. 이때 GFP 가 표지된 Rsf-1 을 넣어주어서 DNA 손상 부위로 Rsf-1 이 모이는 것을 관찰할 수 있었다(Figure 2C). 또한, 이러한 현상이 세포 특이적인 현상이 아니라 정상세포에서도 일어나는 지를 확인하기 위하여 정상 세포인 RPE-1 세포에서도 microirradiation 을 통해 확인하였다. RPE-1 세포에서도 DNA 손상 부위에 γ-H2AX 와 Rsf-1 이 동시에 검출됨으로써 세포 내에 DNA 가 손상
되었을 때 크로마틴 리모델링 인자인 Rsf-1 이 손상 부위로 이동한다는 것을 확인할 수 있었다(Figure 2D, upper panel). 또한, Rsf-1 의 결합체인 SNF2h 단백질이 Rsf-1 과 함께 DNA 손상 부위로 이동하여 축적되는 것을 확인하였다(Figure 2D, lower panel).
C. Rsf-1 contributes to SNF2h recruitment to DNA damage sites
Rsf-1 의 결합체인 SNF2h 는 Rsf-1 이외에도 다른 여러 단백질들과 결합하여 크로마틴 리모델링 인자로서의 역할을 수행할 수 있다. 따라서 Rsf-1 이 DNA 손상 부위로 이동하는 것이 SNF2h 와의 SNF2h 의존적으로 일어나는 지를 확인하고자 하였다. 이를 위해 siRNA 를 사용해 SNF2h 의 발현을 저하시킨 다음 Rsf-1-GFP 를 넣어준 후 손상 부위로 모이는 시점을 확인해보았다. Rsf-1 은 microirradiation 후 10 초 후에 빠르게 DNA 손상 부위로 축적되고 10 분까지 유지되었다. 또한, SNF2h 의 발현을 저하시킨 세포에서도 Rsf-1 이 정상적으로 DNA 손상 부위에 빠르게 축적되고 유지되었다. 따라서, Rsf-1 이 DNA 손상 부위로 빠르게 이동할 때에는 SNF2h 비의존적으로 일어난다는 것을 확인하였다 (Figure 3A, B, C). 한편, SNF2h 가 DNA 손상 부위로 이동하는 것이 Rsf-1 의존적으로 일어나는 지를 조사하였다. SNF2h 는 10 초까지 빠르게 축적되어 이후 10 분까지 유지되었다. 반면 Rsf-1 의 발현을 siRNA 를 사용하여 저하시킨 세포에서 SNF2h 를 관찰하였을 때, 대조군에 비해 SNF2h 의 축적이 약화되는 것을 관찰하였다(Figure 3D, E, F). 이러한 결과들을 통해 Rsf-1 은 SNF2h 를 DNA 손상 부위로 모집시키는데 영향을 미치나 SNF2h 와는 독립적으로 DNA 손상 부위로 이동한다는 것을 알 수 있었다.
D. Assembly of γ-H2AX at the DSB-flaking chromatin is regulated by Rsf-1
앞선 결과에서 Rsf-1 이 비교적 빨리 DNA 손상 부위로 모이는 것을 관찰하였고(Figure 1) DNA 손상 반응에서 빠른 시간 내에 관여한다고 알려져 있는 H2AX 와 손상 부위로 모이는 시간을 비교해 보았다. 그 결과 Rsf-1 이 H2AX 보다 더 빨리 DNA 손상 부위로 모이는 것을 확인 할 수 있었다(Figure 4A, B). Rsf-1 은 크로마틴 리모델링 인자 중 하나이기 때문에 DNA 손상이 발생하였을 때 바로 손상 부위로 모여 크로마틴을 열어주거나 히스톤을 교체시켜 H2AX 를 DNA 손상 부위로 모이게 도와줄 것이라는 가설을 세우고, 그것을 증명하기 위하여 siRNA 를 사용하여 Rsf-1 을 knock-down 시킨 다음 H2AX 의 변화를 관찰하였다. H2AX 의 경우 5 분까지 꾸준히 DNA 손상 부위로 이동하고 그 후 축적이 감소된 반면, Rsf-1 발현이 저하되면 H2AX 가 DNA 손상 부위로 축적이 일어나지 않는 것을 확인하였다(Figure 4C, D). 이러한 결과들은 손상된 크로마틴 부위로 H2AX 가 이동하는데 있어서 Rsf-1 이 조절한다는 것을 제시하고 있다.
E. Rsf-1 accumulation at the DNA damage site requires PARP1 activity
그 다음으로 Rsf-1 이 DNA 손상 부위의 어떤 신호를 인지하여 모이는지 확인해보고자 하였다. DNA 손상 시 가장 빠른 신호는 PARP1 에 의한 것으로, 손상 부위에 Poly(ADP-ribosyl) chains (PAR chains) 이 형성되고 이와 결합하여
et al. 2008). Rsf-1 또한 PARP1 과의 연관성이 있을 것이라고 생각되어 제일 먼저
V5 가 표지 된 Rsf-1 을 과발현 시킨 다음 DNA 손상을 주는 약물인 phleomycin 을 처리한 것과 처리하지 않은 세포를 수확하여 면역 침강법을 사용하여 PARP1 과의 결합을 확인해보았다. 그 결과 정제된 PARP1 단백질과 Rsf-1 이 결합하는 것을 확인할 수 있었다(Figure 5A). 또한 PARP1 은 PAR chain 을 형성하여 다음 신호를 전달 시키는데 Rsf-1 역시 이러한 신호를 인식하고 DNA 손상 부위로 모이는지 확인해 보기 위해 PAR chain 을 형성하는 PARP1 의 활성을 없애는 억제제를 처리하고 Rsf-1 의 변화를 관찰하였다. 그 결과 PARP1 의 활성이 감소하게 되면 Rsf-1 이 DNA 손상 부위로 모이는 것이 30% 줄어드는 것을 확인하였다(Figure 5B, C, D). 따라서 Rsf-1 은 PARP1 활성에 의존한 신호를 인식하여 DNA 손상 부위로 모이게 된다는 것을 알 수 있었다.
F. PHD domain of Rsf-1 is required for the DNA damage response
Rsf-1 에는 DNA 에 결합한다고 알려진 DTT domain, Glutamic acid 가 많은 부위, zinc finger PHD domain (Plant homeodomain), Arginine 이 많은 부위 등 특징적인 부분을 가지고 있다(Shamay et al. 2002). DNA 손상 부위로 Rsf-1 이 모이는데 있어서 어떤 부분이 중요한지 mutant 들을 사용하여서 실험을 하였다(Figure 6A). live cell 을 사용하여 kinetics 를 확인해본 결과, WT 의 경우 DNA 손상부위로 잘 축적되는 것을 확인 할 수 있었지만 PHD 도메인이 없는 경우 반으로 줄어드는 것을 확인할 수 있었다. 또한 PHD 가 있는 C-terminus 부분인 D5 의 경우 3.5 배 축적되고 PHD 가 없는 C-terminus 부분인 D6 의 경우는 DNA 손상 부위로 거의 축적되지 않는 것을 관찰할 수 있었다(Figure 6B, C). 그
다음으로는 Singe double-strand break 을 발생시킬 수 있는 LacO-LacI nuclease system 을 사용하여서 다시 한번 확인 해보았다. GFP 가 표지된 Rsf-1 mutant 와 mCherry-LacI-FokI (Wild type 또는 nulcease 기능이 없는 D450A) 를 함께 세포에 이입한 후 세포 내에서 GFP 와 mCherry 가 colocalization 되는 정도를 관찰 하였다(Figure 6D). 예상한 바와 같이 Rsf-1 Wild type 의 경우 71.6%가 DNA 손상 부위로 모이고 D5 와 D6 의 경우 43.5%, 43.9%의 비율로 DNA 손상 부위로 모이는 것을 확인하였다. 이에 반해 PHD 가 없는 mutant 의 경우 24%만이 DNA 손상 부위로 모이는 것을 확인하여 Wild type 에 비해서 DNA 손상 부위로 모이는데 있어서 문제가 발생되는 것을 확인할 수 있었다(Figure 6E). DNA 손상이 유도 되었을 때, Rsf-1 이 그 부위로 모이는데 있어서 PHD domain 이 중요하다는 것을 알 수 있었다.
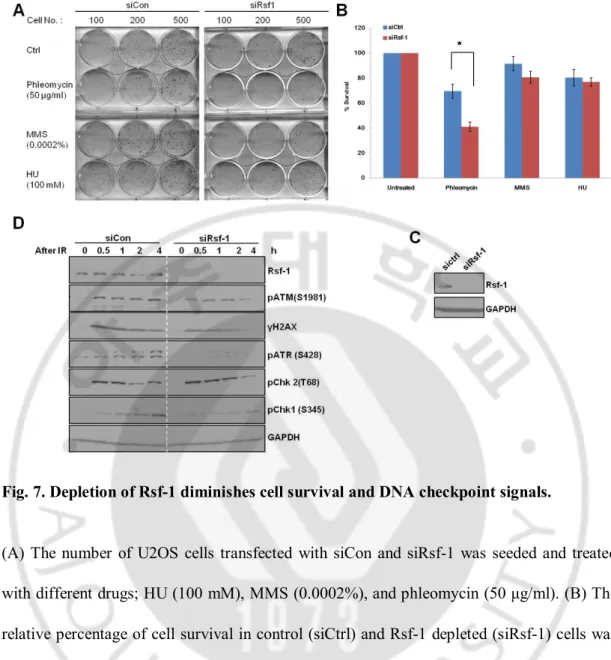
G. Depletion of Rsf-1 diminishes cell survival and DNA damage checkpoint signals
크로마틴 리모델링 인자는 DNA 손상 시 크로마틴 구조를 열어주는 것 뿐만 아니라 DNA 손상이 복구 된 후 다시 크로마틴 구조를 조절시킬 것이라고 생각되고 있다. 앞선 결과에서 DNA 손상 후 30 분까지도 DNA 손상 부위에 남아있는 것을 확인하였고(Figure 2), Rsf-1 이 DNA 손상 복구 체계에도 기능을 할 것이라고 생각하였다. DNA 손상이 발생한 다음 복구가 되기 위해서는 먼저 세포 주기를 멈추게 되는데 세포 주기에 관련된 체크포인트 단백질들 관여한다고 알려져 있다(Niida et al. 2006). 그 기능을 잘 수행하고 있는지 확인해보고자 Ionizing Radiation 을 준 후 각각의 시간 별로 세포를 수확한 다음 체크포인트
활성화 되는 ATR, ATM 의 신호가 30 분부터 4 시간 까지 떨어져 있는 것을 확인하였다. ATR 과 ATM 에 의해 인산화 되어 활성화 되는 체크포인트 단백질인 Chk1, Chk2 또한 4 시간에 현저하게 약화되어져 있는 것을 확인 하였다(Figure 7A). 따라서 Rsf-1 은 DNA 가 손상되면 체크포인트 단백질들의 활성을 조절하여 DNA 손상 복구가 잘 일어날수 있도록 조절 한다는 것을 알 수 있었다. 그 다음으로 Rsf-1 이 없을 때 DNA 손상 복구가 얼마나 잘 일어나는지 확인해보고자 하였다. 세포들에 DNA 를 손상시킬 수 있는 약물들을 처리하고 한 시간 후 배양액을 교체해준 다음 10 일 동안 키운 후 살아남은 세포의 비율을 관찰 하였다. Rsf-1 이 없으면 살아남은 세포가 현저하게 감소되는 것을 확인할 수 있었다(Figure 7B). 외가닥 절단을 유발하는 HU 나 MMS 보다 이중나선절단을 일으키는 Phleomycin 을 처리 하였을 때 손상 복구가 잘 일어나지 않는 것을 확인하였다(Figure 7C). 따라서 Rsf-1 은 DNA 이중나선절단이 발생하였을 때 복구가 잘 일어나도록 작용한다는 것을 알 수 있었다.
Fig. 1. Rsf-1 is recruited to the regions of DNA damage and remained.
Dynamic recruitment of H2AX-GFP, Rsf1-GFP and SNF2h-GFP to DNA damage sites following laser microirradiation. (A) H2AX, Rsf-1 and SNF2h is rapidly recruited to DNA damage sites. Rsf-1 remained after DNA damage upon 10min. (B) Confirm the kinetics of H2AX and Rsf-1 in normal RPE-1 cell line. Scale bars, 10 μm.
Fig. 2. Rsf-1 is recruited to the DNA damage sites.
(A) Endogenous Rsf-1 is recruited to DNA damage sites. DNA damage was generated by laser microirradiation following by immunofluorescence (10 min after damage) with the indicated antibodies. (B) Laser microirradiation induces recruitment of exogenous Rsf-1 to sites of laser-induced DNA damage. (C) Confirm the recruitment of Rsf-1 using FokI system. (D) Confirm the recruitment of γH2AX, SNF2h and Rsf-1 in normal RPE-1 cell line. Scale bars, 10 μm.
Fig. 3. Rsf-1 contributes to SNF2h recruitment to DNA damage sites.
(A) Recruitment of Rsf-1-GFP in the absence of SNF2h. (B) Cells were assayed as in (A). The recovery curves were derived from 10 independent cells for each condition. (C) The level of SNF2h in siSNF2h cells compared to sicontrol, was analyzed by western blot. (D) Kinetics of SNF2h-GFP to DSB sites. (E) The curves was monitored in a time course following laser microirradition. (F) The level of Rsf-1. NFU, normalized fluorescence units. Error bars, SEM. Scale bars, 10 μm.
Fig. 4. Assembly of γ-H2AX at the DSB-flaking chromatin is regulated by Rsf-1.
(A) Kinetics of H2AX and Rsf-1. (B) Cells were assayed as in (A). The recovery curves were derived from 10 independent cells for each condition. (C) The relocation of H2AX and Rsf-1 to DSB(s) in control and knock-down of Rsf-1 cells. (D) Normalized fluorescence of H2AX. NFU, normalized fluorescence units. Error bars, SEM. Scale bars, 10 μm.
Fig. 5. Rsf-1 accumulation at DNA damage sites requires PARP1 activity.
(A) Shown is in vitro IP analysis of Rsf-1-V5 and PARP1 protein. The membrane was denatured and renatured and incubate the membrane with purified bait PAPR1 proteins. (B) Kinetics of PARP1 and Rsf-1 after DNA damage. Cells were treated with (-) and without PARP1 inhibitor (AG-4361) prior to microirradiation. (C, D) Quantification of result in (B). NFU, normalized fluorescence units. Error bars, SEM. Scale bars, 10 μm.
Fig. 6. PHD domains of Rsf-1 is required for the damage response.
(A) Schematic presentation of domains for 1. (B) U2OS cells were transfected with Rsf-1-WT, Rsf-1-ΔPHD, Rsf-1-D5 and Rsf-1-D6 and followed by laser microirradiation. (C) Quantitative analysis in (B). (D) Localization of Rsf-1-GFP mutants and FokI wild type(WT) and FokI mutant (D450A). (E) Quantitative analysis of the single foci percentage. NFU, normalized fluorescence units. Error bars, SEM. Scale bars, 10 μm. *P < 0.5, **P < 0.01, ***P < 0.005 by Student’s t-test.
Fig. 7. Depletion of Rsf-1 diminishes cell survival and DNA checkpoint signals.
(A) The number of U2OS cells transfected with siCon and siRsf-1 was seeded and treated with different drugs; HU (100 mM), MMS (0.0002%), and phleomycin (50 μg/ml). (B) The relative percentage of cell survival in control (siCtrl) and Rsf-1 depleted (siRsf-1) cells was calculated. (C) Level of Rsf-1. (D) U2OS cells were transfected with si control (siCon) or siRNA for Rsf-1(siRsf-1) and harvested at the indicated time points after treated IR (10 Gy). DNA damage checkpoint activation were examined by western blot. *P < 0.5 by Student’s t-test.
Ⅳ
. 고 찰
DNA 가 절단되게 되면 많은 단백질들에 의해 복구 신호 체계가 전달되어진다. 히스톤과 단백질 수식화를 통해 신호를 전달하고, 신호를 인지하여 DNA 손상 부위로 모인 단백질들에 의해 복구가 이루어진다고 알려져 있다. DNA 는 뉴클레오솜에 감싸져서 염색체를 구성하는데, 이 구조를 조절하는데에는 크로마틴 리모델링 인자들이 관여한다. 이를 토대로 최근 DNA 손상 시 DNA 복구 조직을 효율적으로 조절하는데 있어서 손상 신호 전달 단백질과 복구 단백질 이외에도 크로마틴 리모델링 인자들이 연관있다는 연구가 이루어지고 있다. 크로마틴 리모델링 인자 중 하나인 CHD4 의 경우 DNA 가 손상되면 손상 부위로 이동하여 손상 신호를 전달하고 G2/M 체크포인트를 작동시켜 세포사멸을 막아서 DNA 손상 복구가 일어날 수 있게 도와준다고 알려져 있다(Smeenk et al. 2010). 또한 ISWI 에 속하는 WSTF 는 H2AX 의 타이로신 142 번 잔기를 인산화 시키는 새로운 인산화 효소로서의 작용과 이 인산화가 DNA 손상 반응에 중요한 역할을 한다는 것이 밝혀진 바 있다(Xiao et al. 2009). ACF1 도 DNA 이중 나선 절단이 일어났을 경우 손상 부위로 이동하여 DNA 손상 복구에 필요한 인자라는 것이 알려졌다(Lan et al. 2010). 많은 연구가 진행 되었음에도 불구하고 크로마틴 리모델링 인자들이 어떤 신호에 의해서 DNA 손상 부위로 이동하며 손상 복구 체계 이 외에 어떤 작용을 하는지에 대한 연구는 이루어지지 않았다.따라서 본 연구에서는 크로마틴 리모델링 인자인 Rsf-1 이 DNA 가 손상 되었을 때 다른 크로마틴 리모델링 인자들과 어떻게 다르게 작용하며 어떤 단계에서 기능을 하는지 의문을 가지게 되었고 먼저 Rsf-1 이 DNA 손상 부위로 모이는 것을 관찰하였다(Fig. 1). Time lapse 를 통해 U2OS 세포주에서 H2AX 는 5 분에 손상 부위로 모이는 정도가 최고치를 이루었으며 10 분이 지난 후에는 손상 부위에서 거의 보이지 않았다. 흥미롭게도 Rsf-1 과 SNF2h 의 경우에는 30 분까지도 손상 부위에 모여있는 것을 확인할 수 있었다. 정상세포인 RPE-1 세포의 경우 U2OS 세포보다 H2AX 의 단백질이 적어 비교적 늦게 DNA 손상 부위로 모여 10 분째까지 남아 있을 것으로 예상한다(Fig. 2). 이를 통해 Rsf-1 이 DNA 손상이 발생하면 그 즉시 손상 부위로 가서 크로마틴 구조를 변화시키고 DNA 복구가 일어난 후에도 크로마틴 구조를 변화시킬 것이라고 생각해 볼 수 있을것이다. Rsf-1 은 SNF2h 와 함께 RSF 복합체를 구성하는데 둘의 상관관계에 의해서 크로마틴 리모델링 인자의 역할인 뉴클레오솜 구조의 변화를 일으킨다고 알려져 있다. Rsf-1 이 DNA 손상 부위로 모이는 것은 SNF2h 와 관계없이 독립적으로 일어나는 일이며 Rsf-1 이 없을 시 SNF2h 가 DNA 손상 부위로 모이지 않는 것으로 보아 Rsf-1 이 먼저 DNA 손상 부위로 가고 SNF2h 를 손상 부위로 오게 유도하는 역할을 하는 것을 확인 하였다(Fig. 3). 이로써 Rsf-1 은 DNA 손상 시 ATPase SNF2h 가 없어도 어떠한 신호에 의해서 또는 다른 단백질과의 결합을 통해 DNA 손상 신호 체계를 이루고 역할을 할 것이라는 것을 예상할 수 있다. DNA 손상 신호 체계를 구성 하는 것에 있어 가장 중요한 것이 히스톤 변형체인 H2AX 인데, H2AX 는 인산화효소인 ATM 에 의해 인산화 되어 손상 복구 신호를 전달한다고 알려져 있다(Rogakou et al. 1998; Burma et al. 2001; Stucki et al. 2005).
Rsf-1 이 H2AX 보다 먼저 DNA 손상 부위로 모이는 것으로 보아 H2AX 에 의한 신호를 인지하고 DNA 손상 부위로 모이는 것은 아니라는 것을 알 수 있었다(Fig. 4A, B). 또한 Rsf-1 이 뉴클레오솜 구조를 변화시킬 수 있는 크로마틴 리모델링 인자이기 때문에 구조 변화를 통해 손상 부위의 H2AX 가 인산화 되고 다른 히스톤과 교환이 될 가능성이 있다는 것을 생각할 수 있었고, 실험을 통해 Rsf-1 이 없을 경우 H2AX 가 DNA 손상 부위로 모이는 것이 현저하게 약화되는 것을 관찰 함으로써 확인할 수 있었다(Fig. 4C, D). DNA 손상 이후 바로 반응 하는 신호 전달 체계는 PARP1 에 의한 것인데, PARP1 은 주로 DNA 손상 복구에 있어서 중요한 역할을 한다고 알려져 있다. PARP1 이 없을 경우 세포 생존율이 현저하게 감소하고, 비상동적 말단 결합(Nonhomologous end joining, NHEJ)을 일으키는 KU 복합체와 경쟁하여 손상 복구에 관여한다고 보고되었다(Wang et al. 2006; Hochegger et al. 2006; Sugimura et al. 2008). 또한 DNA 이중 가닥 절단이 발생되면 PARP1 이 활성화 되어 DNA 손상 부위 에 poly(ADP-ribose) 사슬 (PAR chain)을 만들어 DSB 센서인 MRN 복합체를 DNA 손상 부위에 모이게 만든다는 것도 보고된 바 있다(Haince et al. 2008). Rsf-1 이 DNA 손상 후 빠른 시간 안에 손상 부위로 모이기 때문에 PARPRsf-1 활성을 인지하는 지 확인해보고자 PARP1 억제제를 처리한 후 Rsf-1 의 변화를 살펴보았다. 그 결과 Rsf-1 이 PARP1 활성 의존적으로 DNA 손상 부위로 모이는 것을 확인할 수 있었다(Fig. 5). PARP1 억제제는 PAR 사슬을 만드는 활성을 억제하는 약물인데 처리하였을 경우 PARP1 또한 DNA 손상 부위로 모이는 것이 약간 감소하는 것으로 보여졌다. 이는 PARP1 이 스스로를 poly(ADP-ribosyl)tion 시키면서 완전하게 활성화 된다고 알려져 있는데 다른 히스톤이나 단백질을
리보실화 시키지 못하는것 뿐만 아니라 스스로도 리보실화 시키지 못해서 DNA 손상 부위로 모이는 것이 감소되었을 가능성도 있다.
Rsf-1 에는 여러 특징적인 부분들이 존재하는데 이중에서 가장 잘 알려진 부위는 PHD 도메인이다. PHD 도메인은 주로 단백질-단백질 결합을 한다고 알려져 있다. Rsf-1 은 C-terminus 부분에 NLS(nucleus localization sequence)가 존재하여 C-terminus 가 없는 경우 세포 내에서 세포질에 발현 되는 것을 알 수 있었다(Fig. 6A). 하나의 DNA 이중 나선 손상을 발생시켜 확인한 결과 wild 타입의 경우 71.6%, D5 와 D6 의 경우 42.5%, 43.9%의 비율로 DNA 손상 부위로 모이지만 PHD 부분이 없을 경우 24% 만이 DNA 손상 부위로 모이는 것을 확인하였고 이는 PHD 부분이 Rsf-1 이 DNA 손상 부위로 모이는 것에 있어서 중요한 역할을 한다는 것을 제시해주고 있다(Fig. 6B, C, D). D3 와 D4 의 경우 세포질에 존재하기 때문에 손상 부위로 모이는 정도를 확인하지 못하였다. 세포 핵 안에 존재할 수 있는 DNA 를 만들어서 확인 해 보면 PHD 도메인이 중요한 것을 좀 더 확신할 수 있을 것이다. 또한 PHD 는 PARP1 과의 결합에 있어서 중요한 부분이라는 것을 면역침강법을 사용하여서 확인할 수 있었다(Fig. 6G). 따라서 Rsf-1 은 PHD 도메인을 통해 PARP1 과 결합하고 PARP1 의 활성에 의해 Rsf-1 이 DNA 손상 부위로 모인다는 것을 알 수 있었다. Rsf-1 이 DNA 손상 이후 30 분까지도 DNA 손상 부위로 모이는 것을 확인하였고(Fig. 2), 이는 Rsf-1 이 DNA 손상 복구에 있어서도 역할을 할 것이라는 것을 알 수 있었다. DNA 가 손상 되면 이를 복구 시키기 위해서 세포 주기가 멈추게 되는데 그것을 조절하는 단백질의 활성을 확인 해봄으로써 Rsf-1 이 DNA 손상 복구에도 영향을 끼친다는 것을 확인하였다. 또한 세포 생존율을 확인해봄으로써 Rsf-1 이 DNA 이중 나선 절단에 있어서 손상 복구 신호와 그
단백질들을 조절 한다는 것을 알 수 있었다(Fig. 7). Rsf-1 이 DNA 손상 신호 전달 체계에서 매우 빠른 시기에 관여를 하며 그 다음 단계로 신호를 전달하는 것을 용이하게 해준다는 것을 확인하였고 손상 복구 신호에도 영향이 있다는 것을 확인하였다. DNA 손상 복구 신호에도 여러 가지 경로를 통해 일어나고 복구 되기 때문에 Rsf-1 이 어떤 곳에 연관 되어 있는지에 대한 연구가 계속 진행되면 Rsf-1 의 역할과 기능에 대해서 더 많은 이해를 할 수 있을 것이다.
Ⅴ
. 결 론
본 연구에서는 DNA 손상 시 크로마틴 리모델링 인자인 Rsf-1 이 손상 신호 전달 체계에서 손상 부위로 빨리 모여서 작용하고 SNF2h 와 H2AX 가 DNA 손상 부위로 이동하는데 중요하게 요구된다는 결과를 얻었다. 또한 PARP1 활성에 의존적으로 DNA 손상 부위로 모이며, PHD 도메인을 통해 PARP1 과의 결합을 통하여 발생한다는 것을 알 수 있었다. 또한 Rsf-1 이 없을 경우 DNA 손상이 발생하면 세포 주기를 멈추는 단백질들의 활성이 낮아져 있고 세포 생존율이 낮은 것을 통해 DNA 손상 시 복구되는 과정에 있어서도 필요하다는 것을 확인할 수 있었다. 따라서 본 논문은 Rsf-1 가 DNA 손상 시 크로마틴 리모델링 인자로써의 역할과 기전에 대해서 밝힌 논문으로 앞으로 DNA 손상 신호 전달 체계와 복구과정에서 Rsf-1 의 중요성을 제시한 논문이다.참
고 문 헌
1. Burma S, Chen BP, Murphy M, Kurimasa A and Chen DJ. : ATM
phosphorylates histone H2AX in response to DNA double-strand breaks.
J Biol Chem. 276(45):42462-7, 2001
2. Choi JH, Sheu JJ, Guan B, Jinawath N, Markowski P, Wang TL and
Shih IeM. : Functional analysis of 11q13.5 amplicon identifies
Rsf-1(HBXAP) as a gene involved in paclitaxel resistance in ovarian cancer.
Cancer Res. 69(4):1407-15, 2009
3. Davidson B, Trope' CG, Wang TL and Shih IeM. : Expression of the
chromatin remodeling factor Rsf-1 is upregulated ovarian carcinoma
effusions and predicts poor survival. Gynecol Oncol. 103(3):814-9, 2006
4. Han-Sae Lee, Ji-Hye Park, So-Jung Kim, Su-Jung Kwon and
Jongbum Kwon : A cooperative activation loop among SWI/SNF,
γ-H2AX and H3 acetylation for DNA double-strand break repair. EMBO J.
29(8):1434-45, 2010
5. Hannes Lans, Jurgen A Marteijn and Wim Vermeulen :
ATP-dependent chromatin remodeling in the DNA-damage response.
Epigenetics & Chromatin, 5:4, 2012
6. Helfrid Hochegger, Donniphat Dejsuphong, Toru Fukushima, Ciaran
Morrison, Eiichiro Sonoda, Valérie Schreiber, Guang Yu Zhao,
Alihossein Saberi, Mitsuko Masutani, Noritaka Adachi, Hideki Koyama,
Gilbert de Murcia and Shunichi Takeda : Parp-1 protects homologous
recombination from interference by Ku and Ligase IV in vertebrate cells.
EMBO J. 25(6): 1305–1314, 2006
7. Hiroyuki Niida and Makoto Nakanishi : DNA damage checkpoints in
mammals. Mutagenesis. 21(1): 3–9, 2006
8. Huang JY, Shen BJ, Tsai WH and Lee SC. : Functional interaction
between nuclear matrix-associated HBXAP and NF-kappaB. Exp Cell
Res. 298(1):133-43, 2004
9. Jean-François Haince, Darin McDonald, Amélie Rodrigue, Ugo Déry,
Jean-Yves Masson, Michael J. Hendzel and Guy G. Poirier :
PARP1-dependent Kinetics of Recruitment of MRE11 and NBS1 Proteins to
Multiple DNA Damage Sites. J Biol Chem. 283(2):1197-1208, 2008
10. Lan L, Ui A, Nakajima S, Hatakeyama K, Hoshi M, Watanabe R,
Janicki SM, Ogiwara H, Kohno T, Kanno S andYasui A. : The ACF1
complex is required for DNA double-strand break repair in human cells.
Mol Cell. 40(6):976-87, 2010
11. Loyola A, Huang JY, LeRoy G, Hu S, Wang YH, Donnelly RJ, Lane
WS, Lee SC and Reinberg D. : Functional analysis of the subunits of the
chromatin assembly factor RSF. Mol Cell Biol. , 23(19):6759-68, 2003
12. Manuel Stucki, Julie A. Clapperton, Duaa Mohammad, Michael B.
Yaffe, Stephen J. Smerdon and Stephen P. Jackson : MDC1 Directly
Binds Phosphorylated Histone H2AX to Regulate Cellular Responses to
DNA Double-Strand Breaks. Cell, 123( 7):1213-1226, 2005
13. Mao TL, Hsu CY, Yen MJ, Gilks B, Sheu JJ, Gabrielson E, Vang R,
Cope L, Kurman RJ, Wang TL and Shih IeM. : Expression of Rsf-1, a
chromatin-remodeling gene, in ovarian and breast carcinoma. Hum
Pathol. 37(9):1169-75, 2006
14. Minli Wang, Weizhong Wu, Wenqi Wu, Bustanur Rosidi, Lihua
Zhang, Huichen Wang,1 and George Iliakis : PARP-1 and Ku compete
for repair of DNA double strand breaks by distinct NHEJ pathways.
15. Perpelescu M, Nozaki N, Obuse C, Yang H and Yoda K. : Active
establishment of centromeric CENP-A chromatin by RSF complex. J
Cell Biol. 185(3):397-407, 2009
16. Rogakou EP, Pilch DR, Orr AH, Ivanova VS and Bonner WM. :
DNA double-stranded breaks induce histone H2AX phosphorylation on
serine 139. J Biol Chem. 273(10):5858-68, 1998
17. Schultz N, Lopez E, Saleh-Gohari N and Helleday T. :
Poly(ADP-ribose) polymerase (PARP-1) has a controlling role in homologous
recombination. Nucleic Acids Res. 31(17):4959-64, 2003
18. Shamay M, Barak O and Shaul Y. : HBXAP, a novel PHD-finger
protein, possesses transcription repression activity. Genomics.
9(4):523-9, 2002
19. Shamay M, Barak O, Doitsh G, Ben-Dor I and Shaul Y. : Hepatitis B
virus pX interacts with HBXAP, a PHD finger protein to coactivate
transcription. J Biol Chem. 277(12):9982-8, 2002
20. Shanbhag NM, Rafalska-Metcalf IU, Balane-Bolivar C, Janicki SM
and Greenberg RA. : ATM-dependent chromatin changes silence
transcription in cis to DNA double-strand breaks. Cell. 141(6):970-81,
2010
21. Sheu JJ, Guan B, Choi JH, Lin A, Lee CH, Hsiao YT, Wang TL,
Tsai FJ and Shih IeM. : Rsf-1, a chromatin remodeling protein, induces
DNA damage and promotes genomic instability. J Biol Chem.
285(49):38260-9, 2010
22. Shih IeM, Sheu JJ, Santillan A, Nakayama K, Yen MJ, Bristow RE,
Vang R, Parmigiani G, Kurman RJ, Trope CG, Davidson B and Wang
TL. : Amplification of a chromatin remodeling gene, Rsf-1/HBXAP, in
ovarian carcinoma. Proc Natl Acad Sci U S A. 102(39):14004-9, 2005
23. Smeenk G and van Attikum H. : The chromatin response to DNA
breaks: leaving a mark on genome integrity.
Annu Rev Biochem.
82:55-80, 2013
24. Smeenk G, Wiegant WW, Marteijn JA, Luijsterburg MS, Sroczynski
N, Costelloe T, Romeijn RJ, Pastink A, Mailand N, Vermeulen W and
van Attikum H. : Poly(ADP-ribisyl)ation links the chromatin remodeler
SMARCA5/SNF2H to RNF168-dependent DNA damage signaling. J
25. Smeenk G, Wiegant WW, Vrolijk H, Solari AP, Pastink A, van
Attikum H. : The NuRD chromatin-remodeling complex regulates
signaling and repair of DNA damage. J Cell Biol 190(5):741-9, 2010
Sugimura K, Takebayashi S, Taguchi H, Takeda S and Okumura K. :
PARP-1 ensures regulation of replication fork progression by
homologous recombination on damaged DNA. J Cell Biol.
183(7):1203-12, 2008
26. Xiao A, Li H, Shechter D, Ahn SH, Fabrizio LA,
Erdjument-Bromage H, Ishibe-Murakami S, Wang B, Tempst P, Hofmann K, Patel
DJ, Elledge SJ, Allis CD. : WSTF regulates the H2A.X DNA damage
response via a novel tyrosine kinase activity. Nature. 457(7225):57-62,
2009
- ABSTRACT -
A functional role of chromatin remodeling factor, Rsf-1 in
the DNA damage signaling pathway
Sujin Jo
Department of Biomedical sciences The Graduate School, Ajou university (Supervised by Professor Hyeseong Cho)
As a member of imitation switch (ISWI) family in ATP-dependent chromatin
remodeling factors, RSF complex consists of SNF2h ATPase and Rsf-1. Although it
has been reported that SNF2h ATPase is recruited to DNA damage sites (DSBs) in
poly(ADP-ribosyl) polymerase 1 (PARP1)-dependent manner in DNA damage
response (DDR), the function of Rsf-1 is still elusive. Here we show that Rsf-1 is
recruited to DSBs confirmed by various cellular analyses (immunofluorescent
microscope, micro-irradiation and stably integrated reporter system at a single DNA
double-strand break). Rsf-1 rapidly accumulated at the DSB sites. Signal of γ-H2AX
is gradually reduced at 10 minutes after micro-irradiation whereas signals of Rsf-1
and SNF2h are still retained over 30 minutes after DNA damage. Moreover,
depletion of Rsf-1 attenuates the recruitment of SNF2h and γ-H2AX. In addition,
Rsf-1 accumulation at DNA damage sites requires PARP1 activity through
interacting with PHD domain. Finally, we demonstrate that depletion of Rsf-1
diminishes DNA damage checkpoint signals and repair. Thus, these results reveal a
new function of chromatin remodeler Rsf-1 in coordinating DNA signaling and repair.
key words : DNA damage, chromatin remodeling factor, Rsf-1, PARP1, DNA damage checkpoint