저작자표시-비영리-동일조건변경허락 2.0 대한민국 이용자는 아래의 조건을 따르는 경우에 한하여 자유롭게 l 이 저작물을 복제, 배포, 전송, 전시, 공연 및 방송할 수 있습니다. l 이차적 저작물을 작성할 수 있습니다. 다음과 같은 조건을 따라야 합니다: l 귀하는, 이 저작물의 재이용이나 배포의 경우, 이 저작물에 적용된 이용허락조건 을 명확하게 나타내어야 합니다. l 저작권자로부터 별도의 허가를 받으면 이러한 조건들은 적용되지 않습니다. 저작권법에 따른 이용자의 권리는 위의 내용에 의하여 영향을 받지 않습니다. 이것은 이용허락규약(Legal Code)을 이해하기 쉽게 요약한 것입니다. Disclaimer 저작자표시. 귀하는 원저작자를 표시하여야 합니다. 비영리. 귀하는 이 저작물을 영리 목적으로 이용할 수 없습니다. 동일조건변경허락. 귀하가 이 저작물을 개작, 변형 또는 가공했을 경우 에는, 이 저작물과 동일한 이용허락조건하에서만 배포할 수 있습니다.
이학 석사학위 논문
간암의 전사체 선택 접합 프로파일링
아 주 대 학 교
대 학 원
의생명과학과/
분자의학전공
간암의 전사체 선택 접합 프로파일링
지도교수
우 현 구
이 논문을 이학 석사학위 논문으로 제출함.
2014년
6월
아 주 대 학 교
대 학 원
의생명과학과/
분자의학전공
서 미 경
서미경의 이학 석사학위 논문을 인준함.
심사위원장
우
현
구
인
심 사 위 원
백
은
주
인
심 사 위 원
강
호
철
인
아 주 대 학 교
대 학 원
2014년 06월 20일
- 국문요약 -
간암의
전사체 선택 접합 프로파일링
간암의 발달은 다단계의 복잡한 과정을 거쳐 이루어지며, 다양한 분자생물학적인 기전이 관여하고 있음이 알려져 있다. 최근 차세대 시컨싱(Next generation sequencing) 기술 발전은 유전체 전장에 걸친 염기서열 확인과 전사체의 발현 측정을 가능하게 하였다. 특히, 전사체 서열분석(RNA sequencing)은 기존의 마이크로어레이 분석기법과 달리 전사체의 선택 접합에 의한 이소(isoform) 발현을 측정할 수 있게 하였다. 암의 발달 과정에도 선택 접합에 의한 다양한 유전자의 이소 발현이 관여하고 있음이 알려져 있다. 이에, 본 연구에서는 간암의 발달에 미치는 선택 접합의 영향을 알아보기 위하여 61 명 간암 환자의 전사체 서열분석을 통해 선택 접합 프로파일링을 시행하였다. 전사체의 이소 발현 프로파일에 기반하여 환자들을 클러스터링(clustering) 분석을 시행한 결과, 2 개의 간암 환자의 아형이 있음을 관찰하였다. 선택 접합 프로파일링이 간암 환자를 임상적인 예후가 다른 두 개의 그룹으로 나눌 수 있음을 확인하였다. 본 연구에서 발굴한 간암 아형의 유전자 발현을 분석한 결과, 세포사멸의 조절(Regulation of apoptosis), 세포 이동의 조절(regulation of cell migration), 세포 부착의 조절(regulation of cell adhesion), 세포 주기의 조절(regulation of cell cycle), 국소부착(focal adhesion), 액틴 세포골격 조절(regulation of actin cytoskeleton), mTOR 신호전달경로, 종양 관련 신호전달과정등이 관여하고 있음을 밝혔다. 특히, 간암 아형에 따라 WNT5A 가 선택 접합에 의해 일어나 이소 발현을 하고 있음을 관찰하였고, WNT5A 의 선택 접합이 간암의 재발과 종양 발달에 영향을 미칠 것으로 생각된다. 이상의 연구 결과는, 전체 전사체 선택 접합 프로파일링에 기반한 간암 환자의 분류를 통해서 이질적인 간암 환자들을 임상 및 병리학적 특징이 다른 아형으로 분류할 수 있음을 제안하고, 전사체 선택 접합이 간암의 형성 및 발달의 복합적인 조절 변화에 대한 단서를 제공할 수 있다. 핵심어 : 전사체 서열분석, 간세포암종, HCC, 선택 접합
차
례
국문요약 ··· ⅰ 차례 ··· ⅲ 그림차례 ··· ⅴ 표차례 ··· ⅵ Ⅰ. 서론 ··· 1 Ⅱ. 연구대상 및 방법 ··· 10 A. 시료 ··· 10 B. RNA-sequencing ··· 10 C. RNA 서열분석 리드의 맵핑(mapping) ··· 11 D. 전사체 조립(assembly) ··· 12 E. 분류(classification) ··· 13F. 차등 선택 접합(differentially alternative splicing) 분석 ··· 14
G. Functional enrichment 분석 ··· 14
H. 통계학적 검증 ··· 14
Ⅲ. 결과 ··· 17
A. 간암 RNA 서열분석 데이터 분석 ··· 17
B. 분류(classification) ··· 17
C. 차등 선택 접합(differentially alternative splicing) 분석 ··· 32
Ⅴ. 결론 ··· 45 참고문헌 ··· 46 ABSTRACT ··· 59
그림
차례
Fig. 1. The different types of alternative splicing ··· 7 Fig. 2. Schematic for classification based on splicing profile ··· 16 Fig. 3. Base sequence quality ··· 22 Fig. 4. Unsupervised clustering based on 772 genes with splicing switching and
high variability among samples and classification ··· 26 Fig. 5. The distribution of major and minor transcripts ··· 27 Fig. 6. Kaplan–Meier survival curves for recurrence-free survival between two
subtypes ··· 30 Fig. 7. The heatmap of 1,043 genes with differentially alternative splicing between
two subtypes ··· 34 Fig. 8. Network of apoptosis in splicing switching in subtype 1 ··· 35 Fig. 9. Network of Insulin/mTOR signaling pathway in splicing switching in
subtype 1 ··· 36 Fig. 10. Network of cell cycle in splicing switching in subtype 2 ··· 37 Fig. 11. The splicing pattern of WNT5A ··· 38
표
차례
Table 1. Alternative spliced transcripts related to HCC ··· 8 Table 2. Classification of cancer based on alternative splicing profile ··· 9 Table 3. Statistics of 61 HCC data ··· 23 Table 4. Relationship between subtypes and clinicopathologic variables of patients ··· 28 Table 5. Significant biological processes and KEGG pathway of 772 genes for
I. 서 론
간암은 전세계적으로 발병률이 5위에 해당하는 암으로, 사망률은 3위이며, 한국인의 간암 사망률은 2위에 해당한다(El-Serag and Rudolph, 2007)(통계청, 2013). 간암은 우리나라 50대 전후 남자의 주요 사망 원인으로 남자가 여자보다 약 3배 더 사망률이 높다(통계청, 2013). 간암 중 약 75%가 간세포암종(Hepatocellular carcinoma; HCC)에 해당하며 예후가 좋지 않고, 치료약물도 제한적이다. 또한 간암은 형성과정이 다양하고, 복합적인 다단계에 걸쳐 발달하기 때문에 환자에 따라 분자생물학적 기전이 다를 수 있으며, 치료약물의 반응 차이가 다양하다. 따라서 간암 환자의 분자생물학적인 기전을 명확하게 파악하는 것이 환자의 효과적인 치료에 중요하기 때문에, 환자의 이질성을 이해하는 것이 시급한 실정이다.
최근 RNA 서열분석(RNA-sequencing) 발전으로, 암 연구에 RNA 서열분석이 응용되고 있으며, 암과 연관된 종양 유전자 및 종양 억제 유전자의 원인 변이(driver variant)와 발현(expression) 이상, 융합(fusion) 유전자에 대한 연구가 많이 진행되고 있다(David and Manley, 2010). 그러나 전체 전사체에 대한 선택 접합(alternative splicing)의 프로파일링에 대한 연구는 드물다(David and Manley, 2010). 하지만, 전사체 선택 접합이 암의 세포주기(cell cycle), 증식(proliferation), 분화(differentiation), 신호전달(signal transduction) pathways, 혈관 신생(angiogenesis), 침습(invasion), 전이(metastasis)에서 중요한 영향을 미친다는 연구 사례가 증가하고 있다(David and Manley, 2010). 전사체 선택 접합은 전사체와 단백질의
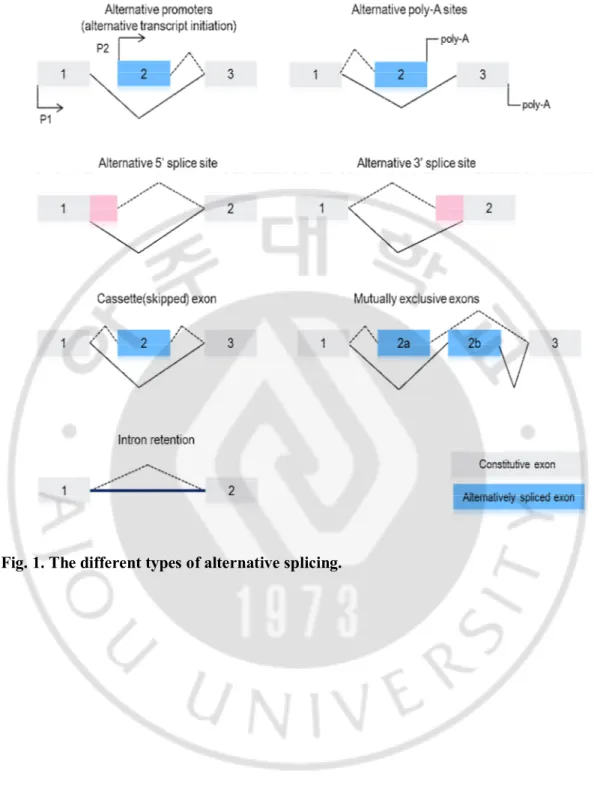
다양성을 증가시키며, 유전자 발현을 조절하고, 단백질의 기능에 영향을 미치는 생물학적 과정이다(Graveley, 2001; Keren et al., 2010). DNA에 저장된 유전 정보는 mRNA로 전사(transcription)된 후 번역(translation) 과정을 통해 단백질로 합성된다. 인간 유전체 프로젝트(human genome project) 완료 후 인간 유전자는 약 2만 5천 개가 존재함이 밝혀졌다(Goodstadt and Ponting, 2006). 이 유전자로 약 10만 개의 단백질이 생산될 수 있는데, 이렇게 전사체와 단백질의 다양성을 설명해 줄 수 있는 기전 중 하나가 선택 접합이다(Valdivia, 2007; Tang and Riva, 2013). 한 개의 유전자의 primary mRNA (pre-mRNA)에서 선택 접합이 일어나 여러 개의 전사체와 단백질이 생산될 수 있고 여러 개의 엑손으로 이루어진 인간 유전자의 90% 이상에서 전사체 선택 접합이 일어난다고 알려져 있으며(Pan et al., 2008; Wang et al., 2008), 선택 접합의 유형은 약 7가지가 알려져 있다(Li et al., 2007)(Fig. 1):
Alternative promoters, Alternative polyadenylation, Alternative 5' SS, Alternative 3' SS, Cassette (skipped) exon, Mutually exclusive exons, Intron retention. 정상 세포와 암 세포의 선택 접합 유형에 대해 분석한 연구에 따르면, 암 세포에서 하나의 엑손이 포함되거나 배제되는 유형인 cassette (skipped) exon은 드문 반면에, 오히려 alternative 5’ 과 3’ 접합자리(splice site) 선택과 intron retention 유형이 더 빈번하게 관찰된다고 한다(Kim et al., 2008). 선택 접합은 mRNA의 안정성(stability), 단백질의 구조 및 기능과 세포 내 분포(localization)의 변화를 가져와서 질병, 암, 약물 내성(resistance)에 영향을 미칠 수 있다(Gabut et al., 2011; Buljan et al., 2012; Ellis et al., 2012; Tang and Riva, 2013). 선택 접합이 일어난 유전자의 전사체가 암에서 생물학적 기능이 다른, 심지어 상반되는 역할을 한다는 사례가 보고되어 있으며(Venables et al., 2006), 가장 잘 연구된 사례는 미토콘드리아 외막의
투과성(mitochondria outer membrane permeabilization)에 영향을 미쳐 세포사멸(apoptosis)에 관여하는 Bcl-x (BCL2L1)이다. Bcl-x는 3개의 엑손으로 구성된 유전자로 엑손 2에 두 가지 alternative 5’ 접합자리가 존재한다. 이 5’ 접합자리를 선택적으로 사용함으로써 Bcl-x 유전자에서 선택 접합이 일어나 두 개의 길이가 다른 전사체를 만든다. 길이가 긴 전사체 Bcl-xL은 233개의 아미노산으로 이루어진 단백질을 만드는 전사체로 길이가 짧은 전사체 Bcl-xS에는 없는 Bcl-2 family motifs BH1과 BH2 도메인을 포함하고 있으며, anti-apoptotic 기능을 하는 반면에, Bcl-xS는 170개의 아미노산 단백질을 만드는 전사체로 pro-apoptotic 기능을 한다(Boise et al., 1993). 간암을 포함한 많은 암에서 anti-apoptotic Bcl-xL 전사체가 과발현(overexpression)되어 있으며, 화학치료에 대한 내성(resistance)과 상관성이 있음이 보고 되어 있다(Mercatante et al., 2001; Takehara et al., 2001; Mercatante et al., 2002). 이 외에 세포사멸에 관여하는 CASP9 (caspase9)도 두 개의 전사체가 세포사멸에 상반된 기능을 한다고 알려져 있다(Shultz et al., 2010). 또한, 암에서 선택 접합이 보고된 유전자는 세포 증식을 조절하는 FGFR2, p53, MDM2, FHIT, BRCA1과 세포 부착(cellular adhesion)과 침습에 관여하는 CD44, Ron, 혈관 신생에 관여하는 VEGF, 전이와 관련된 osteopontin (OPN), 세포사멸에 영향을 미치는 Fas, Bcl-x, caspase2, 다제성약물 내성(multidrug resistance)에 관여하는 MRP-1이 있다(He et al., 2004; Revil et al., 2006; Pajares et al., 2007; Skotheim and Nees, 2007; Klinck et al., 2008; Roundhill and Burchill, 2012).
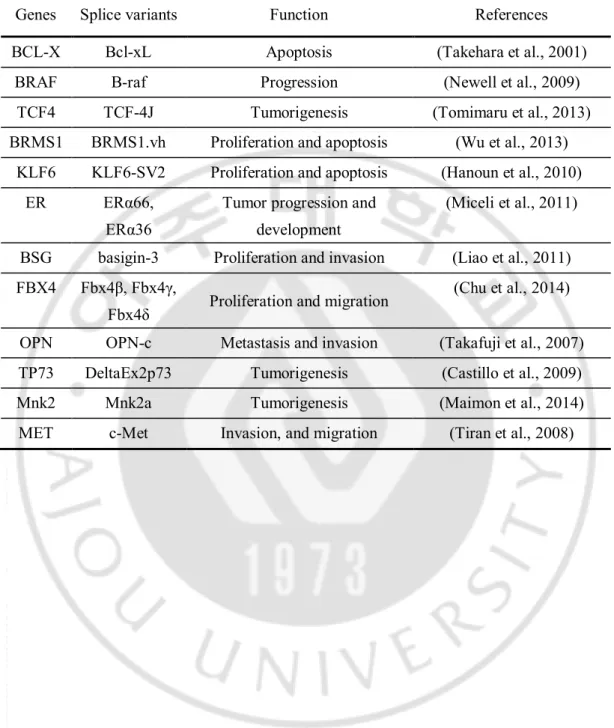
간암에서 알려진 전사체 선택 접합의 유전자로는 Bcl-X, BRAF, TCF4, BRMS1, KLF6, BSG, FBX4, OPN, TP75, MNK2, MET 등이 보고되어 있다(Table 1). 이 중 OPN (osteopontin)은 이 유전자로부터 전사된 mRNA의 선택 접합된(alternative
spliced) 전사체의 종류에 따라 암의 전이에 미치는 영향이 다르다. 전이 유전자 OPN은 phosphoprotein 1 (SPP1) 단백질로 번역이 되어 세포 이동(migration)에
관여하며, 인테그린 수용체(integrin receptor)와 결합하여 세포를
세포예정사(programmed cell death)로부터 보호한다. OPN은 간암을 포함한 결장암, 유방암, 식도암 등 많은 암에서 과발현되어 있으며(Chambers et al., 1996; Thalmann et al., 1999; Tuck and Chambers, 2001; Agrawal et al., 2002; Ye et al., 2003; Schorge et al., 2004), 전이 phenotype과 상관관계가 있음이 보고되어 있다(Ito et al., 2006). OPN 유전자에서 선택 접합이 일어나면 osteopontin-a, osteopontin-b, osteopontin-c의 3개 전사체가 만들어진다. OPN-a는 모든 엑손이 포함되어 있지만, OPN-b는 엑손 5가 없고, OPN-c는 엑손 4가 존재하지 않는다(Hijiya et al., 1994). OPN-a는 암세포와 정상세포에서 모두 발현하며, OPN-b는 발현량이 적어 검출이 어렵다(Mirza et al., 2008). OPN-c는 간 주변의 정상 조직에서는 발현이 되지 않는 반면에, 간 종양 조직에서는 높게 발현되고, 특히 비침습성(non-invasive) 종양보다 침습성(invasive) 종양에서 높게 발현된다(Mirza et al., 2008). 이것을 종합해 보면, OPN-c가 간암의 전이에 중요한 역할을 하며, 잠재적인 진단 마커로 활용될 수 있음을 보여준다고 할 수 있다(Ito et al., 2006; Mirza et al., 2008; Zhang et al., 2013). 암에서 이러한 유전자의 비정상 또는 선택 접합은 질병의 원인 또는 질병에 영향을 미치는 요인이 될 수 있으며(Davuluri et al., 2008; Ragan et al., 2009), 선택 접합으로 만들어진 전사체는 조직 특이적으로 발현하기 때문에 암 치료 표적(target)으로 효과적일 수 있다(Garcia-Blanco et al., 2004).
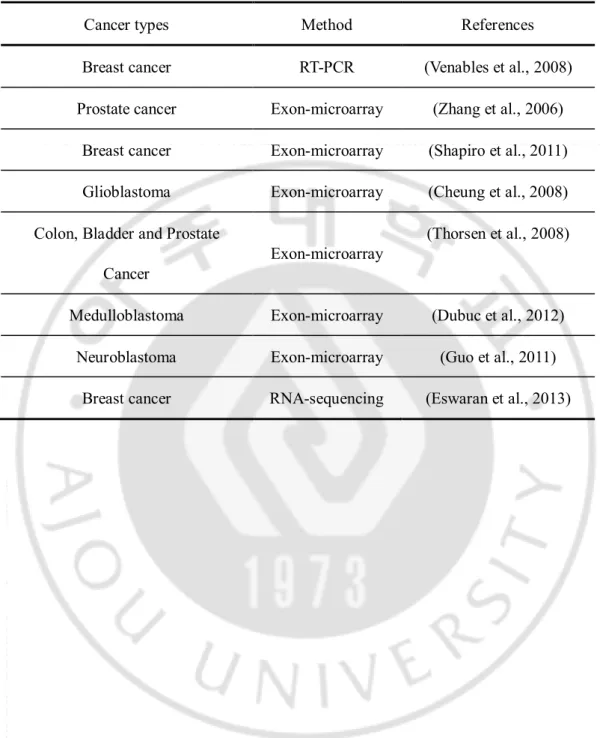
암에 대한 전사체 선택 접합 연구는 기존 방법의 RT-PCR (reverse transcription-PCR)과 엑손과 엑손 사이의 접합(junction) 지점에 탐침자(probe)를
제작하여 선택 접합을 확인하는 엑손-마이크로어레이(exon-microarray)로 가장 많이 연구가 되어 있다(Table 2). High-throughput RT-PCR을 사용하여 21개의 정상 및 26개의 유방암 종양 조직을 대상으로 600개의 암 연관 유전자의 선택 접합에 대해 프로파일링을 한 연구가 있다(Venables et al., 2008). 이 연구에서는 정상과 종양을 분류할 수 있는 12개의 선택 접합된 전사체를 밝혀냈다. 이 12개의 선택 접합된 전사체로 정상 세포와 종양 세포를 분류할 수 있으며, 조직 병리학적 등급(histopathologic grade)에 따라 종양을 세부 그룹으로 더 분류할 수 있음을 보여주었다(Venables et al., 2008). 유방암 이외에도 전립선암, 유방암, 뇌암, 결장암 등에서 엑손-마이크로어레이를 사용하여 암환자를 분류한 연구가 있다(Zhang et al., 2006; Cheung et al., 2008; Thorsen et al., 2008; Guo et al., 2011; Shapiro et al., 2011; Dubuc et al., 2012).
현재 차세대 서열분석(Next generation sequencing) 기술의 발달로, RNA 서열분석(RNA-sequencing)을 이용하여 더 총제적이고 정확하게 전체 전사체의 분석이 용이해졌다(Wang et al., 2009). RNA 서열분석을 사용하면, RT-PCR과 다르게 엑손과 엑손 사이의 접합 지점에 탐침자를 제작하지 않아도 전사체를 확인할 수 있으므로, 탐침자가 제작된 제한된 전사체의 확인이 아닌, 모든 전사체 수준에서의 선택 접합 확인이 가능한 장점이 있다. 최근에 RNA 서열분석을 사용하여 전사체 및 선택 접합 프로파일을 통해 유방암 환자를 세부 subtype으로 분류할 수 있음을 보여주는 연구가 보고 되었다(Eswaran et al., 2012). RNA 서열분석을 이용하여 암의 전사체 선택 접합 연구가 증가하고 있지만, 선택 접합된 전사체의 발현량을 기준으로 하는 것이 아닌, 전사체의 선택 접합 프로파일링을 기반으로 암 환자를 분류(classification)한 연구는 드물며, 간암
환자를 대상으로 한 연구는 거의 없는 실정이다. 본 연구에서는, 환자들간에도 이질성이 크다고 알려진 간암 환자의 RNA 서열 분석을 통해, 간암의 전체 전사체에 대한 선택 접합 프로파일을 분석하였다. 그리고 선택 접합 프로파일링에 기반하여 61명의 HCC 환자를 예후가 유의하게 다른 환자 아형으로 분류함으로써, 선택 접합이 간암의 발암 및 발달 기전에 서로 다른 역할을 할 수 있음을 제안하고자 한다.
Table 1. Alternative spliced transcripts related to HCC.
Genes Splice variants Function References
BCL-X Bcl-xL Apoptosis (Takehara et al., 2001)
BRAF B-raf Progression (Newell et al., 2009)
TCF4 TCF-4J Tumorigenesis (Tomimaru et al., 2013)
BRMS1 BRMS1.vh Proliferation and apoptosis (Wu et al., 2013) KLF6 KLF6-SV2 Proliferation and apoptosis (Hanoun et al., 2010)
ER ERα66,
ERα36
Tumor progression and development
(Miceli et al., 2011) BSG basigin-3 Proliferation and invasion (Liao et al., 2011) FBX4 Fbx4β, Fbx4γ,
Fbx4δ Proliferation and migration
(Chu et al., 2014) OPN OPN-c Metastasis and invasion (Takafuji et al., 2007) TP73 DeltaEx2p73 Tumorigenesis (Castillo et al., 2009)
Mnk2 Mnk2a Tumorigenesis (Maimon et al., 2014)
Table 2. Classification of cancer based on alternative splicing profile.
Cancer types Method References
Breast cancer RT-PCR (Venables et al., 2008)
Prostate cancer Exon-microarray (Zhang et al., 2006) Breast cancer Exon-microarray (Shapiro et al., 2011) Glioblastoma Exon-microarray (Cheung et al., 2008) Colon, Bladder and Prostate
Cancer
Exon-microarray
(Thorsen et al., 2008)
Medulloblastoma Exon-microarray (Dubuc et al., 2012) Neuroblastoma Exon-microarray (Guo et al., 2011)
Ⅱ
. 연구대상 및 방법
A. 시료
61 명의 HCC 환자의 종양 조직과 정상 간 조직은 각각 아주대 병원에서 제공하였다. 이 연구의 프로토콜은 임상시험심사위원회(Institutional Review Board)에 의해 승인되었다. 61 건의 HCC 와 7 건의 정상 간 조직의 total RNA 는 mirVana™ miRNA Isolation Kit (Life technology)를 사용하여 제조사의 지시에 따라 추출하였다. RNA 의 양은 NanoDrop spectrophotometer (NanoDrop Technologies, Wilmington, DE)을 사용하여 정량화하였고, RNA integrity 분석은 Agilent 2100 Bioanalyzer (Agilent Technologies, Santa Clara, CA)를 사용하였다. RNA integrity number (RIN)가 7 이상인 샘플만 RNA 서열분석을 하였다.
B. RNA-sequencing
Oligo 폴리 T 가 부착되어 있는 자성(magnetic) 비드를 사용하여, total RNA 에서 폴리 A 가 포함되어 있는 mRNA 를 분리하였다. 분리된 mRNA 는 작은 단편(fragment)으로 조각을 냈다. 그 후 조각난 mRNA 는 랜덤 헥사머 프라이머(Invitrogen, Carlsbad, CA, USA)를 사용하여 역전사 과정을 거쳐 첫 번째 cDNA (complementary DNA) 가닥(strand)으로 전사시킨 다음에 이어서 두 번째 cDNA 가닥으로 합성하였다. 그 후 End-repair 과정을 거친 다음에 cDNA 단편의 3’ 말단에 single A 염기를 추가하였다. cDNA 에 adapter 를 붙인 후, 2% 아가로즈 겔을 사용하여 이를 분리하였고, PCR 을 사용하여 증폭하여 최종 cDNA library 를
제작하였다. cDNA library 는 Illumina TruSeq RNA Sample Prep Kit v2 (Illumina, San Diego, CA)를 사용하여 제작하였다. 증폭된 샘플의 RNA 서열분석은 paired-end read 방식으로 Illumina HiSeq 2000 을 사용하여 시행하였다.
C. RNA 서열분석 리드의 맵핑(mapping)
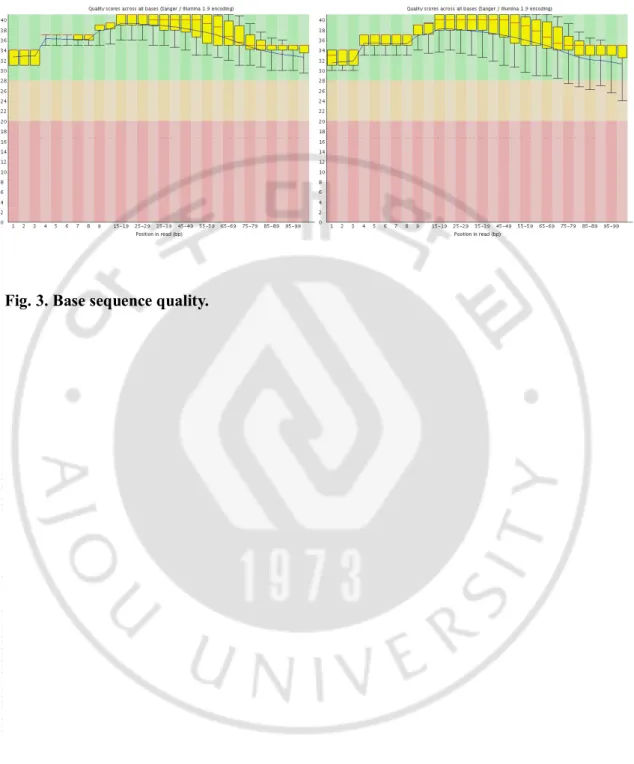
일루미나(Illumina) RNA 서열분석 데이터로 나온 100 bp 길이의 paired-end 리드(2 x 100 reads)는 고품질의 리드만 맵핑에 사용하기 위해, FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc)를 사용하여 염기서열 품질을 확인하였다. FASTX Toolkit 의 fastq_masker 옵션을 사용하여 염기서열 품질(base quality)이 20 이하인 염기는 N 으로 처리하였다. RNA 서열분석은 짧은 단편으로 된 cDNA 의 염기서열을 해독한 후, 이 짧은 서열 단편인 리드(read)를 참조 유전체(reference genome)에 맵핑(mapping)하는 방법이다. 엑손의 경계인 접합 연결지점(splice junction)을 인지하여 리드를 엑손에 맵핑하는 Tophat2 (v2.0.8)에는 short read aligner 인 Bowtie2 가 내장되어 있다. Tophat2 를 사용하여 일루미나(Illumina) RNA 서열분석으로 나온 mRNA 의 리드를 RefSeq 유전자 모델을 사용하여 인간 참조 유전체(hg19)에 맵핑하였다(parameter: -G). 레퍼런스로 사용한 hg19 와 RefSeq GTF 파일은 일루미나에서 제공하는 iGenome (http://support.illumina.com/sequencing/sequencing_software/igenome.ilmn)에서 파일을 내려 받아 사용하였다. 다시 말하면, cDNA 단편에 해당하는 짧은 길이의 리드는 하나의 전사체의 정보를 모두 포함하지 못하기 때문에, 이 리드를 다시 유전체에 맵핑하고, 전사체로 재조립하는 단계를 거쳐 분석하게 된다. 리드를
유전체에 맵핑할 때, Tophat2 에 포함되어 있는 Bowtie2 로 인간 참조 유전체(hg19)에 짧은 리드를 맵핑하게 된다. 이때 리드가 참조 유전체에 맵핑이 되면, 이 리드는 잠재적인 엑손이라고 할 수 있다. 하지만, 초기에 참조 유전체에 맵핑이 되지 않은 리드인 initially unmappable (IUM) 리드가 생기게 된다. 접합 자리(splice site)를 찾기 위해서, 초기에 확인된 잠재적인 엑손으로 2 개의 엑손 조합을 만든 후, 이 2 개의 엑손의 사이를 seed 라고 명칭하고, 이 seed 에 IUM 리드를 맵핑해본다. IUM 리드가 seed 에 맵핑이 되면, 옆의 서열로 확장하는 방식으로 서열을 검색한다(seed and extend 방식). 그 이후 Tophat2 를 사용하여 유전체에 최종 맵핑된 리드는 Picard 프로그램(http://picard.sourceforge.net)을 사용하여 라이브러리 제작으로 나올 수 있는 PCR duplicate 를 제거하였다.
D. 전사체 조립(assembly)
매핑된 리드는 Cufflinks (Cufflinks v 0.8.3) 프로그램을 사용하여 발현된 유전자와 전사체(transcript)의 종류를 확인하고, 발현량은 각각 샘플의 맵핑된 리드의 총 수와 엑손의 길이를 고려하여 정규화된 수치인 FPKM (Fragments Per Kilobase of transcript per Million mapped reads) 단위로 정량화하였다(Trapnell et al., 2010). 서열분석을 하기 위해 조직의 mRNA 로 library 를 구축하는 과정에서 샘플마다 맵핑된 리드의 총 수가 다양해질 수 있고, 길이가 짧은 전사체보다 길이가 긴 전사체에 리드가 더 맵핑될 수 있는 bias 가 있기 때문에, 맵핑된 리드의 총 수와 전사체의 길이로 발현값을 보정하는 것이 필요하다. 이를 보정하기 위해 FPKM 단위로 발현량을 정량화하였다. 그 후, 계산을 용이하게하기 위해 전사체의 FPKM 에 1 을 더한 값을 log2 로 전환하여 분석에 사용하였다.
E. 분류(classification)
종양 샘플에서의 선택 접합 프로파일링을 하기 위해, 정상 간 조직에서 두 개 이상의 전사체를 발현하고 있는, 즉 선택 접합이 일어난 유전자를 확인하였다. 그리고 이 유전자를 대상으로 7 건의 정상 간 샘플에서 전사체의 평균 발현량을 계산한 후, 각 유전자마다 평균 발현량이 가장 많은 전사체를 major 전사체, 두 번째로 많은 전사체를 minor 전사체로 명칭하였다. 간암 환자의 특성 및 분자 기전을 반영하여 환자를 분류하기 위하여 의미가 있는 정보를 가지고 있는(informative) 유전자 세트의 선별 과정이 필요하며, 이 과정은 중요하다. 이를 위해 먼저, 61 건의 간암 샘플에서 major 전사체와 minor 전사체간의 발현량에 대한 비율이 가장 다양한(variable) 유전자를 선별하였다. 이것은 전체 종양 샘플에서 각 유전자의 major 전사체와 minor 전사체의 발현량에 대한 비율(major 전사체 발현량/minor 전사체 발현량)인 fold change (FC) 즉, splicing ratio 에 대한 표준편차(standard deviation)를 유전자마다 계산한 후, 1 이상의 표준편차를 보이는 유전자를 선별하여 해결하였다. 그 다음 선별된 유전자를 대상으로 전체 샘플의 약 10%인 6 명 이상에서 major 전사체가 minor 전사체로의 전위(switching)가 일어나는 유전자를 선별하였다. 이렇게 하여클러스터링(unsupervised hierarchical clustering)을 통해 61 건의 간암 샘플을 2 개의 subtype 으로 분류(classification)하였다.
F. 차등 선택 접합(differentially alternative splicing) 분석
분류된 두 subtype 에서 차등 선택 접합(differentially alternative splicing)이 일어나고 있는 유전자를 확인하기 위해서 t-test p-value < 0.01 인 기준과 두 subtype 의 splicing ratio 에 대한 평균값에 대한 비율이 1 보다 큰 기준을 모두 만족시키는 유전자를 선별하였다. 차등 선택 접합 분석은 R Bioconductor 를 사용하였다. 전체적인 분석 모식도는 그림 1 과 같다(Fig. 2).
G. Functional enrichment 분석
차등 선택 접합이 일어나고 있는 유전자는 DAVID (DAVID: Database for Annotation, Visualization, and Integrated Discovery (Dennis et al., 2003)를 사용하여 Gene Ontology (GO)와 Kyoto Encyclopedia of Genes and Genomes (KEGG) 분석을 시행하였다. 생물학적 유의성(biological significance) 분석을 위해서 p value < 0.05을 사용하였다. 계층형 클러스터링(hierarchical clustering)은 Cluster 3.0 소프트웨어를 사용하였으며 heatmap과 계통도(dendrogram)는 JavaTreeView(1.1.6r4) 소프트웨어를 사용하였다. 차등 선택 접합이 일어나고 있는 유전자의 Network 시각화는 GeneMANIA Cytoscape plugin을 사용하였다(Shannon et al., 2003).
분류된 subtype 사이에서 병리임상학적 인자의 관련성에 대한 통계학적 검정은 Fisher’s exact 검사를 시행하였으며 유의 수준은 p<0.05 으로 판정하였다. 생존 곡선은 Kaplan–Meier 방법으로 구하고, 생존곡선간의 비교는 log-rank 검정을 사용하였다. 상관관계(correlation) 분석은 Pearson correlation coefficient 방법을 사용하였다.
Fig.2. Schematic for classification based on splicing profile. Raw reads sequenced by
Illumina sequencer were checked for read quality and preprocessed using FastQC and FASTX Toolkit, respectively. High quality of reads were mapped and then assembled into transcsripts using Tophat2 and Cufflinks, respectively. In normal tissues, major and minor transcripts per gene were defined according to expression of transcript. Then, genes with highly variable ratio of splicing and switching in HCC tissues were selected. Finally, HCC patients were classified into two subtypes using unsupervised clustering and classifiers were identified.
Ⅲ
. 결과
A. 간암 RNA 서열분석 데이터 분석
간암 환자의 paired-end 리드 RNA 서열분석은 일루미나(Illumina) 플랫폼을 사용하였다. 데이터를 분석하기 전에 서열분석 리드의 염기서열 품질(base sequence quality)을 FastQC 를 사용하여 확인하였다. 염기서열 품질은 Phred quality score 로 계산되는데, 이것은 읽힌(calling) 서열의 염기가 잘못 읽혔을 확률을 점수로 환산하여 나타낸 값이다. 평균 30 (Phred quality score) 이상으로, 이는 염기 서열이 잘못 읽힐 확률이 1/1000 임을 의미하며, 99.9%의 정확도로 서열이 전반적으로 정확하게 읽혀졌음을 확인한 후, 다음 분석을 진행하였다(Fig. 3). 평균 약 6 천만 (60,427,096) 개의 리드가 생산되었고, 그 중 약 81.80 %에 해당하는 평균 약 4 천 9 백만 (48,930,023) 개의 리드가 참조 유전체(hg19)에 맵핑되었다(Table 3). 한 샘플 이상에서 발현하고 있는 전사체는 총 35,694 개로 이는 19,015 유전자에 해당하였다.
B. 분류(classification)
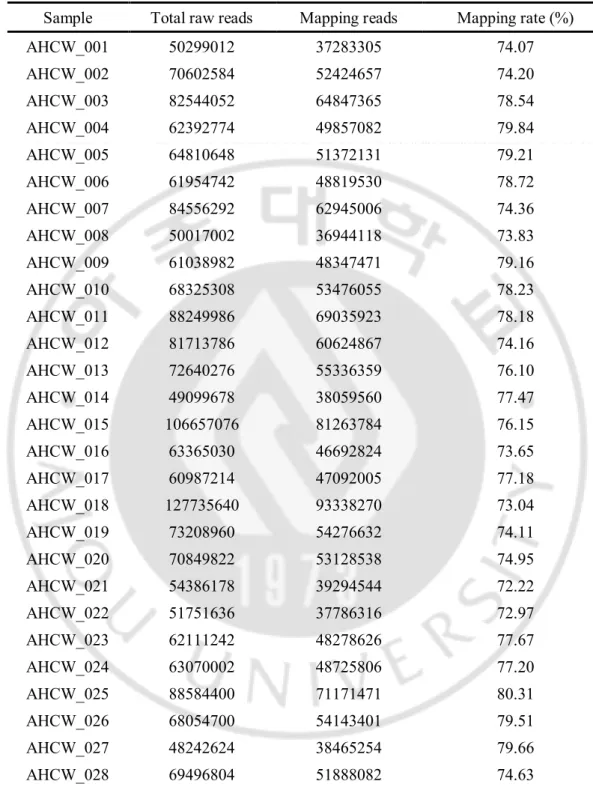
HCC 샘플의 전사체 선택 접합 프로파일링을 위해, 정상 간 조직에서의 전사체 선택 접합의 패턴을 확인하고자 하였다. 7 명의 HCC 환자의 정상 간 조직 mRNA 에서 선택 접합이 일어나 두 개 이상의 전사체를 발현하는 유전자를 대상으로 샘플 전체에서 각 전사체의 평균 발현량이 가장 많은 전사체를 두발현이 가장 많은 전사체를 major 전사체, 그 다음으로 발현이 많은 전사체를 minor 전사체라고 명칭하였다. 그 후, 간암 환자의 특성 및 분자 기전을 반영하여 환자를 분류하기 위하여, major 전사체와 minor 전사체간의 발현량 차이가 61 건의 HCC 샘플에서 가장 다양(variable)하면서 의미가 있는 정보를 가지고 있는 유전자를 선별하고자 하였다. 이를 위해 전체 샘플에서, splicing ratio 를 의미하는, major 전사체와 minor 전사체의 발현량에 대한 비율(major 전사체 발현량/minor 전사체 발현량)인 fold change (FC)에 대한 표준편차(standard deviation)를 유전자마다 구한 후, 1 이상의 표준편차를 보이고 유전자를 선별하였다. 그리고 전체 샘플의 약 10%에 해당하는 6 명 이상에서 major 전사체가 minor 전사체로 전위가 일어나고 있는 772 개의 유전자 세트를 선별하였다. 이 772 개의 유전자의 major 전사체와 minor 전사체 발현량에 대한 비율인 splicing ratio 를 기반으로 비감독 계층형 클러스터링(unsupervised hierarchical clustering)을 실시하여 61 건의 HCC 샘플이 2 개의 subtype 으로 나뉘고 있음을 확인하였다(Fig. 4). Subtype 2 를 보았을 때, subtype 1 보다 정상 조직의 major 전사체에서 minor 전사체로 전위되는 즉, splicing switching 이 일어나는 유전자가 더 많이 존재하고 있음을 확인할 수 있었다(Fig. 4).
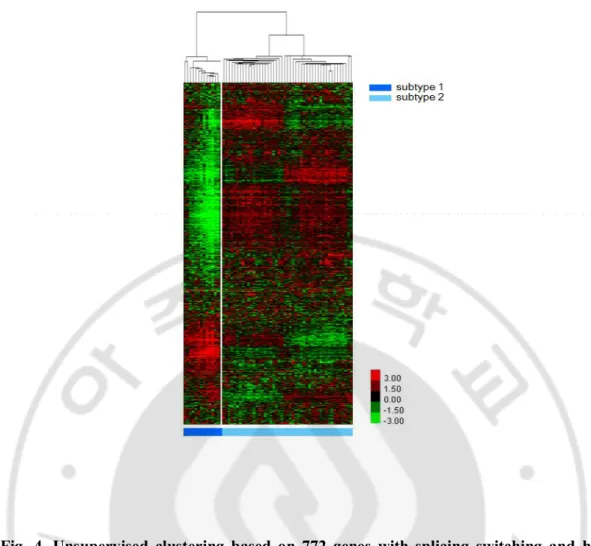
HCC 종양에서 두 전사체의 발현량을 비교해 보았을 때, 두 전사체간의 평균 발현량은 강한 정비례 관계를 보이고 있음을 확인하였다(Pearson’s Correlation coefficient=0.73, p<0.001) (Fig. 5A). 이는 major 전사체의 발현량이 많아질 때, minor 전사체의 발현량이 적어져서 해당 유전자에서 major 전사체가 우세해지는 경향을 보이는 것보다는 major 전사체와 minor 전사체의 발현량이
같은 발현 패턴을 보이고 있음을 의미한다. 그 다음 HCC 종양과 정상 조직에서의 major 와 minor 의 발현량에 대한 비율 패턴을 비교해보았을 때, major 전사체/minor 전사체 발현량에 대한 비율(fold change)이 뚜렷한 양의 상관관계를 보이고 있음을 확인하였다(Pearson’s Correlation coefficient=0.49, p<0.001) (Fig. 5B). 이것은 HCC 환자의 종양 조직에서도 정상 간 조직과 마찬가지로 발현되는 major 전사체와 minor 전사체의 발현량에 대한 비율 패턴이 크게 달라지지 않고, 대체로 그대로 패턴이 유지되고 있음을 의미한다. 즉, 정상 간 조직에서의 major 전사체가 종양 조직에서 minor 전사체로 발현량의 순위가 바뀌는 경우는 많지 않은 것으로 보인다. 그렇기 때문에 종양 샘플에서 major 전사체에서 minor 전사체로 전위가 일어나는 유전자는 암 생물학 측면에서 암에 기여하는 중요한 역할을 할 수 있음을 시사하며, 선택 접합 전위가 일어나서 종양 세포가 선호하는 전사체가 달라짐으로써 세포 증식, 전이, 세포사멸, 생존, 대사 등의 신호전달(signaling)에서 종양 발달에 이점을 얻을 수 있을 것으로 보여진다. 그리고 선택 접합 유형을 분석해 본 결과, 772 개의 유전자 중 약 60%에 해당하는 462 개의 유전자가 7 가지의 선택 접합 유형 중 전사 시작 지점(transcript start site)을 다르게 사용하는 alternative promoters (또는 alternative transcript initiation)에 해당하였다. 또한, 524 (68%)개의 유전자는 선택 접합으로 인해 하나의 유전자에서 서로 다른 단백질을 생산하는 것을 확인되었다. 이것으로 보아 동일한 유전자에서 선택 접합이 일어나 다른 단백질이 생산되어 종양 세포에서 정상 세포와는 다른 기능을 하고 있을 가능성이 있는 것으로 사료된다.
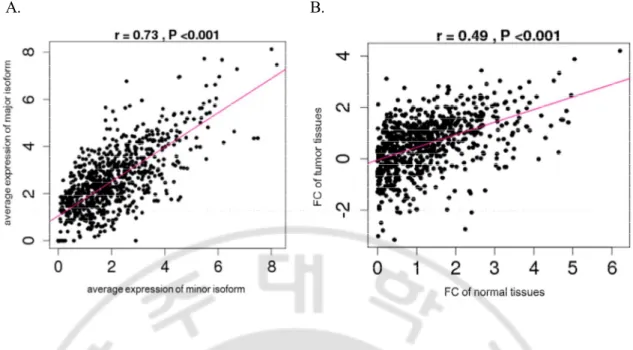
다음으로 두 subtype 간의 임상 관련 정보를 비교하여 보았다. 두 subtype 간의 유의한 임상 및 병리학적인 특징은 24 개월 이내의 재발(recurrence), 종양의 크기, 간내 전이(intrahepatic metastasis)로 확인되었다(Table 4). Subtype 1 에서 24 개월 이내에 재발하고(Fisher’s exact test, p-value=0.002), 종양의 크기는 5 cm 이상이며(Fisher’s exact test, p-value= 0.024), 간내 전이가 있는 환자들이 더 많이 분포하고 있음을 확인하였다(Fisher’s exact test, p-value= 0.033). 간내 전이는 간암 환자의 예후를 불량하게 하는 요인 중 하나이다. 이것으로 보아 subtype 1 이 subtype 2 보다 더 공격적인 종양의 특성을 가진 것으로 생각된다. 그 이외에 성별, 간암의 진단 표지가 되는 AFP (알파태아단백) 수치(> 400 ng/ml), 혈관 침습(invasion) 등 다른 유의한 차이가 나는 임상 및 병리학적인 특징은 확인할 수 없었다. 또한, 두 전사체간의 발현량에 대한 비율에 기반하여 분류된 두 subtype 에서 재발로 인한 생존 기간에 차이를 보이는 지를 알아보기 위하여 Kaplan-meier 생존 분석을 하였다. 그 결과 샘플의 수가 적어서 유의한 값을 얻지는 못하였지만, 곡선상으로는 subtype 1 의 그룹에서 재발 없이 생존하는 기간이 더 짧은 것으로 보아 subtype 1 이 더 공격적인 phenotype 을 보이고 있음을 관찰할 수 있었다(Fig. 6). 하나의 유전자에서 선택 접합이 일어난 2 개의 전사체간의 발현량에 대한 비율이 환자들간에 다양하면서, 선택 접합 전위가 일어나는 772 개의 유전자들이 관여하는 생물학적 기능을 알아보기 위해, DAVID 를 사용하여 GO 와 KEGG 분석을 하였다. 세포사멸의 조절(regulation of apoptosis), 세포 이동의 조절(regulation of cell migration), 세포부착의 조절(regulation of cell adhesion),
세포주기의 조절(regulation of cell cycle)에 해당하는 생물학적 기능에서 유전자들이 과출현(overrepresent)하고 있는 것을 확인할 수 있었다(Table 5). 또한, 국소부착(focal adhesion), 액틴 세포골격의 조절(regulation of actin cytoskeleton), mTOR signaling pathway 를 포함한 암에 관련된 pathway 에 관여하는 유전자에서 선택 접합이 환자들 사이에서 다르게 일어나고 있음을 확인하였다(Table 5).
Table 3. Statistics of HCC data.
Sample Total raw reads Mapping reads Mapping rate (%)
AHCW_001 50299012 37283305 74.07 AHCW_002 70602584 52424657 74.20 AHCW_003 82544052 64847365 78.54 AHCW_004 62392774 49857082 79.84 AHCW_005 64810648 51372131 79.21 AHCW_006 61954742 48819530 78.72 AHCW_007 84556292 62945006 74.36 AHCW_008 50017002 36944118 73.83 AHCW_009 61038982 48347471 79.16 AHCW_010 68325308 53476055 78.23 AHCW_011 88249986 69035923 78.18 AHCW_012 81713786 60624867 74.16 AHCW_013 72640276 55336359 76.10 AHCW_014 49099678 38059560 77.47 AHCW_015 106657076 81263784 76.15 AHCW_016 63365030 46692824 73.65 AHCW_017 60987214 47092005 77.18 AHCW_018 127735640 93338270 73.04 AHCW_019 73208960 54276632 74.11 AHCW_020 70849822 53128538 74.95 AHCW_021 54386178 39294544 72.22 AHCW_022 51751636 37786316 72.97 AHCW_023 62111242 48278626 77.67 AHCW_024 63070002 48725806 77.20 AHCW_025 88584400 71171471 80.31 AHCW_026 68054700 54143401 79.51 AHCW_027 48242624 38465254 79.66 AHCW_028 69496804 51888082 74.63
AHCW_029 89413086 71168235 79.52 AHCW_030 60809538 45607857 74.95 AHCW_031 60816812 48761900 80.18 AHCW_032 58246558 54091580 92.87 AHCW_033 70819258 69209186 97.73 AHCW_034 49305402 42773786 86.75 AHCW_035 43732914 38152049 87.24 AHCW_036 46596464 40880768 87.73 AHCW_037 44483162 38964831 87.59 AHCW_038 47173546 41129295 87.19 AHCW_039 48344898 42018824 86.91 AHCW_040 44096796 37709149 85.51 AHCW_041 46586352 40944720 87.89 AHCW_042 64165018 45079050 70.25 AHCW_043 54792982 46815546 85.44 AHCW_044 50416494 43159773 85.61 AHCW_045 51473022 44339680 86.14 AHCW_046 46577462 39197566 84.16 AHCW_047 57796962 43847642 75.86 AHCW_048 46547060 35373855 76.00 AHCW_049 46991460 34924800 74.32 AHCW_050 46917086 35511606 75.69 AHCW_051 48556368 36591786 75.36 AHCW_052 51057462 39115691 76.61 AHCW_053 42519988 41501091 97.60 AHCW_054 47996398 47262839 98.47 AHCW_055 54794062 43694961 79.74 AHCW_056 35474790 34425445 97.04 AHCW_057 53604692 52624078 98.17 AHCW_058 55590868 54365422 97.8 AHCW_059 54134258 52830985 97.59 AHCW_060 56912746 55764720 97.98
AHCW_061 52562420 51973706 98.88
Total 3686052834 2984731404 -
Fig. 4. Unsupervised clustering based on 772 genes with splicing switching and high variability among samples and classification. 61 HCCs can be separated in to two distinct
subtypes. This shows heatmap of 772 genes with splicing switching and high variability among samples. Major transcript means the most expressed transcript in normal tissue and minor transcript means that of second expressed transcript. The red part of this figure shows the major transcript is more expressed than minor transcript, and the green part shows the minor transcript is more expressed than major transcript.
A. B.
Fig. 5. The distribution of major and minor transcripts. A. The correlation of average
expression of major and minor transcripts in HCC tumor tissues. B. The correlation of splicing ratio (major transcript expression/minor transcript expression) of HCC tumor and normal tissues. Here, FC (fold change) is referred to as splicing ratio.
Table 4. Relationship between subtypes and clinicopathologic variables of patients.
Variable Subtype1 (n=14)
Subtype2
(n=47) P value Odd Ratio 95% CI Recurrence in 24 months YES 10 11 0.002* 7.62 1.77-40.33 NO 4 35 Tumor size >5 cm 9 13 0.024* 4.57 1.128-20.93 <=5 cm 5 34 MILAN criteria 1 10 20 0.073 3.31 0.80-16.60 0 4 27 Intrahepatic metastasis YES 5 4 0.033* 5.24 0.90-33.27 NO 8 35
Portal vein invasion
YES 7 15 0.343 2.04 0.50-8.27
NO 7 31
Microvascular invasion
YES 9 25 0.555 1.50 0.37-6.63
NO 5 21
Bile duct invasion
YES 0 4 0.562 0.00 0.00-4.61 NO 14 38 AFP > 400 ng/ml 6 17 0.756 1.32 0.31-5.20 < 400 ng/ml 8 30 Gender Male 9 38 0.277 0.43 0.09-2.05 Female 5 9
Fig. 6. Kaplan–Meier survival curves for recurrence-free survival between two subtypes. Log-rank test showed insignificant differences between the two subtypes
Table 5. Significant biological processes and KEGG pathway of 772 genes for classification.
Term P value
GO:0042981~regulation of apoptosis 2.86x10-6
GO:0030155~regulation of cell adhesion 1.45x10-5 GO:0030334~regulation of cell migration 1.69x10-4
GO:0051726~regulation of cell cycle 1.15x10-2
hsa04510:Focal adhesion 1.45x10-3
hsa05200:Pathways in cancer 3.65x10-3
hsa04810:Regulation of actin cytoskeleton 6.98x10-3
C. 차등 선택 접합(differentially alternative splicing) 분석
2 개의 subtype 에서 차등 선택 접합(differentially alternative splicing)이 일어나고 있는 유전자 및 생물학적 기능을 알아보기 위해, t-test p value < 0.01 기준과 두 그룹간의 splicing ratio 에 대한 평균 비율이 1 보다 큰 기준을 모두 만족하는 1,043 개 유전자를 선별하였다. 1,043 개의 유전자 중 283 개는 subtype 2 보다 더 공격적인 종양의 특징을 보이는 그룹인 subtype 1 에서 major 전사체를 minor 전사체보다 더 높게 발현하고 있었고(major 전사체의 발현량 > minor 전사체의 발현량), 760 개 유전자는 minor 전사체를 major 전사체보다 더 높게 발현하고 있었다(major 전사체의 발현량 < minor 전사체의 발현량)(Fig. 7). 다시 말하면, 예후가 더 좋지 않은 subtype 1 에서 760 개의 유전자가 major 전사체에서 minor 전사체로 전위가 일어나 있음을 확인하였다.
두 subtype 간의 차등 선택 접합이 일어난 유전자의 생물학적 기능(biological function)을 이해하여 두 그룹의 특징을 알아보고자 DAVID 를 사용하여 GO 분석을 시행하였다. GO 분석은 유전자 세트 분석에 대한 기능적 주석(functional annotation)과 분류(classification)에 대한 정보를 제공해준다. Subtype 1 에서 하나의 유전자의 선택 접합된 두 전사체간에서 전위가 일어난 유전자의 GO term 은 세포부착(cell adhesion), 세포이동(cell migration), 콜라겐 대사과정(collagen metabolic process), 세포사멸(apoptosis)로 이에 관련된 유전자가 과출현하고 있었으며(Fig. 8) 종양에서 변화가 일어난다고 알려진 대사(metabolism)의 글루코오스 대사 과정(glucose metabolic process)의 GO term 에 해당하는 유전자가 과출현하고
있었다(Warburg, 1956). KEGG pathway 로는 인슐린 신호과정(Insulin signaling) pathway, mTOR signaling pathway 에서 두 그룹간의 차등 선택 접합이 일어나는 유전자가 관여하고 있음을 확인하였다(Fig. 9). Subtype 2 에서는 세포주기(cell cycle)에 관여하는 유전자에서 선택 접합이 일어난 두 전사체간의 전위가 일어나고 있었다(Fig. 10). 결론적으로, 암 발달에 영향을 미치는 생물학적 기능을 가진 유전자에서 샘플간의 차등 선택 접합이 일어나고 있음을 확인하였다.
두 그룹간에 차등 선택 접합이 일어난 유전자 목록 중 WNT5A 가 포함되어 있었다. 본 연구에서 WNT5A 는 정상 샘플에서 WNT5A-L 이 major 전사체로, WNT5A-S 는 minor 전사체로 발현되고 있었다(Fig. 11). WNT5A 는 종양의 증식과 전이에 관여하는 유전자로 선택 접합이 일어나면 짧은 길이의 WNT5A-S 와 긴 길이의 WNT5A-L 이 생산된다. 이 두 단백질이 종양 증식에 서로 상반된 기능을 한다고 알려져 있다(Bauer et al., 2013). 더 공격적인 종양의 특성을 갖는 subtype 1 에서 WNT5A-L:WNT5A-S 의 비율이 정상 간 조직에서와 다르게 감소되어 있었다. 요약하자면, 차등 선택 접합 분석으로 WNT5A 유전자를 확인할 수 있었고, 임상적인 예후가 좋지 않은 subtype 1 에서 anti-proliferation 기능을 하는 WNT5A-L 에서 pro-proliferation 기능을 하는 WNT5A-S 로 전위가 일어나 있는 것을 확인하였다.
Fig. 7. The heatmap of 1,043 genes with differentially alternative splicing between two subtypes. The genes with splicing switching is shown in green. In subtype 2, the number of
genes with splicing switching is 283, and that of subtype 1 is 760. The number in parentheses is the p-value from DAVID. In subtype 1, the genes with splicing switching involved in cell adhesion, cell migration, collagen metabolic process, apoptosis, glucose metabolic process and insulin/mTOR signaling pathway and in subtype 2, involved in cell cycle.
Fig. 8. Network of apoptosis in splicing switching in subtype 1. Network was visualized
by GeneMANIA Cytoscape plugin. Nodes represent genes involved in the apoptosis function, and edges represent the relationships between linked genes.
Fig. 9. Network of Insulin/mTOR signaling pathway in splicing switching in subtype 1.
Network was visualized by GeneMANIA Cytoscape plugin. Nodes represent genes involved in Insulin/mTOR signaling pathway, and edges represent the relationships between linked genes.
Fig. 10. Network of cell cycle in splicing switching in subtype 2. Network was visualized
by GeneMANIA Cytoscape plugin. Nodes represent genes involved in the cell cycle function, and edges represent the relationships between linked genes.
Fig. 11. The splicing pattern of WNT5A. WNT5A-L is a major transcript and WNT5A-S
is a minor transcript in the present study. WNT5A-L is a transcript with anti-proliferative effect and WNT5A-S is a transcript with pro-proliferative effect.
Ⅳ
. 고찰
RNA 서열분석은 전체 전사체 및 선택 접합 분석을 가능하게 한다. 선택 접합은 단백질의 구조, 기능, 분포(localization)의 변화를 가져와서 질병, 암, 약물 내성에 영향을 미친다고 알려져 있다(Gabut et al., 2011; Buljan et al., 2012; Ellis et al., 2012) 그러므로 선택 접합에 대한 연구는 암의 진단, 예후, 치료의 이해에 대해 기여할 수 있다(Bonomi et al., 2013). 본 연구에서는 RNA 서열분석을 사용하여 61 명의 HCC 환자의 전사체 선택 접합을 프로파일링을 하였고, 이를 기반으로 최초로 간암 환자를 임상적 예후가 다른 그룹으로 분류하였다. 간암 종양 조직에서 정상 간 조직과 다르게 조절되는 선택 접합된 전사체를 조사하고, 간암 샘플 내에서도 다르게 조절되는 선택 접합 유전자와 이에 해당하는 생물학적 기능을 알아보고자 하였다. 이를 위해, 정상 간 조직에서 선택 접합이 일어나는 유전자를 확인한 후, 각 유전자의 major 전사체와 minor 전사체의 발현량에 대한 비율이 간암 종양 샘플에서 가장 다양(variable)하면서, major 전사체에서 minor 전사체로 전위가 일어나는 772 개의 유전자를 선별하였다. 이에 해당하는 유전자의 기능적인 면을 살펴보기 위하여 GO 분석을 실시한 결과 세포사멸의 조절(Regulation of apoptosis), 세포 이동의 조절(regulation of cell migration), 세포 부착의 조절(regulation of cell adhesion), 세포 주기의 조절(regulation of cell cycle)과 관련된 유전자들이 enriched 되어 있음을 확인하였다(Table 5). 세포 부착의 조절과 세포 이동의 조절은 암의 침습과 관련된 기능이며, 세포사멸 또한 종양세포에서 비정상적으로 조절되는 기능에 해당한다. 이렇게 간암 환자의 종양 조직에서 정상 간 조직과 달리 major
전사체가 minor 전사체로 전위되는 유전자는 종양 세포가 암에서 생존하는 데 이점을 줄 수 있는 것으로 사료된다. 종양 세포는 종양 형성을 위해 정상적인 세포사멸을 회피하며, 암에서 선택 접합된 전사체가 세포사멸을 비정상적으로 조절하는 사례가 많이 알려져 있다(Takehara et al., 2001). 예를 들어, 리간드 및 receptor (FasL, Fas, LARD), adaptor 단백질 및 regulator (TRAF2, TRAF3, MyDD88, MADD, Apaf-1, survivin, Smac/Diablo), Bcl-2 family (Bcl-x, Bak, Bid, Bim), caspase 및 caspase-like 단백질(caspase-2, caspase-9, caspase-10, FLIP), caspase 타겟 단백질(ICAD, CAD, Acinus) 등 세포사멸에 관여하는 많은 유전자에서 선택 접합이 일어나는 것이 보고 되어 있다(Schwerk and Schulze-Osthoff, 2005). 또한, 세포사멸은 선택 접합을 통해 세포 주기 과정과 연결되어 있다고 알려져 있으며(Moore et al., 2010), 세포 주기 과정에서 선택 접합이 일어난 유전자는 p53 family 인 p53, p63, p73 이 있다(Courtois et al., 2004). 본 연구의 간암 샘플에서 기존에 세포사멸과 세포 주기에서 선택 접합이 일어난다고 알려진 유전자가 많이 포함되어 있지는 않았지만, 이는 기존의 많은 암 연구가 전사체 수준이 아닌 특정 유전자에 초점을 맞춘 연구이기 때문에 알려진 전사체의 수가 적기 때문일 수 있다. 본 연구에서 확인된 유전자인 CASP2, CASP9 등 알려진 유전자가 포함되어 있었으며, 이 생물학적 과정에서 선택 접합이 정상과 다르게 영향을 받고 있음을 확인할 수 있었다. 또한, 이 유전자 세트를 기반으로 비감독
클러스터링을 하여 분류된 두 subtype 간에 Fisher test 를 실시하여
임상병리학적인 차이를 알아본 결과(Fig. 4), subtype 1 이 24 개월 이내에 재발이 일어나며, 종양의 크기가 더 크고, 간내 전이가 있음과 상관관계가 있음을 확인하였다(Table 4). 이것으로 보아 전사체간의 전위가 일어난 유전자에 기반한
분류로 확인된 subtype 1 에 해당하는 환자들이 subtype 2 의 환자들보다 더 공격적인 암의 phenotype 을 가지고 있으며 예후가 더 좋지 않을 것으로 보여진다. 이러한 결과를 종합해 볼 때, 본 연구는 선택 접합 프로파일을 기반으로 비감독 클러스터링을 사용하여 환자를 분류하는 것이 임상적 예후에서 유의하게 차이가 나는 환자들의 subtype 을 밝혀낼 수 있음을 제시하고 있다. 그 후, 두 subtype 에서 다르게 조절되고 있는 전사체 선택 접합이 일어나는 유전자를 확인하고 생물학적 기능을 확인하여 두 subtype 의 특징을 이해하기 위해서, 차등 선택 접합이 일어난 1,043 개의 유전자를 조사하였다. Subtype 2 에서 subtype 1 에 비해 대부분의 유전자에서 major 전사체의 발현이 minor 전사체의 발현보다 높은 반면에, subtype 1 은 암의 침습과 관련된 세포부착, 세포이동, 세포사멸, 글루코오스 대사 과정에 관여하는 유전자에서 major 전사체에서 minor 전사체로의 전위가 일어나고 있음을 확인하였다(Fig. 7). 또한, 인슐린 신호과정과 mTOR signaling pathway 에 관여하는 유전자에서 두 그룹간에 차등 선택 접합에 영향을 미치고 있음을 확인하였다(Fig 9). 비록 mTOR/인슐린 신호과정에서 선택 접합이 일어난다고 알려진 유전자가 선별된 차등 선택 접합 유전자 목록에 포함되어 있지는 않았지만, 이 신호과정은 암에서 선택 접합의 영향을 받고 있음이 보고 되어 있다(Panasyuk et al., 2009; Ben-Hur et al., 2013). 그 예로 mTOR 유전자의 선택 접합으로 생긴 mTORbeta 는 종양 유전자로의 기능을 하며, mTORalpha 와는 달리 세포 주기의 G1 기를 조절하여 종양 세포의 증식을 증가시킴이 보고되어 있다(Panasyuk et al., 2009). 그리고 S6K1 도 선택 접합이 일어나 길이가 짧은 전사체를 만들어 mTORC1 과 결합하여 활성을 증가시킬 수
있으며, 4E-BP1 의 비활성화와 몇 가지 종양 유전자 및 anti-apoptotic 기능을 하는 유전자의 단백질 번역을 증가시킨다고 보고되어 있다(Ben-Hur et al., 2013). 또한,
인슐린 내성이 증가할 경우 간암의 발병률이 높아진다는 보고가
있으며(Nkontchou et al., 2010; Chao et al., 2011), 인슐린 신호과정 중 선택 접합에 대해서 연구된 유전자로는 인슐린 수용체(insulin receptor; IR)가 있다. 인슐린 수용체(IR)는 선택 접합이 일어나면, 엑손 11 번이 포함되지 않는 IR-A 와 포함되는 IR-B, 두 가지 전사체를 만든다(Belfiore et al., 2009). IR-A 는 proliferative 효과를 가지는 반면에, IR-B 는 인슐린의 대사 효과(metabolic effect)를 보인다고 알려져 있으며, HCC 종양에서 IR-B 에서 IR-A 로의 전위가 자주 관찰된다고 보고 되어 있다(Belfiore et al., 2009). 하나의 유전자에서 선택 접합이 일어난 major 전사체와 minor 전사체는 포함되거나 배제되는 엑손의 차이로 인해 암에 기여하는 도메인(domain)의 차이를 가져올 수 있고, 이러한 도메인의 존재 유무에 따라 단백질간의 상호 작용이 달라지기도 하며, 도메인이 catalytic 자리인 경우에는 단백질의 활성에 영향을 미칠 수 있다(Venables, 2004). 또한, 선택 접합으로 포함되거나 배제되는 서열에 핵 또는 세포질로 이동하는 서열이 포함되어 있는 경우에 세포 내에서 단백질의 분포(localization)에 영향을 받게 된다(Venables, 2004). 그렇기 때문에 같은 유전자에서 발현되었더라도 선택 접합이 일어난 서로 다른 전사체는 잠정적으로 다양한 기능을, 심지어는 동일한 기능에 대해 상반된 역할을 할 수 있다(Feng et al., 2013). Subtype 1 에서 major 와 minor 전사체간의 전위가 일어난 WNT5A 는 Wnt 신호과정에 관여하는 리간드로 종양의 발달에 기여하는 것으로
알려진 유전자이다(Bi et al., 2014). WNT5A 는 유방암, 위암, 췌장암, 비소세포폐암에서 종양 유전자(oncogene)로 기능하며, 과발현되면 종양의 증식, 침습, 전이 그리고 종양 성장과 전이에 필요한 혈관 신생(angiogenesis)을 증가시킨다고 알려져 있지만(Fernandez-Cobo et al., 2007; Kanzawa et al., 2013; Lee et al., 2014), 이와는 상이하게 결장암에서는 체세포 결실(somatic deletion)과
hypermethylation 으로 인해 비활성화(inactivation)되어 있으며, 종양 억제
유전자(tumor suppressor gene)의 역할을 한다고 알려져 있다(Ying et al., 2008; Liao et al., 2011; Stefater et al., 2011; Abdelmaksoud-Dammak et al., 2014; Bi et al., 2014; Cheng et al., 2014). Wnt 신호과정은 간암에서 비정상적으로 활성화된다고 알려진 기전 중 하나이며(Takigawa and Brown, 2008), 간암에서 WNT5A 는 종양 억제 유전자로 작용한다고 알려져 있다(Geng et al., 2012). WNT5A 는 증식과 혈관 신생에 관여하는 유전자로 선택적인 프로모터를 사용하며, 서로 다른 전사 시작 자리를 사용하여 선택 접합이 일어나서 길이가 긴 전사체인 WNT5A-L 과 길이가 짧은 전사체인 WNT5A-S 인 2 개의 단백질을 생산되는데, 이 두 개의 단백질의 기능이 상반되는 것으로 보고 되어 있다(Bauer et al., 2013). WNT5A-L 은 종양 증식을 저해하는 반면에, WNT5A-S 는 종양의 성장을 증가시킨다(Bauer et al., 2013). 본 연구에서 WNT5A 는 정상 샘플에서 WNT5A-L 이 major 전사체로, WNT5A-S 는 minor 전사체로 발현되고 있었다. 더 공격적인 종양의 특성을 갖는 subtype 1 에서 anti-proliferation 기능을 하는 WNT5A-L 이 pro-proliferation 기능을 하는 WNT5A-S 로 접합 전위가 일어나는 것을 확인할 수 있었다(Fig. 11). 이것으로 보아 추후 기능 실험이 추가되어야 하겠지만, WNT5A 유전자의 전사체 선택 접합이 HCC 의 증식과 전이에 영향을 미칠 수 있을 것으로 시사된다. 또한,
간암 종양 조직에서 정상 조직과 다르게 major 전사체에서 minor 전사체로의 전위가 일어난 유전자 목록에 이미 암에서 선택 접합이 중요한 역할을 한다고 알려진 유전자인 세포 주기 check point 의 기능을 하는 BRCA1 과 암의 침습에 두 개의 단백질이 다른 기능을 한다고 알려진 ERBB3 와 세포사멸에 상반된 기능을 한다고 보고된 CASP9 (capase9)이 포함되어 있었다(Orban and Olah, 2003; Lin et al., 2008; Shultz et al., 2010). 본 연구에서는 간암 환자들에서 다르게 선택 접합이 일어난 전사체로 인해 간암 발달에 다른 영향을 미칠 수 있음을 제시하였다. 이상의 연구 결과들은 HCC 의 환자들간의 차등 선택 접합이 일어나고 있는 유전자가 암과 관련된 생물학적 과정에 관여하고 있음을 제시하고 있으며, 선택 접합이 일어난 유전자에 대한 전사체 수준에서의 연구가 간암의 이질성과 관련한 분자 기전을 규명하는 데에 새로운 방향성을 제시할 수 있음을 시사해 주고 있다. 또한, 선택 접합 프로파일링에 기반한 간암 환자의 분류가 간암 환자간의 임상 병리학적으로 다른 특성을 지닌 subtype 을 예측하는 데 유용할 수 있음을 시사한다.
Ⅴ
. 결론
본 논문에서는 RNA 서열분석을 사용하여 간암 환자의 전사체 및 선택 접합 프로파일링을 하였고, 하나의 유전자에서 선택 접합이 일어난 두 전사체간의 발현량에 대한 비율에 기반하여 환자들을 분류하는 것이 환자들의 임상적 예후 차이를 밝혀줄 수 있음을 확인하였다. 한 유전자에서 선택 접합된 두 전사체간의 발현량에 대한 비율을 기반으로 한 간암 환자의 분류 방법이 환자의 특성과 분자기전을 이해하는 데 유용할 수 있음을 제시하고, 선택 접합이 환자에 따른 간암의 발생기전 및 종양 발달에 기여하는 기전을 규명하는 데에 중요한 영향을 미칠 수 있음을 시사한다.참고문헌
1. 통계청, 2012 년 사망원인통계, 2013
2. Abdelmaksoud-Dammak R, Miladi-Abdennadher I, Saadallah-Kallel A, Khabir A, Sellami-Boudawara T, Frikha M, Daoud J, Mokdad-Gargouri R: Downregulation of WIF-1 and Wnt5a in patients with colorectal carcinoma: clinical significance.
Tumour Biol, 2014
3. Agrawal D, Chen T, Irby R, Quackenbush J, Chambers AF, Szabo M, Cantor A, Coppola D, Yeatman TJ: Osteopontin identified as lead marker of colon cancer progression, using pooled sample expression profiling. J Natl Cancer Inst 94: 513-521, 2002
4. Bauer M, Benard J, Gaasterland T, Willert K, Cappellen D: WNT5A encodes two isoforms with distinct functions in cancers. PLoS One 8: e80526, 2013
5. Belfiore A, Frasca F, Pandini G, Sciacca L, Vigneri R: Insulin receptor isoforms and insulin receptor/insulin-like growth factor receptor hybrids in physiology and disease. Endocr Rev 30: 586-623, 2009
6. Ben-Hur V, Denichenko P, Siegfried Z, Maimon A, Krainer A, Davidson B, Karni R: S6K1 alternative splicing modulates its oncogenic activity and regulates mTORC1.
Cell Rep 3: 103-115, 2013
7. Bi L, Liu X, Wang C, Cao Y, Mao R, Li P, Geng M: Wnt5a involved in regulation of the biological behavior of hepatocellular carcinoma. Int J Clin Exp Pathol 7:
987-995, 2014
8. Boise LH, Gonzalez-Garcia M, Postema CE, Ding L, Lindsten T, Turka LA, Mao X, Nunez G, Thompson CB: bcl-x, a bcl-2-related gene that functions as a dominant regulator of apoptotic cell death. Cell 74: 597-608, 1993
9. Bonomi S, Gallo S, Catillo M, Pignataro D, Biamonti G, Ghigna C: Oncogenic alternative splicing switches: role in cancer progression and prospects for therapy.
Int J Cell Biol 2013: 962038, 2013
10. Buljan M, Chalancon G, Eustermann S, Wagner GP, Fuxreiter M, Bateman A, Babu MM: Tissue-specific splicing of disordered segments that embed binding motifs rewires protein interaction networks. Mol Cell 46: 871-883, 2012
11. Castillo J, Goni S, Latasa MU, Perugorria MJ, Calvo A, Muntane J, Bioulac-Sage P, Balabaud C, Prieto J, Avila MA, Berasain C: Amphiregulin induces the alternative splicing of p73 into its oncogenic isoform DeltaEx2p73 in human hepatocellular tumors. Gastroenterology 137: 1805-1815 e1801-1804, 2009
12. Chambers AF, Wilson SM, Kerkvliet N, O'Malley FP, Harris JF, Casson AG: Osteopontin expression in lung cancer. Lung Cancer 15: 311-323, 1996
13. Chao LT, Wu CF, Sung FY, Lin CL, Liu CJ, Huang CJ, Tsai KS, Yu MW: Insulin, glucose and hepatocellular carcinoma risk in male hepatitis B carriers: results from 17-year follow-up of a population-based cohort. Carcinogenesis 32: 876-881, 2011 14. Cheng R, Sun B, Liu Z, Zhao X, Qi L, Li Y, Gu Q: Wnt5a Suppresses Colon Cancer
by Inhibiting Cell Proliferation and Epithelial-Mesenchymal Transition. J Cell
15. Cheung HC, Baggerly KA, Tsavachidis S, Bachinski LL, Neubauer VL, Nixon TJ, Aldape KD, Cote GJ, Krahe R: Global analysis of aberrant pre-mRNA splicing in glioblastoma using exon expression arrays. BMC Genomics 9: 216, 2008
16. Chu X, Zhang T, Wang J, Li M, Zhang X, Tu J, Sun S, Chen X, Lu F: Alternative splicing variants of human Fbx4 disturb cyclin D1 proteolysis in human cancer.
Biochem Biophys Res Commun 447: 158-164, 2014
17. Courtois S, Caron de Fromentel C, Hainaut P: p53 protein variants: structural and functional similarities with p63 and p73 isoforms. Oncogene 23: 631-638, 2004 18. David CJ, Manley JL: Alternative pre-mRNA splicing regulation in cancer:
pathways and programs unhinged. Genes Dev 24: 2343-2364, 2010
19. Davuluri RV, Suzuki Y, Sugano S, Plass C, Huang TH: The functional consequences of alternative promoter use in mammalian genomes. Trends Genet 24: 167-177, 2008
20. Dennis G, Jr., Sherman BT, Hosack DA, Yang J, Gao W, Lane HC, Lempicki RA: DAVID: Database for Annotation, Visualization, and Integrated Discovery. Genome
Biol 4: P3, 2003
21. Dubuc AM, Morrissy AS, Kloosterhof NK, Northcott PA, Yu EP, Shih D, Peacock J, Grajkowska W, van Meter T, Eberhart CG, Pfister S, Marra MA, Weiss WA, Scherer SW, Rutka JT, French PJ, Taylor MD: Subgroup-specific alternative splicing in medulloblastoma. Acta Neuropathol 123: 485-499, 2012
22. El-Serag HB, Rudolph KL: Hepatocellular carcinoma: epidemiology and molecular carcinogenesis. Gastroenterology 132: 2557-2576, 2007
23. Ellis JD, Barrios-Rodiles M, Colak R, Irimia M, Kim T, Calarco JA, Wang X, Pan Q, O'Hanlon D, Kim PM, Wrana JL, Blencowe BJ: Tissue-specific alternative splicing remodels protein-protein interaction networks. Mol Cell 46: 884-892, 2012
24. Eswaran J, Cyanam D, Mudvari P, Reddy SD, Pakala SB, Nair SS, Florea L, Fuqua SA, Godbole S, Kumar R: Transcriptomic landscape of breast cancers through mRNA sequencing. Sci Rep 2: 264, 2012
25. Eswaran J, Horvath A, Godbole S, Reddy SD, Mudvari P, Ohshiro K, Cyanam D, Nair S, Fuqua SA, Polyak K, Florea LD, Kumar R: RNA sequencing of cancer reveals novel splicing alterations. Sci Rep 3: 1689, 2013
26. Feng H, Qin Z, Zhang X: Opportunities and methods for studying alternative splicing in cancer with RNA-Seq. Cancer Lett 340: 179-191, 2013
27. Fernandez-Cobo M, Zammarchi F, Mandeli J, Holland JF, Pogo BG: Expression of Wnt5A and Wnt10B in non-immortalized breast cancer cells. Oncol Rep 17: 903-907, 2007
28. Gabut M, Samavarchi-Tehrani P, Wang X, Slobodeniuc V, O'Hanlon D, Sung HK, Alvarez M, Talukder S, Pan Q, Mazzoni EO, Nedelec S, Wichterle H, Woltjen K, Hughes TR, Zandstra PW, Nagy A, Wrana JL, Blencowe BJ: An alternative splicing switch regulates embryonic stem cell pluripotency and reprogramming. Cell 147: 132-146, 2011
29. Garcia-Blanco MA, Baraniak AP, Lasda EL: Alternative splicing in disease and therapy. Nat Biotechnol 22: 535-546, 2004
protein in hepatocellular carcinoma associated with poor prognosis. World J
Gastroenterol 18: 1328-1338, 2012
31. Goodstadt L, Ponting CP: Phylogenetic reconstruction of orthology, paralogy, and conserved synteny for dog and human. PLoS Comput Biol 2: e133, 2006
32. Graveley BR: Alternative splicing: increasing diversity in the proteomic world.
Trends Genet 17: 100-107, 2001
33. Guo X, Chen QR, Song YK, Wei JS, Khan J: Exon array analysis reveals neuroblastoma tumors have distinct alternative splicing patterns according to stage and MYCN amplification status. BMC Med Genomics 4: 35, 2011
34. Hanoun N, Bureau C, Diab T, Gayet O, Dusetti N, Selves J, Vinel JP, Buscail L, Cordelier P, Torrisani J: The SV2 variant of KLF6 is down-regulated in hepatocellular carcinoma and displays anti-proliferative and pro-apoptotic functions.
J Hepatol 53: 880-888, 2010
35. He X, Semenov M, Tamai K, Zeng X: LDL receptor-related proteins 5 and 6 in Wnt/beta-catenin signaling: arrows point the way. Development 131: 1663-1677, 2004
36. Hijiya N, Setoguchi M, Matsuura K, Higuchi Y, Akizuki S, Yamamoto S: Cloning and characterization of the human osteopontin gene and its promoter. Biochem J 303 ( Pt 1): 255-262, 1994
37. Ito T, Hashimoto Y, Tanaka E, Kan T, Tsunoda S, Sato F, Higashiyama M, Okumura T, Shimada Y: An inducible short-hairpin RNA vector against osteopontin reduces metastatic potential of human esophageal squamous cell carcinoma in vitro and in