저작자표시-비영리-변경금지 2.0 대한민국 이용자는 아래의 조건을 따르는 경우에 한하여 자유롭게 l 이 저작물을 복제, 배포, 전송, 전시, 공연 및 방송할 수 있습니다. 다음과 같은 조건을 따라야 합니다: l 귀하는, 이 저작물의 재이용이나 배포의 경우, 이 저작물에 적용된 이용허락조건 을 명확하게 나타내어야 합니다. l 저작권자로부터 별도의 허가를 받으면 이러한 조건들은 적용되지 않습니다. 저작권법에 따른 이용자의 권리는 위의 내용에 의하여 영향을 받지 않습니다. 이것은 이용허락규약(Legal Code)을 이해하기 쉽게 요약한 것입니다. Disclaimer 저작자표시. 귀하는 원저작자를 표시하여야 합니다. 비영리. 귀하는 이 저작물을 영리 목적으로 이용할 수 없습니다. 변경금지. 귀하는 이 저작물을 개작, 변형 또는 가공할 수 없습니다.
이학 석사학위 논문
유전자 발현 프로파일 데이터를
이용한 조건 특이적인 단백질-단백질
상호작용 예측방법
아 주 대 학 교 대 학 원
의학과/의학전공(이)
정 우 진
유전자 발현 프로파일 데이터를 이용한
조건 특이적인 단백질-단백질 상호작용
예측방법
지도교수 이 기 영
이 논문을 이학 석사학위 논문으로 제출함.
2013 년 8 월
아 주 대 학 교 대 학 원
의학과/의학전공(이)
정 우 진
정우진의 이학 석사학위 논문을 인준함.
심사위원장 이 기 영 인
심사위원 서 해 영 인
심사위원 박 래 웅 인
심사위원 박 선 인
아 주 대 학 교 대 학 원
2013 년 6 월 21 일
- 국문 요약 -
유전자 발현 프로파일 데이터를 이용한 조건 특이적인
단백질-단백질 상호작용 예측 방법
단백질-단백질 상호작용(PPIs, protein-protein interactions)은 세포 내에서 방 대한 네트워크를 이루는 기능적인 핵심 단위이며, 각 세포의 기능 및 상태는 세 포 내 PPIs 네트워크 구성에 따라 달라진다. 현재까지 공개된 대부분의 PPIs 데 이터베이스에는 개별 PPI가 관찰된 세포조건에 대한 주석정보가 부재하며, 이는 특정 세포의 PPIs를 정확히 이해하는데 제약점이 되고 있다. 한편, 유전자 발현 프로파일(GEP, gene expression profiles) 데이터는 여러 세포조건을 가지고 있지만 이를 이용한 조건 특이적인 PPIs 예측정도를 조사한 연구는 많지 않다.
본 연구에서는 줄기세포 및 종양세포에서 조건 특이적인 PPIs 데이터를 구 축하고, 이 중 배아 줄기세포(ESC, embryonic stem cell)의 GEP 를 이용한 ESC-특 이적 PPIs 예측정도를 여러 정규화 방법 (Normalization methods) 및 유전자 공동 발현패턴 (Gene co-expression pattern) 측정법을 통해 종합적으로 비교분석하였다. 이를 바탕으로 GEP 데이터를 이용한 유전체 수준의 조건 특이적인 PPIs를 예측 하는데 있어 적합한 방법론을 제안하는데 그 연구목적이 있다.
조건 특이적인 PPIs 데이터 구축을 위해 주요 공개 PPIs 데이터베이스를 통합한 후, 논문초록에서 줄기세포 및 종양세포 관련 키워드로 모두 검색하였다. 이어, 논문들을 직접 큐레이션한 후 개별 PPI에 줄기세포 및 종양세포의 세부 종 류(subtype)까지 주석정보를 추가하였다. GEP 데이터는 NCBI GEO 에서 수집한 후, 실험대상을 관심 세포조건별로 정리하였다. 그 중 ESC에 해당하는 GEP 데이터
를 6가지 정규화 방법으로 전처리하고, 각각의 데이터를 이용하여 ESC-특이적 PPIs의 공동발현 정도를 6가지 측정방법으로 계산하였다. 공동발현 측정결과는 ESC-특이적 PPIs, 일반적인 조건에서의 PPIs (General PPIs), GEP 데이터에서의 임 의 유전자 쌍 그리고 단백질 상호작용이 없는 유전자 쌍에 걸쳐서 비교하고, ESC-특이적 PPIs 에서 가장 높은 발현 연관성을 가지는 동시에 General PPIs 의 발현 연관성과 가장 유의한 차이를 보이는 정규화 및 공동발현 측정법을 선정하 였다. 먼저, 구축된 조건 특이적인 PPIs 데이터는 총 4,161의 단백질과 8,347 개 의 단백질 상호작용으로 구성되며, ESC의 경우 371 개의 단백질과 603 개의 상호 작용에 해당하며, 이는 기존에 알려진 줄기세포 중 가장 큰 네트워크이다. 정규 화 및 전처리된 ESC 조건의 GEP 데이터를 가지고 여러 공동발현 측정법을 비교 한 결과, Human 에서는 Quantile 방법과 MIk 방법이, Mouse에서는 추가 정규화하 지 않은 MIk 방법이 가장 유의하게 나타났다. 유의한 전체 방법을 비교한 결과 Boundary 정규화 방법과 상호정보량에 기반한 공동발현 측정법 (MI, MIk)이 ESC-특이적 PPIs 를 예측하는데 적합하다고 제안할 수 있다.
본 논문에서는 조건 특이적인 PPIs 데이터를 구축한 후 GEP 데이터를 이 용한 조건 특이적인 PPIs 예측할 수 있는 방법을 종합적으로 비교분석하였다. General PPIs와 발현 연관성에서 유의한 차이가 있는 ESC-특이적 PPIs는 배아줄기 세포의 고유기능을 이해하는데 참고할 수 있으며, 구축한 조건 특이적인 PPIs 데 이터는 여러 세포 조건에서 새로운 PPIs를 예측하기 위한 중요한 참고자료로써 활용될 수 있다.
핵심어 : 단백질-단백질 상호작용, 조건 특이적인 단백질 상호작용, 유전자 발현 프로파일, 유전자 공동발현 패턴, 상호정보량, 정규화 방법, 배아줄기세포
차 례
국문요약 ··· ⅰ 차례 ··· ⅳ 그림 차례 ··· ⅵ 표 차례 ··· ⅶ 약어 ··· ⅷ Ⅰ. 서론 ··· 1 A. 연구배경 ··· 1 B. 관련 연구 ··· 3 C. 연구목적 ··· 6 Ⅱ. 연구대상 및 방법 ··· 8 A. 유전자 발현 프로파일 데이터 ··· 8 B. 단백질-단백질 상호작용 데이터 ··· 10 C. 조건 특이적인 단백질-단백질 상호작용 데이터 구축 및 검증 ··· 12 D. 유전자 공동발현 연관성 측정법 ··· 13 E. 조건 특이적인 단백질 상호작용 예측을 위한 유전자 공동발현 연관성 측정법 비교 ··· 17 Ⅲ. 결과 ··· 19 A. 조건 특이적인 단백질 상호작용 데이터 구축 및 검증 결과 ··· 19 B. 유전자 공동발현 연관성 측정법 비교결과 ··· 32 Ⅳ. 고찰 ··· 36Ⅴ. 결론 ··· 39 참고문헌 ··· 40 ABSTRACT ··· 44
그림 차례
Fig. 1. Methods Overview ··· 7
Fig. 2. DNA Microarray experiment and gene expression profiles data ··· 9
Fig. 3. Procedure for quantile normalization method ··· 10
Fig. 4. Protein-protein interactions in cellular signalling pathway ··· 11
Fig. 5. Gene co-expression measures of known condition-specific PPIs ··· 14
Fig. 6. Comparison of co-expression measures in predicting condition-specific PPIs · 18 Fig. 7. Integrated results of public PPI databases ··· 19
Fig. 8. Results of literature-curated condition-specific PPIs ··· 20
Fig. 9. Network of human stem cells PPIs ··· 22
Fig. 10. Network of mouse stem cells PPIs··· 23
Fig. 11. Mouse POU5F1 & NANOG network ··· 23
Fig. 12. Common PPIs across different conditions of stem cells and cancers ··· 24
Fig. 13. Comparison of normalization and co-expression measures in predicting ESC-specific PPIs ··· 33
Fig. 14. Results of methods showing ESC-specific PPIs of significant difference to general PPIs ··· 34
Fig. 15. Distribution of the most significant ESC-specific PPIs ··· 34
표 차례
Table 1. Statistics of species in stem cells-specific PPIs ··· 21
Table 2. GO enrichment test of stem cells using protein set ··· 25
Table 3. GO enrichment test of stem cells using protein interactions ··· 25
Table 4. Result of full GO enrichment test of mouse stem cells using protein set ··· 26
Table 5. Result of full GO enrichment test of human stem cells using protein set ··· 28
약 어
PPIs: Protein-protein interactions GEP: Gene expression profiles ESC: Embryonic stem cell GO: Gene Ontology
I. 서 론
A. 연구배경
단백질-단백질 상호작용(PPIs, protein-protein interactions)은 세포 내에서 다양한 신호전달 경로 및 대사, 전사 네트워크를 매개하는 기능적인 핵심 단위이다. 세포의 외부자극 및 환경에 따른 반응은 세포 내부의 PPIs 의 변화를 통해 일어나며, PPI 의 변화는 단백질 기능의 차이를 만들어 낸다. 예를 들어, TBL1X 단백질은 상호작용하는 단백질 파트너에 따라서 다른 복합체를 형성하게 되고, 그에 따른 억제자(Repressor) 또는 활성자(Activator) 역할을 달리 하게 된다 (Perissi 등, 2004; Wang 등, 2011). 따라서 특정 세포의 PPIs 네트워크는 해당 세포 및 단백질의 동적인 기능을 이해하는데 포괄적인 실마리를 제공할 수 있다 (Lee 등, 2013; Przytycka 등, 2010).
오늘날의 Yeast two-hybrid, Mass spectrometry 등의 대용량 실험기술의 발달은 세포에서 생체 내(In vivo) 전체 단백체 수준(Proteome-wide)의 PPIs 네트워크를 동정할 수 있는 가능성을 가져왔다 (Ewing 등, 2007; Rual 등, 2005). 하지만 여전히 실험적으로 모든 단백질 간 물리적 상호작용을 동정하기에는 많은 비용과 시간이 소요되며, 따라서 현재 특정 세포에서 중요한 기능을 한다고 예상되는 일부 단백질을 위주로 실험이 진행하고 있다 (Sung 등, 2013). 하지만 보다 종합적인 이해를 위해서는 특정 세포조건에 활성화된 전체 단백질 상호작용 네트워크를 알 필요가 있으며 이를 위해서 전산적인 예측이 보조적인 정보를 제공할 수 있다 (Shoemaker 와 Panchenko, 2007; Skrabanek 등, 2008).
위와 같은 대용량 실험데이터를 포함하는 PPI 데이터는 현재 각 실험데이터가 보고된 문헌에서 직접 큐레이션한 결과가 여러 데이터베이스를 통해 공유되고 있으며, 이 데이터를 이용하여 다양한 기능유전체학적 연구가 진행되고 있다 (Bader 등, 2008). 하지만 공개된 대부분의 PPI 데이터베이스에는 특정 PPI 의 세포조건에 대한 주석정보가 부재하며, 이는 실제 실험한 세포의 다양한 조건들이 혼재되어 있거나 생체 밖 (In vitro)의 데이터도 포함되어 있다 (Bossi 와 Lehner, 2009). 위와 같은 데이터는 전체 PPI 네트워크의 특성 또는 특정 생물종에서 기능적인 정보를 제공할 수 있으나, 특정 세포조건에서의 동적인 기능을 이해하는데에는 제한점이 될 수 있다. 보다 엄밀한 분자생물학적 메커니즘 연구를 위해서는 PPI 의 동적인 기능이 고려된 세포조건 정보가 필요하다 (Ideker 와 Krogan, 2012).
본 연구에서는 PPI 데이터를 세포조건의 유무에 따라 다음과 같이 구분하였다: General PPI, 세포조건이 없는 PPI; Condition-specific PPI, 세포조건이 알려진 PPI.
현재까지의 주된 관심은 전체 단백질 사이에서 상호작용을 보이는 일부 단백질쌍을 찾아내는 것이다. 이어 상호작용하는 전체 단백질쌍 중에서 일부만이 특정 세포조건에서 활성화를 보이며 그러한 활성화가 곧 해당 세포의 특유한 기능을 나타낸다. 이러한 세포조건이 알려진 PPI 데이터 (Condition-specific PPIs)는 세포조건이 알려지지 않은 General PPI 데이터에 비교하여 데이터량이 부족하며, 대용량 실험방법 중 일부는 실제 세포환경을 완벽히 반영하는데 한계를 보인다. 따라서 다양한 전산학적 예측연구가 필요한 실정이다 (Yousefi 등, 2012).
한편, 기능유전체학의 중요한 데이터인 유전자 (또는 전사체, mRNA) 발현 프로파일 (GEP, gene expression profile) 은 한번에 특정 세포 조건 내의 모든 유전자의 발현량을 측정할 수 있어서 유용한 자료원이 될 수 있다 (Gitter 등, 2013). 실제로 NCBI GEO 데이터베이스에는 여러 생물종에 걸쳐 다양한 세포 및 자극에 따른 GEP 데이터를 공개하고 있으며, 이를 활용한 다양한 유전체 수준의 기능 연구가 가능하다 (Barrett 등, 2011; Barrett 등, 2013) . 그 중 하나로 GEP 데이터 상의 모든 유전자 간의 공동 발현패턴 (Gene co-expression pattern)을 분석함으로 이를 해당 세포 조건의 기능분석에 적용시킨 연구가 다수 진행되어 왔다 (Lee 등, 2004; Torkamani 등, 2010). 특히 PPI 로 알려진 두 유전자 사이의 발현패턴의 상관성 (correlation)이 임의의 유전자 쌍에 비해 높은 것으로 보고되고 있고 있으며, 이를 근거로 상관성이 높은 유전자 쌍일수록 PPI 가 존재할 가능성이 높다고 가정할 수 있다 (Jansen 등, 2002; von Mering 등, 2002).
B. 관련 연구 유전자 공동발현과 PPI 사이의 관계에 대한 연구는 마이크로어레이 기술이 개발된 이래부터 많이 있어왔으며, 실제로 공동 발현패턴 및 네트워크를 기반한 기능적 분석이 특정 세포의 생물학적 메커니즘을 이해하는데 유효함을 보여주고 있다 (Cai 등, 2010; Torkamani 등, 2010). 하지만 유전자 공동 발현패턴에 기반한 PPI 예측시도의 대부분은 GEP 데이터의 세포조건과 무관한 물리화학적인 단백질 상호작용의 가능유무만을 확인해왔다 (Xulvi-Brunet 와 Li, 2010). 조건 특이적인 PPI 를 예측하기가 어려운 이유는 예측결과를 평가할 수 있는 충분한 정답셋 PPI 가 부재하기 때문이다. 따라서, 특정 세포조건에서의 PPI 를 예측하기 보다는 기능적으로 유의한
공동발현 네트워크를 찾아낸 후 특정 질병이나 매커니즘의 핵심적인 인자나 특정 신호경로를 발굴하는데 활용하는 방식으로 기존 연구가 진행되어 왔다 (Zhao 등, 2010).
GEP 데이터의 세포조건과 무관한 General PPI 를 예측하는 연구에서는 주로 상관계수 기반 측정법 (Correlation coefficient)을 이용하여 공동발현 패턴을 측정하였다. PPI 는 보다 구체적으로 단백질 복합체 (Protein complex)의
일부이거나 효소반응의 참여자일 수 있으며, General PPI 를 이용한
공동발현정도를 측정한 결과, 단백질 복합체에서 가장 높은 상관성을 나타내었다 (Das 등, 2012; Jansen 등, 2002; Sprinzak 등, 2006). 단백질 복합체에 참여하는 PPI 는 효소반응의 PPI 보다 강한 결합상태를 가지며 따라서 진화적으로 보존된 것으로 알려져 있다 (Brown 와 Jurisica, 2007). 이러한 PPI 사이의 발현양 역시 효소반응과 같은 일시적인 상호결합이나 무작위 단백질쌍에 비교했을 때 상관성이 더 높게 나타난 것이다. 하지만, 효소반응과 같은 약한 상호결합력을 가지는 PPI 의 발현양은 임의 유전자쌍과 비교해서 상관성에 큰 차이가 없는 것으로 나타났으며, GEP 데이터 상에서 전체 유전자쌍 사이의 상관성을 계산한 결과 높은 상관성을 가지는 유전자쌍이라 해도 General PPI 에 해당하지 않거나 임의 유전자쌍과 유의한 차이가 없다고 보고된 바 있다 (Bhardwaj 와 Lu, 2005; Xulvi-Brunet 와 Li, 2010). 한편, 물리적인 PPI 네트워크 대신 기능성 유전자 네트워크를 예측한 연구도 많이 수행되었다. 이 중 대부분은 GEP 데이터의 세포조건을 고려하여 해당 세포의 분자생물학적 연구를 하는데 응용한 것으로, 새로운 단백질의 기능이나 전사인자의 표적유전자를 예측할 수 있다. 이와 같은 기능성 네트워크의 구축을 위해 사용되는 공동발현 측정법은 상관계수 방법 외에도
상호정보량 (Mutual information)을 기반한 측정법이 다수 존재한다. 대표적으로 B 세포의 GEP 데이터를 이용한 B-cell interactome (BCI) 은 특정 단백질이 B 세포의 특정 조건에서 어떤 새로운 기능을 수행하는지를 예측해내었으며 이를 실험적으로 검증하였다 (Lefebvre 등, 2010; Mani 등, 2008; Wang 등, 2009). 이 외에도 A.thaliana, C. elegans, M. musculus 에서 각 조직별 (Tissue-specific)로 다른 유전자 네트워크를 구축하고 해당 조직에서 중요한 유전자를 중심으로 새로운 기능을 예측하고 그 결과를 Gene Ontology 및 실험으로 검증한 바 있다 (Chikina 등, 2009; Guan 등, 2012; Pop 등, 2010). 이와 비슷한 목적으로 특정 질병이나 세포에서 알려지지 않은 중요한 인자를 찾는 방법으로 General PPI 데이터와 GEP 데이터만을 통합함으로써 기능적으로 유의한 단백질 모듈 (Module)을 찾을 수도 있다 (Lin 등, 2010; Yoon 등, 2011).
위에 언급한 연구들의 한계점은 ⅰ) General PPI 를 특정 세포조건의 GEP 데이터의 비교함으로 PPI 의 동적성 (Dynamics)를 반영하지 못한 점과 ⅱ)
GEP 데이터의 세포조건을 반영한 기능적 네트워크를 예측하였으나 이를
해당조건의 물리적인 PPI 와 비교검증하지 못한 점을 들 수 있다. 본 연구에서는 이러한 한계점을 극복하고자 GEP 데이터와 같은 세포조건의 PPI 데이터를 줄기세포 (Stem cell)와 종양세포 (Cancer)에 걸쳐 구축하여, 여러 유전자 공동발현 측정법을 비교하였다. 공동발현 패턴은 주로 상관계수나 상호정보량을 기반으로 측정되며, 이들 사이의 비교연구는 있었으나 비교기준이 각 연구마다 상이하며 특히 Condition-specific PPI 를 이용한 비교는 존재하지 않았다 (Priness 등, 2007; Song 등, 2012).
C. 연구목적
본 연구에서는 Condition-specific PPIs 를 예측하는데 응용할 수 있는 유전 자 공동발현 측정법을 비교하기 위해 아래 2가지 전략을 시도했다. 먼저, Condition-specific PPIs를 구축하기 위해 General PPIs 중에서 줄기세포와 종양세포 관련 논문을 큐레이션하였다. 다음으로 GEP데이터를 여러 정규화 방법으로 전처 리하고, 이를 주요 상관계수 및 상호정보량 측정법으로 공동발현 정도를 계산하 였다. 방법을 비교하는 기준으로는 Condition-specific PPIs 및 General PPIs 데이터 에서 각각 공동발현 정도를 계산하고, 이 결과의 차이를 이용하였다.
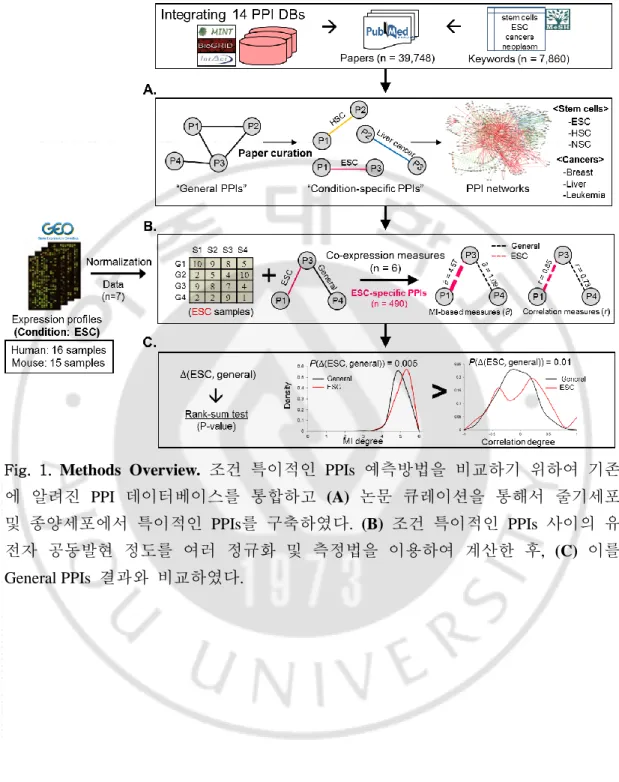
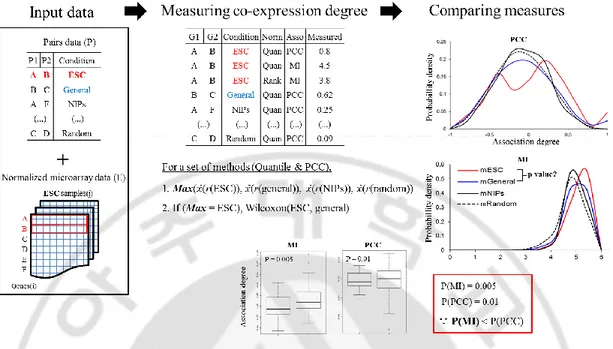
Fig. 1. Methods Overview. 조건 특이적인 PPIs 예측방법을 비교하기 위하여 기존 에 알려진 PPI 데이터베이스를 통합하고 (A) 논문 큐레이션을 통해서 줄기세포 및 종양세포에서 특이적인 PPIs를 구축하였다. (B) 조건 특이적인 PPIs 사이의 유 전자 공동발현 정도를 여러 정규화 및 측정법을 이용하여 계산한 후, (C) 이를 General PPIs 결과와 비교하였다.
II. 연구대상 및 방법
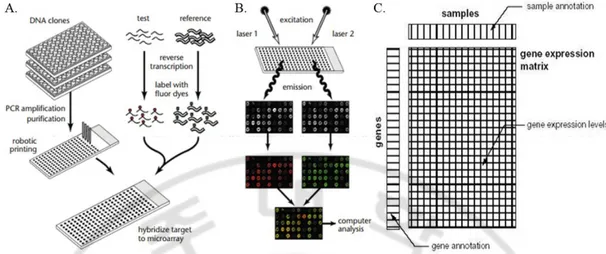
A. 유전자 발현 프로파일 데이터 유전자 발현 프로파일 데이터는 DNA 마이크로어레이 (Microarray) 칩 기술을 통해 주로 얻어지며, 한번에 관심있는 샘플에서 수만개 유전자의 발현량을 동시에 측정할 수 있는 대용량 빅데이터이다. 이는 아직 기능이 알려지지 않은 유전자를 세포시스템 맥락에서 규명하거나 특정 세포의 기능을 정확히 이해하기 위해 효과적인 정보를 제공할 수 있다. Fig. 2 는 실제 마이크로어레이 실험을 통한 한 세포의 전체 유전자 발현양을 측정하는 방법을 나타내며 동시에 전산학적 분석의 대상인 최종 데이터 구조를 보여주고 있다. 본 연구에서는 배아줄기세포 (ESC, embryonic stem cell)의 PPIs 를 기준으로 여러 방법을 비교하기 위해 Mouse 와 Human 에서 각각 1 개씩의 ESC 조건 GEP 데이터를 선택하였다. 해당 데이터는 NCBI GEO 에서 가져왔으며, 해당 ID 는 다음과 같다: GSE22651, GSE9563.GEO에서 내려받은 두 GEP 데이터는 정규화, ID 변경 등의 전처리를 실 시하였다. Probe ID는 NCBI Gene Symbol 로 변경하였고, 하나의 유전자에 여러 probe 가 맵핑 시 중앙값으로 그 발현값을 변경하였다. GEP 데이터를 정규화 방 법 (Normalization)에 따른 분석결과의 차이를 비교하기 위하여 원 데이터 형태와 추가적인 6가지 정규화 방법으로 전처리한 데이터를 만들었다.
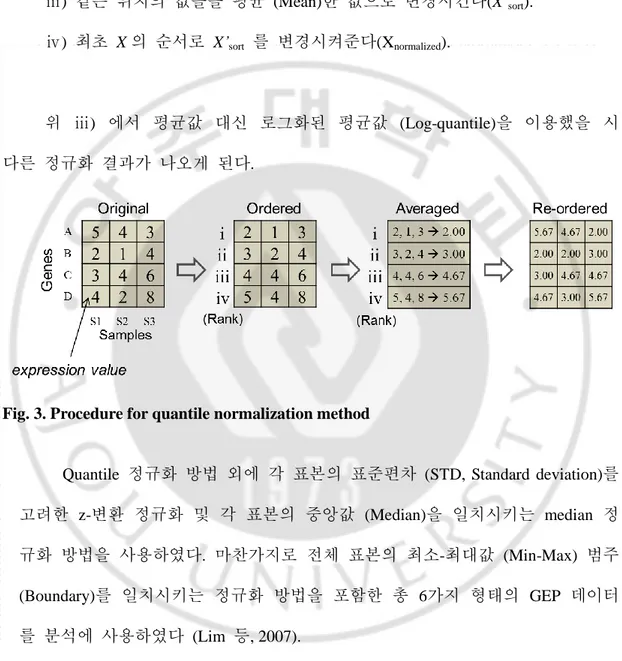
Fig. 2. DNA Microarray experiment and gene expression profiles data. (A) 마이크로어 레이 위에 알려진 유전자의 탐지자를 고정하고 샘플로부터 RNA를 추출한 후 형 광처리하여 마이크로어레이에 혼합하는 과정. (B) 마이크로어레이를 이미지로 스 캔하여 형광의 정도를 측정하여 수치로 변경시키는 과정. (C) 매트릭스 형태로 변 환된 최종 GEP 데이터 구조. 그림출처 (Duggan 등, 1999) 마이크로어레이 실험은 많은 양의 탐지자 (Probe)와 표본 (Samples)을 대 상으로 대량의 데이터를 얻는 과정이므로 동일한 표본이라 할지라도 반복에 따 라서 데이터의 오차가 발생할 수 있다. 이러한 오차는 실제 통계적 검정력을 약 화시킬 수 있는 요인이 될 수 있으므로 오차를 제거하여 추후 분석의 왜곡을 최 소화시켜야 한다 (Barrett 등, 2011). 본 연구에서는 이러한 표본 간 오차 (Between-sample error)에 대한 정규화를 실시하였으며, Quantile 정규화 방법을 포함한 다른 방법을 적용하여 표본 간의 범위를 일치시키거나 분포를 유사하게 만들었다. 아 래는 Quantile 정규화 방법에 대한 간략한 설명이다 (Bolstad 등, 2003). Quantile 방법이란 데이터를 오름차순으로 정렬하였을 시 같은 위치에 해 당하는 것을 말하며, 이 때의 데이터의 값을 평균값으로 일치시키는 것이다 (Fig. 2). 본 연구에서는 총 3가지 방법으로 Quantile 정규화를 실시하였고 아래는 해당 절차이다.
ⅰ) n 개 표본의 데이터(X)를 모두 오름차순으로 정렬한다. ⅱ) 정렬된 데이터(Xsort)의 각 i 번째 값을 n 차원 상에 위치시킨다. ⅲ) 같은 위치의 값들을 평균 (Mean)한 값으로 변경시킨다(X’sort). ⅳ) 최초 X 의 순서로 X’sort 를 변경시켜준다(Xnormalized). 위 ⅲ) 에서 평균값 대신 로그화된 평균값 (Log-quantile)을 이용했을 시 다른 정규화 결과가 나오게 된다.
Fig. 3. Procedure for quantile normalization method
Quantile 정규화 방법 외에 각 표본의 표준편차 (STD, Standard deviation)를 고려한 z-변환 정규화 및 각 표본의 중앙값 (Median)을 일치시키는 median 정 규화 방법을 사용하였다. 마찬가지로 전체 표본의 최소-최대값 (Min-Max) 범주 (Boundary)를 일치시키는 정규화 방법을 포함한 총 6가지 형태의 GEP 데이터 를 분석에 사용하였다 (Lim 등, 2007).
B. 단백질-단백질 상호작용 데이터
단백질-단백질 상호작용 (PPIs, protein-protein interactions) 데이터는 실제 세포 생체 내의 다양한 네트워크를 실험적으로 동정한 것으로, 이를 통해 새로운
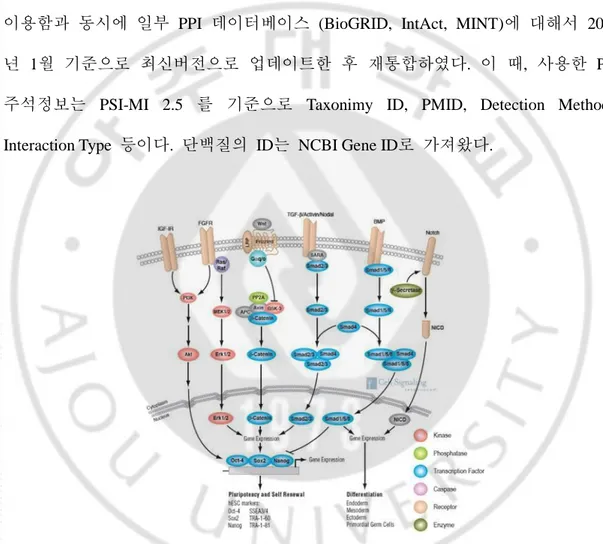
생체 네트워크를 발견하고 기능적인 분석을 실시할 수 있다 (Fig. 3). 이러한 PPI 데이터는 개별 문헌에서 보고된 결과를 직접 큐레이션하여 여러 데이터베이스를 통해 제공되고 있으며, 여러 PPI 데이터베이스를 통합한 형태로 제공되기도 한다 (Turner 등, 2010). 본 연구에서는 그러한 통합된 PPI 데이터베이스 (iRefWeb 4.0)를 이용함과 동시에 일부 PPI 데이터베이스 (BioGRID, IntAct, MINT)에 대해서 2012 년 1월 기준으로 최신버전으로 업데이트한 후 재통합하였다. 이 때, 사용한 PPI 주석정보는 PSI-MI 2.5 를 기준으로 Taxonimy ID, PMID, Detection Methods, Interaction Type 등이다. 단백질의 ID는 NCBI Gene ID로 가져왔다.
Fig. 4. Protein-protein interactions in cellular signalling pathway. 배아줄기세포의 만 능성과 분화에 관련된 신호전달경로는 다양한 단백질 간 상호작용으로 구성된다 (그림출처: CST, Inc)
C. 조건 특이적인 단백질-단백질 상호작용 데이터 구축 및 검증
통합된 PPI 데이터베이스는 이후 고유한 PMID를 가져와서 해당 논문의 초록을 관심 조건에 해당하는 키워드를 통해 검색하였다. 전체 논문의 초록은 MEDLINE의 2011년 11월 자 데이터를 이용하였다 (Lin와 Wilbur, 2007). 관심조건 은 줄기세포(Stem cell)와 종양세포(Cancer)였으며, 해당 키워드 정보는 MeSH 와 Cell Ontology 데이터베이스에서 가져왔다 (Bard 등, 2005; Lipscomb, 2000). 이렇게 검색된 논문은 직접 큐레이션을 통해 세포조건 정보를 맵핑하였으며, 첫째, 초록 수준에서의 큐레이션 둘째, 본문 수준에서의 큐레이션을 실시하였다. 종양세포의 경우, 검색된 논문 수가 많아서 초록 수준까지만 큐레이션을 실시하였으며, 줄기 세포의 경우, 본문 수준까지 상세한 큐레이션을 실시하였다. 특정 세포조건이라 하더라도 PPI 실험에 사용한 세포는 다양할 수 있으며, 그러한 세부적인 세포주 (cell line) 정보까지 큐레이션의 대상으로 포함시켰다. 종양세포와 줄기세포 모두 세부종류에 따라 큐레이션을 실시하였다. 해당 큐레이션 결과를 기존의 알려진 PPI 데이터로 맵핑하여 최종적인 조건 특이적인 PPI 데이터를 구축하였다. 구축한 조건 특이적인 PPI의 검증을 위해서 다음 2가지 방법을 이용하였 다. 첫째, 특정 세포조건에 해당하는 PPIs를 구성하는 단백질들을 상호작용 정보 를 생략한 채 단백질 수준에서 Gene Ontology 분석을 실시하였다 (Ashburner 등, 2000). 둘째, PPIs 자체에 의미가 있다고 간주하여 Network Ontology 분석을 실시 하였다 (Wang 등, 2011). 추가적으로 전체 조건 특이적인 PPI 데이터를 이용하여 조건 사이의 공유되는 PPI를 확인하고 공유되는 PPI의 개수에 따른 유의성 검증 을 실시하였다. 유의성 검증은 임의 순열 (Random permutation)을 이용하여 실시 하였으며, 공유하는 PPI가 없는 경우에 정규분포를 가정하여 p-value 를 계산하였 다. 해당 유의값을 가지고 전체 조건 사이의 계층 군집화 (Hierarchical clustering)
를 실시하여 줄기세포 및 종양세포의 여러 조건 사이의 유사성을 확인하였다. 또 한, 연관성 측정 시 비교대상으로 상호작용하지 않는 PPI 데이터 (NIPs, non-interacting protein pairs)를 Negatome 데이터베이스에서 가져왔다 (Smialowski 등, 2010).
D. 유전자 공동발현 연관성 측정법
유전자 공동발현 연관성 측정법은 모두 6 가지 방법을 이용하였으며, 크게 상관성 (Correlation) 및 상호정보량 (MI, mutual information)에 기반하는 방법 두 가지로 나뉠 수 있다 (Song 등, 2012). 상호정보량에 기반하는 방법은 최대정보계수 (MIC, maximal information coefficient), 상보정보량 (MI), 상호정보량-커널 (MIk, MI by kernel density estimator)이며, 상관성에 기반한 방법은 피어슨 상관계수 (Pearson correlation coefficient), 스피어맨 상관계수 (Spearman correlation coefficient), 코사인 상관계수(Cosine correlation)를 사용하였다 (Reshef 등, 2011).
Fig. 5. Gene co-expression measures of known condition-specific PPIs. 상관계수에 기반한 방법과 상호정보량에 기반한 분석 각각 3 개, 총 6 개를 이용하여 유전자 공동발현 연관성을 측정하였다. 1. 피어슨 상관계수 상관계수분석이란 변수간의 관련성을 분석하기 위해 사용하는 방법으로서, 하나의 변수가 다른 변수와 관련성이 있는지를 알 수 있고, 또 관련 정도가 어느 정도인지 알아보기 위한 방법이다. 피어슨 상관계수는 상관계수분석에서 자주 이용되는 계수이며 상관계수 r 은 [-1, 1]의 값을 갖는다. r 의 값이 1 에 가까울수록 두 변수는 양의 상관관계를 나타내게 되고, 서로 유사하다는 것을 의미한다. 반면 r 이 -1 에 가깝다면 두 변수의 관계는 음의 상관관계가 되며 서로 반대방향으로 관계가 있다는 것을 의미한다. r 이 0 에 가깝다면 두 변수 사이에 별로 관계가 없음을 의미한다. N 개의 원소를 갖는 두 벡터 X 와 Y 사이의 피어슨 상관계수는 다음과 같이 정의된다.
)
)
(
)(
)
(
(
2 2 2 2N
Y
Y
N
X
X
N
Y
X
XY
r
pearson
2. 스피어맨 상관계수 비모수분석은 변수들이 양적 변수가 아니어도 될 때 이용할 수 있는 상관계수분석 방법으로, 스피어맨 상관계수와 같은 방법들이 있다. 스피어맨 상관계수는 변수의 순위배열을 사용하여 변수간의 상관관계를 분석하는 방법으로 피어슨 상관계수와 마찬가지로 상관계수는 [-1, 1]의 값을 갖는다. 한편 스피어맨 상관계수는 X 와 Y 의 순위배열 Dx 와 Dy 를 사용하여 다음 식으로 나타낼 수 있다.)
1
(
)
(
6
1
2 2
N
N
Dy
Dx
r
spearman 3. 코사인 계수 두 변수간의 유사성 측정을 위한 다른 방법으로 코사인계수가 있다. 코사인계수 방법에서 계수값은 [-1, 1]의 범위를 갖게 되는데, 두 변수간의 유사성은 계수값이 클수록 높게 된다. 왜냐하면 코사인계수는 두 변수 사이의 각을 측정해서 코사인 값으로 나타내어 주는데 유사성이 높을수록 각이 작고, 코사인값은 1 에 가까워지기 때문이다. X 와 Y 의 코사인 계수는 다음 식에 의하여 구할 수 있다. 2 2 cosY
X
XY
r
ine
4. 상호정보량 상호정보량이란 한 랜덤 변수가 다른 랜덤 변수에 대해 담고 있는 정보량을 의미하며, 어떤 확률 분포에 대해 엔트로피 (Entropy)라고 불리는 양을 통해서 측정할 수 있다. 이 엔트로피는 랜덤 변수의 불확실성에 대한 척도이며, 상호정보량(I)은 한 랜덤 변수(X)에 대한 지식으로 인해 다른 랜덤 변수(Y)에 대한 불확실성이 줄어드는 정도를 의미한다. GEP 데이터에서의 두 유전자의 발현값의 패턴 역시 같은 맥락에서 연관성으로 측정할 수 있다. 상호정보량은 „0‟부터 양의 정수값을 가질 수 있으며, 0 에 가까울수록 두 변수사이의 독립성이 큰 정수값을 가질수록 종속성이 커진다. 5. 커널상호정보량
본 방법은 연속형 변수를 k 개로 분류한 후, Kernel density estimator 를 이용하여 확률밀도함수를 추정한 후 적분된 값에 대하여 상호정보량을 계산하게 된다. 6. 최대정보계수 이 방법은 두 랜덤 변수로 구성된 데이터가 있을 때 각각의 변수에 대하여 적당한 grid 형태로 구간을 나누어서 정보량이 최대가 되게 하는 지점을 찾는 것을 나타낸다. 이 방법은 선형 및 비선형 데이터 모두에서 숨겨진 관계를
찾아내는데 일관적인 결과를 나타내었으며, 여러 형태의 데이터에 걸쳐 사용될 수 있다.
E. 조건 특이적인 단백질 상호작용 예측을 위한 유전자 공동발현 연관성 측정법 비교
총 6 가지 방법에 기반한 유전자 공동발현 연관성 측정은 큐레이션한 배아줄기세포 (ESC, embryonic stem cell)에서 특이적인 PPIs 데이터를 대상으로 하였다. 즉, Fig. 6 과 같이 준비된 다양한 Pairs 데이터 사이의 연관성을 GEP 에서 측정하고 그 결과를 비교하였다. 먼저 ESC-specific PPI 연관성 결과가 가장 높게 나온 결과만을 선택하여 조건특이성을 보이지 않는 방법은 고려하지 않았다. 이어 ESC-specific PPI 연관성 결과를 세포조건이 알려지지 않은 단백질 상호작용 (General PPI)의 결과와 비교함으로써 실제로 조건 특이적인 PPI 를 얼마나 더 예측할 수 있는지를 확인하였다. 두 분포의 차이가 유의한지를 확인하기 위해서 Wilcoxon signed-rank 검사를 실시하였고 해당 결과 나오는 P-value 값으로 여러 연관성 방법 및 정규화 방법을 비교하였다.
Fig. 6. Comparison of co-expression measures in predicting condition-specific PPIs. 배아줄기세포(ESC)의 GEP 데이터를 여러 정규화 방법으로 전처리한 후,
ESC-specific PPIs 로 여러 연관성 측정법으로 계산을 한다. 모든 PPIs 사이의
III. 결과
A. 조건 특이적인 단백질-단백질 상호작용 데이터 구축 및 검증 결과
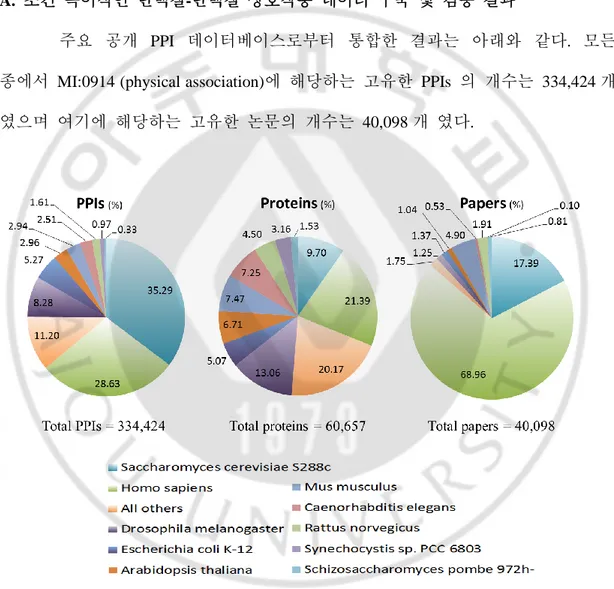
주요 공개 PPI 데이터베이스로부터 통합한 결과는 아래와 같다. 모든 종에서 MI:0914 (physical association)에 해당하는 고유한 PPIs 의 개수는 334,424 개 였으며 여기에 해당하는 고유한 논문의 개수는 40,098 개 였다.
Fig. 7. Integrated results of public PPI databases. 총 14 개의 PPI 데이터베이스를 통합한 결과를 전체 단백질 상호작용, 단백질, 논문 개수 별로 구분하였다. 색깔과 구획은 생물종의 비율에 해당한다.
아래 Fig. 8 은 줄기세포와 종양세포에서 조건특이적인 PPIs 를 큐레이션한 결과이다. 각 조건에 해당하는 개수는 고유한 개수이지만 다른 조건 사이에 공유되는 PPIs 가 존재할 수 있으며, 줄기세포 전체 종에서 큐레이션된 PPIs 의 개수는 2,441 개이며 종양세포에서는 모두 6,090 개의 PPIs 가 확인되었다. 종양세포의 경우, 데이터양의 방대함으로 인해 일부 주요 조건에 대한 큐레이션만 실시하였다. 종양세포는 큐레이션된 PPI 의 결과로써 최초 키워드를 통해 논문 초록을 검색한 후, 각 종양형태 별로 알려진 PPIs 의 개수에 따라 우선적으로 큐레이션을 실시하였다. 이때, PPIs 개수가 500 개 이상인 종양세포만 초록 큐레이션을 실시하였다
Fig. 8. Results of literature-curated condition-specific PPIs. 왼쪽 그림에는 큐레이션 중간 과정을 나타내었으며, 오른쪽에는 최종 큐레이션 결과를 나타내었다.
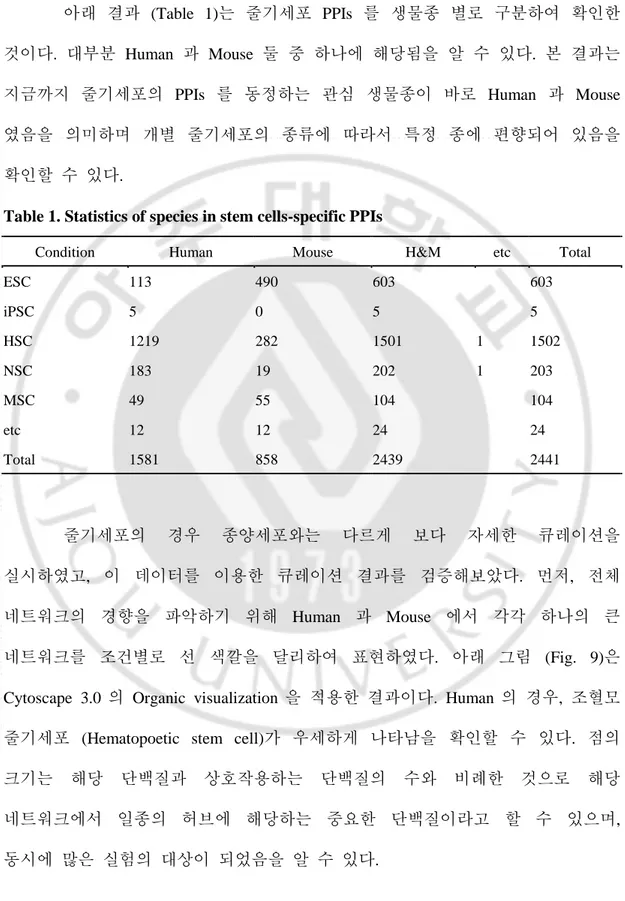
아래 결과 (Table 1)는 줄기세포 PPIs 를 생물종 별로 구분하여 확인한 것이다. 대부분 Human 과 Mouse 둘 중 하나에 해당됨을 알 수 있다. 본 결과는 지금까지 줄기세포의 PPIs 를 동정하는 관심 생물종이 바로 Human 과 Mouse 였음을 의미하며 개별 줄기세포의 종류에 따라서 특정 종에 편향되어 있음을 확인할 수 있다.
Table 1. Statistics of species in stem cells-specific PPIs
Condition Human Mouse H&M etc Total
ESC 113 490 603 603 iPSC 5 0 5 5 HSC 1219 282 1501 1 1502 NSC 183 19 202 1 203 MSC 49 55 104 104 etc 12 12 24 24 Total 1581 858 2439 2441 줄기세포의 경우 종양세포와는 다르게 보다 자세한 큐레이션을 실시하였고, 이 데이터를 이용한 큐레이션 결과를 검증해보았다. 먼저, 전체 네트워크의 경향을 파악하기 위해 Human 과 Mouse 에서 각각 하나의 큰 네트워크를 조건별로 선 색깔을 달리하여 표현하였다. 아래 그림 (Fig. 9)은 Cytoscape 3.0 의 Organic visualization 을 적용한 결과이다. Human 의 경우, 조혈모 줄기세포 (Hematopoetic stem cell)가 우세하게 나타남을 확인할 수 있다. 점의 크기는 해당 단백질과 상호작용하는 단백질의 수와 비례한 것으로 해당 네트워크에서 일종의 허브에 해당하는 중요한 단백질이라고 할 수 있으며, 동시에 많은 실험의 대상이 되었음을 알 수 있다.
Fig. 9. Network of human stem cells PPIs. 줄기세포 큐레이션 결과 중 하나의 네트 워크로 연결되는 PPIs로, 점은 단백질이며 선을 상호작용을 의미한다. 선 색깔은 해당 세포조건을 의미하며 점 크기는 이웃하는 단백질의 정도와 비례한다.
Fig. 10 은 Mouse의 줄기세포 네트워크를 나타낸 것으로, Human과 다르게 ESC의 PPIs가 많이 관찰됨을 알 수 있다. 또한, 잘 알려진 배아줄기세포의 만능 성 전사인자인 POU5F1이 허브로써 가장 많은 단백질과 상호작용하고 있음을 알 수 있다. 그와 상호작용하는 주요한 단백질들로 Sox2, Esrrb, Sall4 등이 있으며 마 찬가지로 많은 단백질 파트너를 공유하고 있음을 알 수 있다. 이어, POU5F1 및 NANOG 상호작용하는 모든 단백질들과 그들 사이에 존재하는 상호작용을 그림 (Fig. 11)으로 표현하였다. 이 네트워크는 실제로 생물학적인 의미를 함의하고 있 으며, 이 중 일부는 아직 알려지지 않은 중요한 메커니즘에 관여하고 있다고 가 정할 수 있다.
Fig. 10. Network of mouse stem cells PPIs. 줄기세포 큐레이션 결과 중 하나의 네트 워크로 연결되는 PPIs로, 점은 단백질이며 선을 상호작용을 의미한다. 선 색깔은 해당 세포조건을 의미하며 점 크기는 이웃하는 단백질의 정도와 비례한다.
Fig. 11. Mouse POU5F1 & NANOG network. ESC 만능성의 주요인자 중 하나인 POU5F1과 NANOG에 각각 상호작용하는 단백질과 그들 사이의 상호작용 네트워 크를 보여준다.
여러 조건들 사이의 PPI 공유정도를 확인한 결과는 아래 (Fig. 12)와 같으며, 해당 결과를 기반으로 군집화 분석을 실시한 결과, 줄기세포와 종양세포가 구분됨을 확인하였다. 이는 줄기세포와 종양세포간의 유사성에 대한 기존 연구에도 불구하고 여전히 차이가 있음을 나타낸다.
Fig. 12. Common PPIs across different conditions of stem cells and cancers. 초록색 레이블은 줄기세포이며 노란색은 종양세포에 해당하며, 매트릭스 내 값은 공유하는 PPI 의 개수이다. 매트릭스 배경색은 공유하는 PPIs 개수의 유의성에 해당된다. 이 유의성을 이용하여 계층군집화를 실시한 결과이다. 또한, GO 및 NOA 분석을 통해 본 큐레이션 결과가 실제 해당 기능을 나타내는지 확인하였다. Table 2 과 Table 3 과 같이, 해당 줄기세포에 밀접한 기능들을 가진 GO term 이 다수 나옴을 알 수 있고, 이를 근거로 본 큐레이션 작업이 생물학적으로 유의한 결과라고 할 수 있다.
Table 2. GO enrichment test of stem cells using protein set
Condition GO ID Enriched GO terms Count % P-value
mESC
GO:0001701 in utero embryonic development 20 7.94 7.92E-08 GO:0043009 chordate embryonic development 23 9.13 1.56E-06 GO:0009792
embryonic development ending in
birth or egg hatching 23 9.13 1.81E-06 GO:0048598 embryonic morphogenesis 13 5.16 0.015065
mHSC
GO:0048534
hemopoietic or lymphoid organ
development 12 6.22 7.83E-04
GO:0030097 hemopoiesis 11 5.7 0.001185
GO:0030099 myeloid cell differentiation 7 3.63 0.001247
mMSC
GO:0014031 mesenchymal cell development 4 9.09 3.77E-04 GO:0042692 muscle cell differentiation 5 11.36 4.13E-04 GO:0048762 mesenchymal cell differentiation 4 9.09 4.27E-04
hNSC
GO:0030182 neuron differentiation 22 19.3 6.08E-11 GO:0030900 forebrain development 14 12.28 3.54E-10 ※ In Condition column, “m” stands for mouse and “h” stands for human.
Table 3. GO enrichment test of stem cells using protein interactions
Condition GO ID Enriched GO terms Count % P-value
mESC
GO:0019827 stem cell maintenance 10 2.69 2.10E-05 GO:0045595 regulation of cell differentiation 24 6.45 7.70E-04 GO:0048856 anatomical structure development 85 22.85 0.0012 GO:0050793 regulation of developmental process 32 8.6 0.0033 GO:0009790 embryo development 18 4.84 0.0035 GO:0032502 developmental process 136 36.56 0.0162
GO:0048513 organ development 29 7.8 0.0245
GO:0048731 system development 17 4.57 0.0311 GO:0010605 negative regulation of macromolecule
metabolic process
70 18.82 0.0346
Table 4. Result of full GO enrichment test of mouse stem cells using protein set
Conditions
GO ID
and enriched GO terms Count % P-valuemESC
GO:0001701~in utero embryonic development 20 7.936508 7.92E-08 GO:0048864~stem cell development 7 2.777778 1.42E-06 GO:0043009~chordate embryonic development 23 9.126984 1.56E-06 GO:0009792~embryonic development ending in birth or
egg hatching
23 9.126984 1.81E-06
GO:0019827~stem cell maintenance 6 2.380952 2.34E-05 GO:0007548~sex differentiation 9 3.571429 0.001319 GO:0002520~immune system development 13 5.15873 0.003387 GO:0045165~cell fate commitment 8 3.174603 0.010482 GO:0001824~blastocyst development 5 1.984127 0.011591 GO:0008406~gonad development 6 2.380952 0.014575 GO:0048598~embryonic morphogenesis 13 5.15873 0.015065 GO:0035050~embryonic heart tube development 3 1.190476 0.02743
mHSC
GO:0048863~stem cell differentiation 5 2.590674 5.59E-04 GO:0048534~hemopoietic or lymphoid organ development 12 6.217617 7.83E-04
GO:0030097~hemopoiesis 11 5.699482 0.001185
GO:0030099~myeloid cell differentiation 7 3.626943 0.001247 GO:0045165~cell fate commitment 8 4.145078 0.002412 GO:0019827~stem cell maintenance 4 2.072539 0.002446 GO:0048864~stem cell development 4 2.072539 0.002788 GO:0045646~regulation of erythrocyte differentiation 3 1.554404 0.009352
GO:0001710~mesodermal cell fate commitment 2 1.036269 0.072355 GO:0040016~embryonic cleavage 2 1.036269 0.072355
mMSC
GO:0014031~mesenchymal cell development 4 9.090909 3.77E-04 GO:0042692~muscle cell differentiation 5 11.36364 4.13E-04 GO:0048762~mesenchymal cell differentiation 4 9.090909 4.27E-04 GO:0060485~mesenchyme development 4 9.090909 4.53E-04 GO:0045445~myoblast differentiation 3 6.818182 5.75E-04 GO:0051094~positive regulation of developmental process 4 9.090909 0.026476 GO:0051146~striated muscle cell differentiation 3 6.818182 0.029486 GO:0045843~negative regulation of striated muscle
development 2 4.545455 0.029777
GO:0048635~negative regulation of muscle development 2 4.545455 0.032707 GO:0045165~cell fate commitment 3 6.818182 0.072484 GO:0007492~endoderm development 2 4.545455 0.083989
Table 5. Result of full GO enrichment test of human stem cells using protein set
Conditions
GO ID
and enriched GO terms Count % P-valuehESC
GO:0045449~regulation of transcription 60 56.60377 6.23E-19
GO:0006350~transcription 44 41.50943 2.99E-11
GO:0045597~positive regulation of cell differentiation 10 9.433962 4.50E-05 GO:0060284~regulation of cell development 9 8.490566 1.28E-04
GO:0007369~gastrulation 6 5.660377 1.57E-04
GO:0009952~anterior/posterior pattern formation 7 6.603774 5.62E-04 GO:0045165~cell fate commitment 6 5.660377 0.0035203 GO:0001701~in utero embryonic development 6 5.660377 0.0094224 GO:0040014~regulation of multicellular organism growth 4 3.773585 0.0100679 GO:0043009~chordate embryonic development 8 7.54717 0.0107563 GO:0009792~embryonic development ending in birth or
egg hatching 8 7.54717 0.0112641 GO:0009798~axis specification 3 2.830189 0.0201871 GO:0007276~gamete generation 8 7.54717 0.0257551 GO:0001707~mesoderm formation 3 2.830189 0.0284217 GO:0048332~mesoderm morphogenesis 3 2.830189 0.0314182 GO:0048568~embryonic organ development 5 4.716981 0.0374391
hNSC
GO:0060284~regulation of cell development 17 14.91228 1.14E-11 GO:0045596~negative regulation of cell differentiation 17 14.91228 2.52E-11 GO:0030182~neuron differentiation 22 19.29825 6.08E-11 GO:0030900~forebrain development 14 12.2807 3.54E-10 GO:0021954~central nervous system neuron development 8 7.017544 6.26E-09 GO:0021953~central nervous system neuron
differentiation 8 7.017544 3.28E-08
GO:0042063~gliogenesis 9 7.894737 5.85E-08
GO:0051960~regulation of nervous system development 13 11.40351 6.06E-08 GO:0048667~cell morphogenesis involved in neuron
differentiation
13 11.40351 1.54E-07
hHSC
GO:0002520~immune system development 40 7.490637 2.55E-13 GO:0048534~hemopoietic or lymphoid organ
GO:0045646~regulation of erythrocyte differentiation 9 1.685393 9.03E-08 GO:0045639~positive regulation of myeloid cell
differentiation
11 2.059925 1.58E-07 GO:0045579~positive regulation of B cell differentiation 6 1.123596 1.22E-06 GO:0002763~positive regulation of myeloid leukocyte
differentiation 8 1.498127 1.79E-06
GO:0002521~leukocyte differentiation 18 3.370787 5.11E-06 GO:0035162~embryonic hemopoiesis 6 1.123596 6.89E-06 GO:0002761~regulation of myeloid leukocyte
differentiation
10 1.872659 1.26E-05 GO:0045577~regulation of B cell differentiation 7 1.310861 1.30E-05 GO:0030098~lymphocyte differentiation 14 2.621723 8.77E-05
hMSC
GO:0045669~positive regulation of osteoblast differentiation
10 19.23077 9.60E-17 GO:0001501~skeletal system development 17 32.69231 1.04E-14 GO:0045667~regulation of osteoblast differentiation 10 19.23077 2.53E-14 GO:0051216~cartilage development 11 21.15385 8.84E-14 GO:0030509~BMP signaling pathway 9 17.30769 2.61E-12 GO:0030278~regulation of ossification 10 19.23077 7.47E-12
GO:0001503~ossification 11 21.15385 8.24E-12
GO:0060348~bone development 11 21.15385 1.62E-11 GO:0001649~osteoblast differentiation 8 15.38462 1.28E-10 GO:0045165~cell fate commitment 10 19.23077 1.43E-09 GO:0051153~regulation of striated muscle cell
differentiation
6 11.53846 9.46E-08 GO:0002051~osteoblast fate commitment 4 7.692308 1.78E-07 GO:0051147~regulation of muscle cell differentiation 6 11.53846 2.65E-07
Table 6. Result of full GO enrichment test of stem cells using protein interactions
GO ID Enriched GO terms P-value R T G O
GO:0045596 negative regulation of cell differentiation 1.70E-06 1334 372 22 17 GO:0051093 negative regulation of developmental process 1.00E-05 1334 372 28 19 GO:0034645 cellular macromolecule biosynthetic process 2.10E-05 1334 372 678 223 GO:0009059 macromolecule biosynthetic process 2.10E-05 1334 372 678 223 GO:0019827 stem cell maintenance 2.10E-05 1334 372 11 10 GO:0090304 nucleic acid metabolic process 2.10E-05 1334 372 755 244 GO:0019219 regulation of nucleobase, nucleoside, nucleotide
and nucleic acid metabolic process
2.80E-05 1334 372 794 254
GO:0051171
regulation of nitrogen compound metabolic
process 2.80E-05 1334 372 794 254
GO:0010556 regulation of macromolecule biosynthetic process 3.50E-05 1334 372 788 252 GO:0006139
nucleobase, nucleoside, nucleotide and nucleic
acid metabolic process 3.80E-05 1334 372 759 244 GO:0044249 cellular biosynthetic process 3.90E-05 1334 372 686 224 GO:0009058 biosynthetic process 3.90E-05 1334 372 686 224 GO:0031326 regulation of cellular biosynthetic process 4.00E-05 1334 372 789 252 GO:0009889 regulation of biosynthetic process 4.00E-05 1334 372 789 252 GO:0010468 regulation of gene expression 5.00E-05 1334 372 798 254 GO:0034641 cellular nitrogen compound metabolic process 5.10E-05 1334 372 761 244 GO:0006807 nitrogen compound metabolic process 5.80E-05 1334 372 762 244 GO:0045449 regulation of transcription 6.60E-05 1334 372 774 247
GO:0006350 transcription 1.80E-04 1334 372 661 214
GO:0060255 regulation of macromolecule metabolic process 3.50E-04 1334 372 824 257 GO:0031323 regulation of cellular metabolic process 4.00E-04 1334 372 825 257 GO:0007399 nervous system development 4.40E-04 1334 372 29 17 GO:0080090 regulation of primary metabolic process 4.80E-04 1334 372 819 255 GO:0045595 regulation of cell differentiation 7.70E-04 1334 372 48 24 GO:0044260 cellular macromolecule metabolic process 0.001 1334 372 848 261 GO:0000122
negative regulation of transcription from RNA
polymerase II promoter 0.0011 1334 372 106 44
GO:0043170 macromolecule metabolic process 0.0011 1334 372 849 261 GO:0048856 anatomical structure development 0.0012 1334 372 234 85
GO:0019222 regulation of metabolic process 0.0019 1334 372 846 259 GO:0050789 regulation of biological process 0.0024 1334 372 908 275 GO:0048519 negative regulation of biological process 0.0032 1334 372 290 100 GO:0044237 cellular metabolic process 0.0032 1334 372 862 262 GO:0050793 regulation of developmental process 0.0033 1334 372 75 32
GO:0009790 embryo development 0.0035 1334 372 36 18
GO:0065007 biological regulation 0.0046 1334 372 914 275
GO:0044238 primary metabolic process 0.0048 1334 372 862 261
GO:0009987 cellular process 0.0049 1334 3721100 323
GO:0050794 regulation of cellular process 0.0052 1334 372 889 268
GO:0006396 RNA processing 0.0059 1334 372 4 4
GO:0008152 metabolic process 0.007 1334 372 877 264
GO:0048523 negative regulation of cellular process 0.011 1334 372 280 94
GO:0032502 developmental process 0.0162 1334 372 427 136
GO:0006355 regulation of transcription, DNA-dependent 0.0191 1334 372 377 121
GO:0045165 cell fate commitment 0.0197 1334 372 14 8
GO:0032526 response to retinoic acid 0.0206 1334 372 7 5
GO:0033189 response to vitamin A 0.0206 1334 372 7 5
GO:0033273 response to vitamin 0.0206 1334 372 7 5
GO:0051252 regulation of RNA metabolic process 0.021 1334 372 378 121 GO:0001710 mesodermal cell fate commitment 0.0215 1334 372 3 3
GO:0031047 gene silencing by RNA 0.0215 1334 372 3 3
GO:0060795
cell fate commitment involved in the formation of
primary germ layers 0.0215 1334 372 3 3
GO:0010629 negative regulation of gene expression 0.0237 1334 372 200 68
GO:0048513 organ development 0.0245 1334 372 75 29
GO:0048731 system development 0.0311 1334 372 40 17
GO:0010605
negative regulation of macromolecule metabolic
process 0.0346 1334 372 210 70
R: Number of genes in reference set; T: Number of genes in test set; G: Number of genes annotated by given term in reference set; O: Number of genes annotated by given term in test set
B. 유전자 공동발현 연관성 측정법 비교결과
Fig. 13 은 여러 공동발현 연관성 및 정규화 방법을 비교한 결과이다. 두 GEP 데이터를 분석한 결과, Mouse 에서는 정규화를 하지 않고 MIk 방법 (P = 9.1E-11)이, Human 에서는 Quantile 정규화 방법을 이용한 MIk 방법이 가장 뛰어난 성능 (P = 5.1E-03)을 보였다. 본 성능평가의 기준은 ESC PPIs 분포가 General PPIs 의 분포보다 더 연관성이 크게 나오는 동시에 두 분포 사이의 차이가 가장 큰 것이다. 흥미롭게도, Random pairs 와 비교한 결과에서도 해당 방법이 가장 유의한 차이를 나타냄을 보여준다. 이 외에도 Mouse 의 경우, Random pairs 와 비교 시 Boundary 의 MI (P = 3.1E-12)와 MIk (P = 1.3E-14) 가, Quantile 의 MIk (P = 7.8E-12)에서 유의한 차이가 있음을 보여주었다. General
pairs 와 비교 시에도 비슷한
결과를 나타내었다 (Boundary & MIk, P = 1.9E-10; Quantile & MIk, P = 5.5E-10).
Human 의 경우 역시, Quantile&MIk 조합 외에도 비슷한 성능의 방법들이 있었다. Random 에서는 Quantile&MI (P = 5.8E-06)와 Boundary&MIk (P = 9.9E-06), General 과 비교 시 Boundary&MIk (P = 2.9E-02)가 이에 해당한다.
실제 P-value 가 가장 낮은 분포 (Human 의 Quantile&MIk, Mouse 의 noNorm&MIk)를 보면 NIPs 데이터를 제외하면 ESC-SC-General-Random 순으로 오른쪽으로 취우침 (즉, 연관성이 높다)을 알 수 있다.
또한 연관성 측정법에 있어 어떤 정규화 방법을 이용하든지 모두 상호정보량에 기반한 방법의 성능이 우수함을 본 데이터를 통해 확인할 수 있다. 이어, ESC 를 Random 과 비교했을 시 보다 General 과 비교했을 시 관찰되는 유의한 차이개수가 적은 것으로 보아 (Mouse, 29 > 25; Human, 20 > 4), 조건 특이적인 PPIs (ESC)도 큰 맥락에서 General PPIs 의 일부임을 보여준다.
Fig. 13. Comparison of normalization and co-expression measures in predicting ESC-specific PPIs. ESC-ESC-specific PPIs 와 general PPIs 사이의 차이를 구하고 가장 유의한 결과에 p-value를 표시하였다. 표시된 noNorm&MIk (Mouse), Quantile&MIk (Human) 외에도 회색을 제외한 부분은 모두 유의한 차이 (P < 0.05)가 있는 결과에 해당한 다.
Fig. 14. Results of methods showing ESC-specific PPIs of significant difference to general PPIs. 정규화 방법에서는 Boundary, median, quantile 이, 공동발현 연관성 방법에서는 상호정보량에 기반한 방법이 유의한 결과를 많이 나타냈다.
Fig. 15. Distribution of the most significant ESC-specific PPIs. ESC PPIs (적색선)와 general PPIs (청색선) 사이의 차이가 가장 유의하게 나온 방법의 결과를 Human과 Mouse에서 각각 나타내었다.
Fig. 16. Distribution of widely significant ESC-specific PPIs. ESC PPIs (적색선)와 general PPIs (청색선) 사이의 차이가 가장 여러 번 유의하게 나온 방법의 결과를 Human과 Mouse에서 각각 나타내었다.
IV. 고찰
본 연구는 마이크로어레이 데이터를 이용한 조건 특이적인 PPIs 예측정도를 확인한 것으로, 이를 위해서 첫째, 조건 특이적인 PPIs 데이터를 구축하고 둘째, 다양한 마이크로어레이 전처리 방법과 유전자 공동발현 측정법을 포괄적으로 비교분석하였다. 실제, ESC 를 대상으로 조건 특이적인 PPIs 사이의 유전자 공동발현 정도가 General PPIs, random pairs 및 NIPs 보다 가장 높게 나왔으며, 이 결과는 Random 과 General PPIs 와 비교했을 시 유의한 차이가 있음을 보여주었다. 특히, ESC 를 Random 과 비교했을 시 General 보다 더 많은 방법조합 (정규화 및 연관성) 사이에서 유의함을 나타내었으며, 가장 유의한 P-value 를 나타낸 것도 random 과의 비교 (Mouse, 29 개 조합, P = 4.2E-15; Human, 20 개 조합, P = 2.7E-06)에서 였다.위와 같은 결과는 타당함과 동시에 여전히 General PPIs 와 조건 특이적인 PPIs 사이의 유사함을 의미하고 있으며, 이는 General PPIs 에 여러 조건 특이적인 PPIs 가 혼재되어 있음을 말해주고 있다. 실제로 본 연구에서 구축한 조건 특이적인 PPIs 데이터는 General PPIs 에서 유래한 일부에 해당한다.
연관성 측정법 및 정규화 방법을 비교한 결과는 흥미롭게도 MIk 방법이 가장 좋은 성능을 나타낸 공동발현 측정법 (Reshef 등, 2011; Song 등, 2012) 이었으며, Mouse 에 있어서는 추가적인 정규화를 하지 않는 raw 데이터에서 가장 좋은 성능을 보여주었다. 이는 본 연구에서 사용한 데이터가 이미 충분한 표본
간 오차가 교정되어있음을 의미한다. 이어 유의하게 나온 정규화 방법은 Quantile 과 Boundary 방법이며, MIk 외에 일반적인 상호정보량(MI) 연관성 방법이 좋은 성능을 나타내었다. 최근에 여러 종류의 데이터에 포괄적으로 적용하여 안정적인 성능을 나타낸 MIC 방법은 General 과의 비교에서 단 1 개의 방법조합 (Median, Human)에서만 유의하게 나왔다. 이는 데이터의 이질성 및 전처리 과정에서의 차이로 인한 결과로 판단된다. 비교대상으로 넣은 NIPs 데이터의 경우, 예상과는 다르게 데이터에 따라서 매우 불규칙한 분포를 나타내었다. Random 과 비슷하거나 좀더 연관성을 가질 것으로 예상하였으나 실제는 어떤 패턴도 파악할 수 없었다. 이는 NIPs 데이터의 특성 상 특정 세포조건만이 고려되었기 때문이라 생각되며, 특정 조건에서 상호작용이 없던 단백질 쌍이라도 다른 조건에서는 상호작용이 가능할 수 있는 네트워크 역동성 (Dynamics)가 반영된 결과라고 생각할 수 있다. 또한 조건 특이적인 PPIs 데이터 자체의 양이 여전히 부족하기 때문에 ESC 의 경우만 해도 Mouse (490 pairs)와 Human (113 pairs)사이의 차이가 존재함을 알 수 있다. HSC 의 경우는 여러 Progenitor 가 포함되어 있기 때문에 조건 특이적인 특성을 확인하기에는 부적절하다. 이는 개별 Progenitor 수준에서 조건 특이적인 PPIs 가 충분히 구축되어야만 분석이 가능할 것이다. 본 연구의 포괄적인 정규화 및 공동발현 측정법 비교결과를 근거로 마이크로어레이 상의 모든 유전자 쌍 사이의 연관성을 측정하여 새로운 조건 특이적인 PPIs 를 예측하는 것을 시도할 수 있다. 하지만, 여전히 PPIs 예측에 있어서 적정 공동발현 정도의 기준이 부재하기 때문에 경험적인 접근을 취해야할 것이다. 본 연구는 현재까지 줄기세포 및 종양세포에서 가능한 최대한의 조건 특이적인 PPIs 데이터를 구축시도하였으며, 여전히 부족한
데이터에 대하여 Interlog 맵핑을 적용하여 ESC 및 다른 조건에서 추가연구를 진행할 수 있을 것이다. 또한, 마이크로어레이 데이터의 정규화 방법 외에 다양한 이산화 방법을 적용하고, 여러 마이크로어레이 데이터를 통합하여 보다 포괄적인 비교분석을 남겨두고 있다.
V. 결론
본 연구를 통해 마이크로어레이를 통한 조건 특이적인 PPIs 예측정도를 여러 방법들 사이에서 비교하고, 새로운 PPIs 예측을 위한 참고정보를 제공하였다. 기존에 알려진 General PPIs 사이의 유전자 공동발현 결과와 일치하게 Condition-specific PPIs 에서는 General PPIs 보다 더 높은 연관성을 나타내었으며, 이러한 결과를 바탕으로 새로운 Condition-specific PPIs 를 예측함에 있어 적절한 방법을 사용하도록 제안할 수 있다. 또한, 추가연구로 다른 세포조건의 PPI 데이터 및 여러 GEP 데이터셋을 통합하여 보다 일관된 결과를 얻을 필요가 있으며, 해당 결과를 바탕으로 GEP 데이터 상의 전체 유전자쌍에 대하여 공동발현 측정을 통한 Condition-specific PPI 를 예측하고 이를 실험적으로 검증해야할 것이다.
참고문헌
1. Anonymous: <2007, Syst. Biol. and Comput. Proteomics, A Context-Specific Network of Protein-DNA and Protein-Protein Interactions Reveals New Regulatory Motifs in Human B Cells, Lefebvre et al.pdf>.
2. Anonymous: <2012, BMC bioinformatics, Comparison of co-expression measures mutual information, correlation, and model based indices., Song, Langfelder, Horvath.pdf>.
3. Ashburner M, Ball CA, Blake JA, Botstein D, Butler H et al.: Gene ontology: tool for the unification of biology. The Gene Ontology Consortium. Nat Genet
25(1):25-29, 2000
4. Bader S, Kuhner S, Gavin AC: Interaction networks for systems biology.
FEBS Lett 582(8):1220-1224, 2008
5. Bard J, Rhee SY, Ashburner M: An ontology for cell types. Genome Biol
6(2):R21, 2005
6. Barrett T, Troup DB, Wilhite SE, Ledoux P, Evangelista C et al.: NCBI GEO: archive for functional genomics data sets--10 years on. Nucleic Acids Res
39(Database issue):D1005-1010, 2011
7. Barrett T, Wilhite SE, Ledoux P, Evangelista C, Kim IF et al.: NCBI GEO: archive for functional genomics data sets--update. Nucleic Acids Res
41(Database issue):D991-995, 2013
8. Bhardwaj N, Lu H: Correlation between gene expression profiles and protein-protein interactions within and across genomes. Bioinformatics
21(11):2730-2738, 2005
9. Bolstad BM, Irizarry RA, Astrand M, Speed TP: A comparison of normalization methods for high density oligonucleotide array data based on variance and bias. Bioinformatics 19(2):185-193, 2003
10. Bossi A, Lehner B: Tissue specificity and the human protein interaction network. Mol Syst Biol 5:260, 2009
11. Brown KR, Jurisica I: Unequal evolutionary conservation of human protein interactions in interologous networks. Genome Biol 8(5):R95, 2007
12. Cai J, Xie D, Fan Z, Chipperfield H, Marden J et al.: Modeling co-expression across species for complex traits: insights to the difference of human and
mouse embryonic stem cells. PLoS Comput Biol 6(3):e1000707, 2010
13. Chikina MD, Huttenhower C, Murphy CT, Troyanskaya OG: Global prediction of tissue-specific gene expression and context-dependent gene networks in Caenorhabditis elegans. PLoS Comput Biol 5(6):e1000417, 2009
14. Das J, Mohammed J, Yu H: Genome-scale analysis of interaction dynamics reveals organization of biological networks. Bioinformatics 28(14):1873-1878, 2012
15. Duggan DJ, Bittner M, Chen Y, Meltzer P, Trent JM: Expression profiling using cDNA microarrays. Nat Genet 21(1 Suppl):10-14, 1999
16. Ewing RM, Chu P, Elisma F, Li H, Taylor P et al.: Large-scale mapping of human protein-protein interactions by mass spectrometry. Mol Syst Biol 3:89, 2007
17. Gitter A, Carmi M, Barkai N, Bar-Joseph Z: Linking the signaling cascades and dynamic regulatory networks controlling stress responses. Genome Res
23(2):365-376, 2013
18. Guan Y, Gorenshteyn D, Burmeister M, Wong AK, Schimenti JC et al.: Tissue-specific functional networks for prioritizing phenotype and disease genes. PLoS Comput Biol 8(9):e1002694, 2012
19. Ideker T, Krogan NJ: Differential network biology. Mol Syst Biol 8:565, 2012 20. Jansen R, Greenbaum D, Gerstein M: Relating whole-genome expression data
with protein-protein interactions. Genome Res 12(1):37-46, 2002
21. Lee HK, Hsu AK, Sajdak J, Qin J, Pavlidis P: Coexpression analysis of human genes across many microarray data sets. Genome Res 14(6):1085-1094, 2004
22. Lee K, Byun K, Hong W, Chuang HY, Pack CG et al.: Proteome-wide discovery of mislocated proteins in cancer. Genome Res, 2013
23. Lefebvre C, Rajbhandari P, Alvarez MJ, Bandaru P, Lim WK et al.: A human B-cell interactome identifies MYB and FOXM1 as master regulators of proliferation in germinal centers. Mol Syst Biol 6:377, 2010
24. Lim WK, Wang K, Lefebvre C, Califano A: Comparative analysis of microarray normalization procedures: effects on reverse engineering gene networks.
Bioinformatics 23(13):i282-288, 2007
25. Lin CC, Hsiang JT, Wu CY, Oyang YJ, Juan HF et al.: Dynamic functional modules in co-expressed protein interaction networks of dilated
cardiomyopathy. BMC Syst Biol 4:138, 2010
26. Lin J, Wilbur WJ: PubMed related articles: a probabilistic topic-based model for content similarity. BMC Bioinformatics 8:423, 2007
27. Lipscomb CE: Medical Subject Headings (MeSH). Bull Med Libr Assoc
88(3):265-266, 2000
28. Mani KM, Lefebvre C, Wang K, Lim WK, Basso K et al.: A systems biology approach to prediction of oncogenes and molecular perturbation targets in B-cell lymphomas. Mol Syst Biol 4:169, 2008
29. Perissi V, Aggarwal A, Glass CK, Rose DW, Rosenfeld MG: A corepressor/coactivator exchange complex required for transcriptional activation by nuclear receptors and other regulated transcription factors. Cell
116(4):511-526, 2004
30. Pop A, Huttenhower C, Iyer-Pascuzzi A, Benfey PN, Troyanskaya OG: Integrated functional networks of process, tissue, and developmental stage specific interactions in Arabidopsis thaliana. BMC Syst Biol 4:180, 2010 31. Priness I, Maimon O, Ben-Gal I: Evaluation of gene-expression clustering via
mutual information distance measure. BMC Bioinformatics 8:111, 2007
32. Przytycka TM, Singh M, Slonim DK: Toward the dynamic interactome: it's about time. Brief Bioinform 11(1):15-29, 2010
33. Reshef DN, Reshef YA, Finucane HK, Grossman SR, McVean G et al.: Detecting novel associations in large data sets. Science 334(6062):1518-1524, 2011
34. Rual JF, Venkatesan K, Hao T, Hirozane-Kishikawa T, Dricot A et al.: Towards a proteome-scale map of the human protein-protein interaction network. Nature 437(7062):1173-1178, 2005
35. Shoemaker BA, Panchenko AR: Deciphering protein-protein interactions. Part II. Computational methods to predict protein and domain interaction partners.
PLoS Comput Biol 3(4):e43, 2007
36. Skrabanek L, Saini HK, Bader GD, Enright AJ: Computational prediction of protein-protein interactions. Mol Biotechnol 38(1):1-17, 2008
37. Smialowski P, Pagel P, Wong P, Brauner B, Dunger I et al.: The Negatome database: a reference set of non-interacting protein pairs. Nucleic Acids Res
38(Database issue):D540-544, 2010